文章信息
- 杨金水, 高全秀, 李兆胜, 邢冠岚, 袁红莉
- YANG Jinshui, GAO Quanxiu, LI Zhaosheng, XING Guanlan, YUAN Hongli
- 小球藻Chlorella variabilis NC64A二酰甘油酰基转移酶基因的克隆表达与功能研究
- Clone, Expression and Function Analysis of Diacylglycerol Acyltransferase Gene from Chlorella variabilis NC64A
- 北京大学学报(自然科学版), 2016, 52(2): 187-192
- Acta Scientiarum Naturalium Universitatis Pekinensis, 2016, 52(2): 187-192
-
文章历史
- 收稿日期: 2014-11-21
- 修回日期: 2014-12-10
- 网络出版日期: 2016-03-16
与日益枯竭的化石燃料相比, 从微藻等有机体中获取生物能源具有可持续性的优点[1]。三酰甘油(TAG)是微藻的主要能量储存形式, 在限氮条件下其储量可占微藻干重的60%[2]。真核生物中TAG的合成途径主要有两种:一种是酰基-辅酶A (CoA)依赖型途径, 一种是非酰基-CoA依赖性途径[3]。酰基-CoA依赖型途径(即Kennedy途径)的最后一步是二酰甘油酰基转移酶(diacylglycerol acytrans-ferase, DGAT)催化酰基-CoA中的脂肪酸转移到二酰甘油(DAG)的sn-3位置, 生成TAG, 它是该途径唯一的限速酶[4]。Boyle等[5]发现, 限氮条件下, 莱茵衣藻油脂含量的提高伴随着DGAT2的转录量增加, 证实了DGAT2在微藻油脂合成中的重要作用。非酰基-CoA依赖型途径首先在酵母和植物中发现, 是在PDAT的作用下, 将磷脂酰胆碱中的脂肪酸转移到DAG上[6]。但是, 对于拟南芥中PDAT突变株及过表达植株的研究表明,PDAT在TAG合成中并未起到重要的作用[7-8], 而DGAT催化的酰化反应是TAG合成的关键步骤。与植物不同, 莱茵衣藻在限氮条件下, PDAT可将膜脂上的酰基转到二酰甘油上, 合成TAG, 从而增加油脂含量[9]。但是, 这一现象在其他微藻中是否具有普遍性, 尚不完全清楚。本研究对小球藻NC64A的DGAT2a基因进行克隆和原核表达, 并进一步分析其保守结构域与活性位点。酶活检测发现, 该DGAT2a具有PDAT活性, 表明微藻在限氮条件下的油脂含量增加可能确实与DGAT2a的PDAT活性相关。
1 材料与方法 1.1 菌株及培养Chlorella variabilisNC64A由内布拉斯加大学Van Etten教授惠赠。将藻种接种于装有100 mL培养基的300 mL三角瓶中, 恒温25℃, 140 rpm光照摇床培养, 光照强度为50 μmol/(m2·s), 光暗比为12:12, 培养时间为7天。每升接种量约为1010个细胞。培养结束后, 6000 rpm离心10分钟收集藻体, 并冷冻干燥。使用IM培养基, 成分为甘油1 g, 酸水解酪蛋白1.385 g, 酵母粉0.1 g, KH2PO4 0.74 g, Na2HPO4 0.207 g, CaCl2·2H2O 0.013 g, FeNaEDTA 0.01 g, MgSO4·7H2O 0.025 g, 微量元素母液1 mL, 定容至1 L, 调节pH至7.0。1 L微量元素母液中包括Al2(SO4)3·18H2O 3.58 g, MnCl2·4H2O 12.98 g, CuSO4·5H2O 1.83 g, ZnSO4·7H2O 3.2 g。
E. coli strain BL21 (DE3)为本实验室保存, 培养条件是37℃, LB培养基中200 rpm振荡培养过夜。抗生素筛选浓度为卡那霉素30 μg/mL。质粒pET30a (+)购自Promega公司。
1.2 RNA提取及PCR克隆表达将IM培养基中培养7天的Chlorella variabilis NC64A转接到无氮的相同培养基中, 继续培养2天后, 8000 rpm, 2分钟收集藻体, 采用TRIZOL Re-agent抽提总RNA, 并进行反转录。根据NCBI数据库中NC64A的dgat2a基因组序列GL433848设计引物PNC64A28 (NdeⅠ) GGAATT CCATATG ATGCTGCGCCTGCCCCTCC和PNC 64A29 (XhoⅠ) CCGCTCGAGCTCCACAAACCTCATGTCCCGC, 下划线为引入的酶切位点。PCR反应条件为: 95℃ 4分钟; 95℃ 30秒, 58℃ 30秒, 72℃ 1.5分钟, 30个循环; 72℃ 10分钟。凝胶电泳检测PCR结果, PCR纯化试剂盒纯化PCR产物。
采用内切酶NdeⅠ和XhoⅠ对PCR纯化后的产物和表达载体pET30a分别进行双酶切, 酶切产物纯化后使用T4DNA连接酶进行连接, 转化E. coli strain BL21 (DE3)感受态细胞, 并涂布在含有30 μg/mL卡那霉素的LB平板上, 最终选出阳性转化子E.coli BL21(DE3)/pET30a-dgat2a。
1.3 dgat2a的诱导表达分别挑取E.coli BL21(DE3)/pET30a-dgat2a和空载体宿主菌E. coli BL21(DE3)/pET30a (做对照)单菌落, 接种于含卡那霉素的LB培养基中, 37℃培养至OD600约为0.6, 然后加入IPTG至终浓度为1 mmol/L, 37℃诱导4小时, 离心收集菌体, 超声破碎, 4℃, 12000 rpm离心10分钟收集沉淀。
1.4 包涵体蛋白溶解及复性使用天恩泽公司的包涵体蛋白溶解及复性试剂盒, 对表达蛋白包涵体进行溶解和复性。
1.5 蛋白浓度测定、纯化及分子量测定使用BCA蛋白定量试剂盒(TakaRa)测定蛋白浓度。使用Ni SepharoseTM 6 Fast Flow纯化目的蛋白。采用SDS-PAGE法测定表观分子量。
1.6 DGAT2a的序列分析及同源建模根据NCBI和JGI Genome Portal (http://genome.jgi-psf.org/NC64A)数据库对DGAT2a进行同源性和保守位点比对[10]。采用SignalP 3.0和TMHMM分析其信号肽和跨膜区域。以SWISS-MODEL预测DGAT2a的三级结构。
1.7 DGAT2a的酶活定性检测采用薄层层析法检测DGAT2a的PDAT酶活[9]。
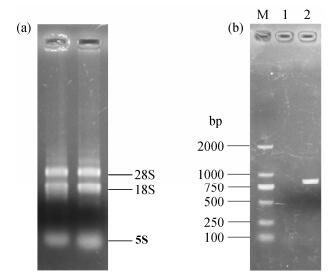
2 结果与讨论 2.1 Chlorella variabilisNC64A的RNA提取及PCR扩增由于限氮条件可以提高微藻油脂含量, 因此选取限氮培养的微藻细胞进行RNA提取, 并反转录cDNA作为后续基因扩增的模板。RNA提取后, 琼脂糖凝胶电泳检测结果如图 1(a)所示, 可以看出, 提取的RNA无基因组DNA参杂, 且无明显降解现象, 反转录合成cDNA后, 以cDNA为模板, 以PNC64A28和PNC64A29为引物PCR扩增dgat2a, 电泳检测结果见图 1(b), 阴性对照无条带, 扩增出900 bp左右的条带, 测序结果为894 bp, 与目的基因片段大小相符。翻译后的氨基酸序列与已有研究的Mortierella ramanniana的MrDGAT2A相似度为40%, 跨度为100%, 因此我们将来源于NC64A的该表达蛋白命名为CvDGAT2a。
|
|
(a) NC64A总RNA的提取; (b) dgat2a的PCR扩增; 1为空白对照, 2为阳性克隆 图 1. Chlorella varibilis NC64A dgat2a的克隆 Figure 1. The dgat2a clone from Chlorella varibilis NC64A |
2.2 dgat2a的诱导表达
重组菌株E. coli BL21/pET30a-dgat2a经IPTG诱导后, 分别将诱导前及诱导后的全菌进行SDS-PAGE电泳检测, 用空载体宿主菌E.coli BL21/ pET30a做对照(图 2)。从图 2可以看出, 与对照(第1, 2泳道)及诱导前(第3泳道)相比, 诱导后重组菌株(第4泳道)在33 kDa左右有一条表达条带, 和预测结果相符, 说明dgat2a已经被诱导表达。超声波破碎上清(第5泳道)无表达条带, 超声波沉淀(第6泳道)有表达条带, 说明目的蛋白是以包涵体的形式存在的。目前,对来源于真核生物的酰基转移酶的异源表达所采用的表达体系有酵母体系和昆虫体系[9, 11-12], 主要由于原核生物可能无法为真核生物来源的蛋白提供正确折叠的内环境。但相比真核表达系统, 大肠杆菌原核表达系统具有简单快速、表达量高的特点, 避免了酵母表达可能造成的糖基化修饰而使蛋白大小发生变化。因此, 本研究中采用大肠杆菌表达体系对dgat2a进行异源表达, 并进一步采用优化表达条件和包涵体复性, 以快速得到具有活性的目的蛋白。

|
| 1. E. coli BL21/pET30a的未诱导菌体; 2. E. coli BL21/ pET30a的诱导菌体; 3. E. coli BL21/ pET30a-dgat2a的未诱导菌体; 4. E. coli BL21/ pET30a-dgat2a的诱导菌体; 5. E. coli BL21/pET30a-dgat2a的超声波上清; 6. E. coli BL21/ pET30a-dgat2a的超声波沉淀 图 2. 诱导表达dgat2a的SDS-PAGE分析 Figure 2. SDS-PAGE analysis ofinduced dgat2a protein expression |
2.3 CvDGAT2a的复性及纯化
将上述离心获得的包涵体进行洗涤, 然后使用包涵体溶解液进行溶解, 分别取洗涤前混合液、洗涤后混合液、包涵体溶解液以及包涵体未溶解部分进行SDS-PAGE检测, 结果如图 3所示。从图 3中第2泳道和第1泳道比较可以看出, 包涵体洗涤后杂带有所减少, 目的条带亮度相差不多, 说明在洗涤过程中洗去了一些可溶性的蛋白, 而目的蛋白损失不多。从第3泳道和第2泳道相比可以看出, 包涵体大部分已溶解。从第5泳道可以看出, 部分杂蛋白未溶解, 目的蛋白基本全部溶解。

|
| 1.洗涤前包涵体混合液; 2.洗涤后包涵体混合液; 3.包涵体溶解液; 4.包涵体未溶解物 图 3. 包涵体洗涤和溶解 Figure 3. SDS-PAGE of washed and dissolved inclusion bodies |
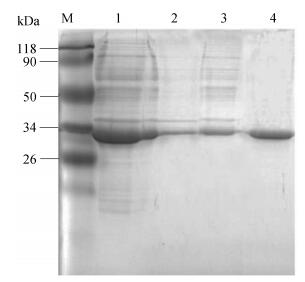
使用包涵体蛋白溶解及复性试剂盒中的复性液对得到的包涵体溶解液进行复性, 复性后使用Ni Sepharose™ 6 Fast Flow填料对目的蛋白进行纯化。从图 4中可以看出, 250 mmol/L咪唑的洗脱液含有较大浓度的目的蛋白, 且其表观分子量为33 kDa, pI为9.48, 这与已有报道的MrDGAT2A和MrDGAT2B相似[11]。虽然目前关于微藻dgat2a的原核表达尚未见报道, 但从图 3可以看到包涵体初步复性率达90%以上。虽然其中可能还存在部分未正确折叠的蛋白, 但采用原核表达体系可提高目的蛋白的表达量和提高效率, 具有一定的可行性。
|
| 1.总复性液; 2.穿透液; 3. 20 mmol/L咪唑洗脱液; 4. 250 mmol/L咪唑洗脱液 图 4. CvDGAT2a Ni离子柱纯化 Figure 4. SDS-PAGE of CvDGAT2a purification with Ni SepharoseTM 6 Fast Flow |
2.4 CvDGAT2a的三维结构及保守结构域分析
TMHMM2.0, Signalp-HMM和target pv1.1综合分析显示, CvDGAT2a的GRAVY为0.094, 推测为疏水蛋白, 且在N段开头有一段由20个氨基酸组成的信号肽序列, 胞内定位为线粒体。
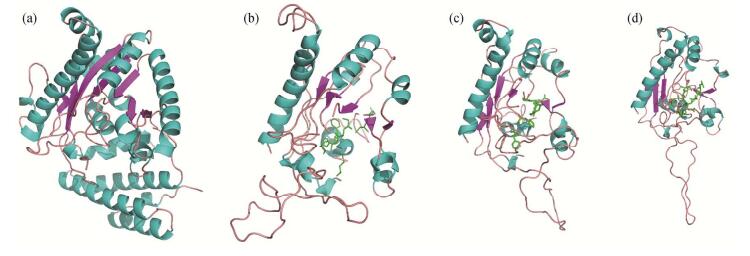
保守结构域分析表明该基因属于Lysophospho-lipid acyltransferases (LPLATs)超家族, 具有二酰甘油酰基转移酶活性。CvDGAT2a与来源于Morti-erella ramanniana的MrDGAT2A及来源于Schizo-sacharomyces pombe的SpDGAT2A具有相似的三级空间结构, 其三级结构中, α-螺旋占28.5% (图 5(b)蓝色部分所示), β-折叠占5.4% (图 5(b)紫红色所示), 序列位点H68, L71, F76, R94, I97和GAA (144-146)组成特定的酰基受体结合口袋(图 5(b)-(d)中草绿色部分所示), 能够结合酰基ACP或者酰基CoA上的酰基, 催化三酰甘油合成的最后一步。
|
| (a)为用于(b)~(d)模拟三级结构的模板liu.q.1.A; (b)~(d)为CvDGAT2a, MrDGAT2A和SpDGAT的三级结构图。草绿色代表酰基结合口袋, 蓝色代表α-螺旋, 紫红色代表β-折叠 图 5. CvDGAT2a三级结构比较 Figure 5. Comparison of the predicted structure of conserved domain of CvDGAT2a, MrDGAT2A and SpDGAT respectively |
将CvDGAT2a和LPLATs超家族中其他蛋白质进行比对, 发现其具有酰基转移酶家族D和E基序中最保守的氨基酸(图 6中灰点所示)。序列比对发现CvDGAT2a与Scheffersomyces stipitis CBS 6054的SsPDAT相似性为32%, 与Arabidopsis thaliana的AtPDAT相似性为24%。基于以上分析, 我们推测CvDGAT2a在具有DGAT活性的同时, 可能具有磷脂二酰甘油酰基转移酶活性。

|
| 图 6. CvDGAT2a与其他同源序列氨基酸比对结果 Figure 6. Sequence alignment of derived DGAT2a polypeptide sequences |
2.5 CvDGAT2a的PDAT酶活性测定
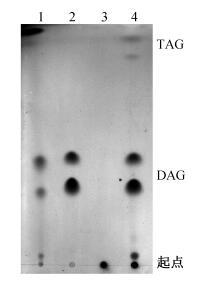
已有研究表明, 微藻在限氮条件下PDAT表达量增加, 可以促进膜脂降解合成三酰甘油[9], 而DGAT表达量的增加, 主要是促进了酰基辅酶A依赖的途径中的TAG含量的增加[5]。但是, 关于DGAT2a是否也参与了非酰基辅酶A依赖的TAG合成途径, 尚未见报道。为了验证CvDGAT2a在非酰基辅酶A依赖的TAG合成途径中是否起作用, 我们对目的蛋白的PDAT酶活进行检测, 结果如图 7所示。反应后, 出现较微弱的TAG条带, 说明目的蛋白确实具有PDAT活性。但是, 从图 7也可以看出, 表达蛋白的酶活不是很高, 反应后还有大量的底物残余, 这可能是因为目的蛋白复性不彻底导致大量蛋白呈非活性折叠状态所致。
|
| 1:三酰甘油标准样品; 2: 1, 2-二油酰-sn-甘油; 3: L-α-磷脂酰胆碱; 4:酶活反应底物 图 7. CvDGAT2a的酶活检测 Figure 7. Enzyme activity analysis of CvDGAT2a |
Li等[2]报道, 莱茵衣藻在限氮培养时, dgat2a转录量在12~48小时之间比正常条件下显著增加。本实验首次证实CvDGAT2a具有PDAT活性, 由此可推测, 限氮条件下dgat2a转录量的增加也许与其参与酰基辅酶A非依赖途径有关。我们推测, 小球藻NC64A在合成油脂的过程中, CvDGAT2a分别通过催化酰基辅酶A依赖的TAG合成途径和酰基辅酶A非依赖TAG合成途径促进油脂含量增加(图 8)。这一方面说明CvDGAT2a作为酶分子的底物的非专一性; 另一方面, 进一步解释了限氮条件下膜脂含量的下降不仅仅只有PDAT的催化, DGAT2a可能也起到一定的促进作用, 这为进一步阐明微藻油脂积累的机理奠定了基础。
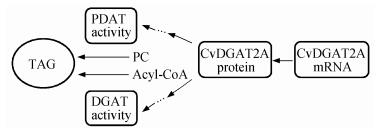
|
| 方框表示限氮条件下上调; PC为磷脂酰胆碱 图 8. CvDGAT2a在TAG合成途径中的作用模式 Figure 8. Model of function of DGAT2a in TAG synthesis |
3 结论
本文首次克隆并在大肠杆菌中异源表达了小球藻NC64A的DGAT蛋白, 其表观分子量为33 KDa, pI为9.48。通过序列分析发现, CvDGAT2a具有LPLATs超家族的保守氨基酸序列, 在限氮条件下, CvDGAT2a具有PDAT活性, 这从一个侧面解释了微藻油脂含量增加的原因, 为进一步的微藻油脂代谢调控奠定了一定的研究基础。
| [1] | Georgianna D R, Mayfield S P. Exploiting diversity and synthetic biology for the production of algal biofuels. Nature , 2012, 488 : 329–335 DOI:10.1038/nature11479 . |
| [2] | Li J, Han D, Wang D, et al. Choreography of transcriptomes and lipidomes of nannochloropsis reveals the mechanisms of oil synthesis in micro-algae. The Plant Cell , 2014, 26 : 1645–1665 DOI:10.1105/tpc.113.121418 . |
| [3] | Kroon J T, Wei W, Simon W J, et al. Identification and functional expression of a type 2 acylCoA:diacyl-glycerol acyltransferase (DGAT2) in developing castor bean seeds which has high homology to the major triglyceride biosynthetic enzyme of fungi and animals. Phytochemistry , 2006, 67 : 2541–2549 DOI:10.1016/j.phytochem.2006.09.020 . |
| [4] | Settlage S B, Kwanyuen P, Wilson R F. Relation between diacylglycerol acyltransferase activity and oil concentration in soybean. J Am Oil Chem Soc , 1998, 75 : 775–781 DOI:10.1007/s11746-998-0225-2 . |
| [5] | Boyle N R, Page M D, Liu B S, et al. Three acyltransferases and nitrogen-responsive regulator are implicated in nitrogen starvation-induced triacylgly-cerol accumulation in Chlamydomonas. Journal of Biological Chemistry , 2012, 287 : 15811–15825 DOI:10.1074/jbc.M111.334052 . |
| [6] | Dahlqvist A, Stahl U, Lenman M, et al. Phospho-lipid:diacylglycerol acyltransferase: an enzyme that catalyzes the acyl-CoA-independent formation of tria-cylglycerol in yeast and plants. Proceedings of the National Academy of Sciences of the United States of America , 2000, 97 : 6487–6492 DOI:10.1073/pnas.120067297 . |
| [7] | Mhaske V, Beldjilali K, Ohlrogge J, et al. Isolation and characterization of an Arabidopsis thaliana knockout line for phospholipid:diacylglycerol trans-acylase gene (At5g13640). Plant Physiol Bioch , 2005, 43 : 413–417 DOI:10.1016/j.plaphy.2005.01.013 . |
| [8] | Stahl U, Carlsson A S, Lenman M, et al. Cloning and functional characterization of a phospholipid:diacyl-glycerol acyltransferase from Arabidopsis. Plant Physiology , 2004, 135 : 1324–1335 DOI:10.1104/pp.104.044354 . |
| [9] | Yoon K, Han D, Li Y, et al. Phospholipid: diacyl-glycerol acyltransferase is a multifunctional enzyme involved in membrane lipid turnover and degradation while synthesizing triacylglycerol in the unicellular green microalga Chlamydomonas reinhardtii. The Plant Cell , 2012, 24 : 3708–3724 DOI:10.1105/tpc.112.100701 . |
| [10] | Blanc G, Duncan G, Agarkova I, et al. The Chlorella variabilis NC64A genome reveals adaptation to photosymbiosis, coevolution with viruses, and cryptic sex. The Plant Cell , 2010, 22 : 2943–2955 DOI:10.1105/tpc.110.076406 . |
| [11] | Lardizabal K D, Mai J T, Wagner N W, et al. DGAT2 is a new diacylglycerol acyltransferase gene family: purification, cloning, and expression in insect cells of two polypeptides from Mortierella ramanniana with diacylglycerol acyltransferase activity. The Journal of Biological Chemistry , 2001, 276 : 38862–38869 DOI:10.1074/jbc.M106168200 . |
| [12] | Liang M H, Jiang J G. Advancing oleaginous microorganisms to produce lipid via metabolic engineering technology. Progress in Lipid Research , 2013, 52 : 395–408 DOI:10.1016/j.plipres.2013.05.002 . |
 2016, Vol. 52
2016, Vol. 52




