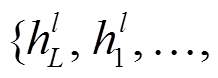
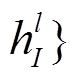 , 其中
, 其中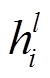 表示第 l 个 LM 层中第 i 个 token 的表示, 每一层输出的计算方法如下:
表示第 l 个 LM 层中第 i 个 token 的表示, 每一层输出的计算方法如下: 北京大学学报(自然科学版) 第60卷 第1期 2024年1月
Acta Scientiarum Naturalium Universitatis Pekinensis, Vol. 60, No. 1 (Jan. 2024)
doi: 10.13209/j.0479-8023.2023.073
中山市引进高端科研机构创新专项资金(2019AG031)资助
收稿日期: 2023–05–10;
修回日期: 2023–07–31
摘要 基于预训练语言模型(LM)和知识图谱(KG)的联合推理在应用于生物医学领域时, 因其专业术语表示方式多样、语义歧义以及知识图谱存在大量噪声等问题, 联合推理模型并未取得较好的效果。基于此, 提出一种面向生物医学领域的可解释推理方法 DF-GNN。该方法统一了文本和知识图谱的实体表示方式, 利用大型生物医学知识库构造子图并进行去噪, 改进文本和子图实体的信息交互方式, 增加对应文本和子图节点的直接交互, 使得两个模态的信息能够深度融合。同时, 利用知识图谱的路径信息对模型推理过程提供了可解释性。在公开数据集 MedQA-USMLE 和 MedMCQA 上的测试结果表明, 与现有的生物医学领域联合推理模型相比, DF-GNN 可以更可靠地利用结构化知识进行推理并提供解释性。
关键词 生物医学; 预训练语言模型; 知识图谱; 联合推理
问答系统是自然语言处理领域的一项热门研究课题。解决问答问题通常需要清楚地理解问题描述的场景, 然后利用相关知识进行推理[1], 最近, 大规模预训练语言模型(language model, LM)[2–3]成为多个问答数据集[4]中的流行解决方案, 并取得优异的性能。基于预训练语言模型和知识图谱(knowle-dge graph, KG)的联合推理模型[5–7]解决了 LM 无法利用外部知识进行结构化推理的问题。进一步地, 在文本上, 预训练语言模型已被证明有助于各种下游NLP 任务[8]。作为文本数据的补充, KG 提供结构化的背景知识, 通过预训练大规模学习两种模态的信息融合[9], 可进一步提高联合推理模型的性能。
尽管基于预训练语言模型和知识图谱的联合推理模型在开放域问答研究中得到广泛应用, 并取得优异性能, 但在生物医学问答(biomedical question and answering, BQA)领域, 联合推理问答(question and answering, QA)模型并未取得较好的效果。原因在于以下 3 个方面。1)生物医学领域专业术语表示方式多样, 语义存在歧义。例如, UMLS[10]和 Sem-Med[11]将 CUI 作为实体标识符, DragBank[12]将BankId 作为实体标识符, MIMIC-III[13]将 ICD-Code作为实体标识符。这些标识符之间的转化复杂, 并且不一一对应, 大大增加了问答数据集和知识库的实体对应难度。2)知识图谱存在大量的事实, 使得访问每一个给定问题的知识图谱十分困难, 虽然用构造知识子图[14]的方法缩减知识图谱有一定的效果, 但知识子图中仍然存在大量不相关实体, 会对联合推理造成干扰。3)联合推理过程中将全部文本作为知识图谱的头结点进行模态交互, 对应的文本实体与图谱实体之间并未进行交互[6–7], 两种模态的信息交互被平均化, 限制了两种模态之间交换有用信息的能力。
为了解决上述问题, 本文提出一种基于深度融合的语言模型与知识图谱联合推理问答模型 DF-GNN。首先, 使用概念唯一标识符(concept unique identifier, CUI)统一文本和知识图谱的实体表示, 使用 Scispacy[15]进行实体链接, 将文本实体与知识图谱实体一一对应, 消除语义歧义; 接着, 在构造知识子图时, 对链接到的实体进行过滤, 根据置信度得分选择链接实体; 然后, DF-GNN 改进了文本和知识图谱的交互方式, 将文本实体与对应的子图实体直接进行信息交互, 使得两个模态的信息深度融合, 提升每个实体对模型推理的影响程度; 最后, 对模型的推理过程进行可视化, 利用知识图谱的路径信息提供可解释性。
本研究在生物医学领域公开数据集 MedQA-USMLE[16]和 MedMCQA[17]上评估 DF-GNN, 使用SemMed 知识库构造知识图谱。为了与预训练联合推理模型[9]进行对比, 我们使用相同的预训练目标对 DF-GNN 模型进行预训练, 验证 DF-GNN 的性能表现。
我们的目标是利用 LM 处理非结构化问题文本, 并联合结构化的 KG 的知识来回答生物医学多项选择问题。在多选题回答(MCQA)的任务中, 一个通用的 MCQA 类型的数据集由上下文段落 c、问题 q和候选答案集合 A 组成, a 为集合 A 中的候选选项, 并且可以访问外部知识源 KG 进行联合推理, KG 提供与多选题内容相关的背景知识。
给定一个 MCQA 的例子(c, q, A)以及知识图谱G, 参照文献[18], 将 c, q 和 a 中的实体与 G 链接起来, 然后从 G 中提取问题–选择对的知识子图 Gsub,并进行去噪, 将(c, q, a)以及 Gsub 作为模型的输入, 得到 a 作为答案的概率, 概率最高的 a 即为问题的最终答案。
本文通过引入外部知识 KG, 利用图神经网络(GNN)来增强 LM[3,19–20], 提出 DF-GNN 方法。如图1 所示, DF-GNN 由 5 个部分组成: 1)问答上下文编码模块, 即学习非结构化问答文本输入表示的 LM层; 2)知识子图提取模块, 包括知识子图的构造和去噪; 3)图编码模块, 即学习结构化知识图谱输入表示的 GAT 层; 4)深度融合模块, 即学习文本和对应知识子图联合表示的深度模态交互 DF 层, 其将底层 LM 层输出的文本表示与 Gsub 的图表示相互融合, 每一对文本实体与子图实体直接交互, 提升每个实体对模型推理的影响程度; 5)答案预测模块。
DF 层中不同颜色的 token-node 对表示从文本链接到知识子图的对应实体对, token 和 node 表示前一层文本和节点嵌入, token_p 和 node_p 表示预融合文本和节点嵌入, token_f 和node_f 表示融合文本和节点嵌入。我们将 LM 层数表示为 N, DF 层数表示为 M, 模型中的总层数为 N+M。
首先, 将上下文段落 c、问题 q 和候选答案 a 用分隔符连接起来, 得到输入 L=[c; q; a]。然后, 将输入 L 馈送到 LM 层, 得到输出表示列表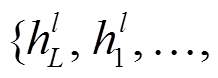
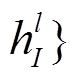 , 其中
, 其中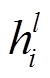 表示第 l 个 LM 层中第 i 个 token 的表示, 每一层输出的计算方法如下:
表示第 l 个 LM 层中第 i 个 token 的表示, 每一层输出的计算方法如下:
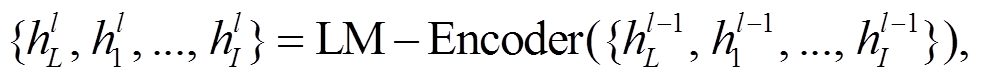 (1)
(1)
其中, 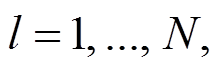 I 是 token 数量, N 是 LM 层数, LM- Encoder(·)是单层 LM 编码器层, 其参数使用预训练模型初始化。
I 是 token 数量, N 是 LM 层数, LM- Encoder(·)是单层 LM 编码器层, 其参数使用预训练模型初始化。
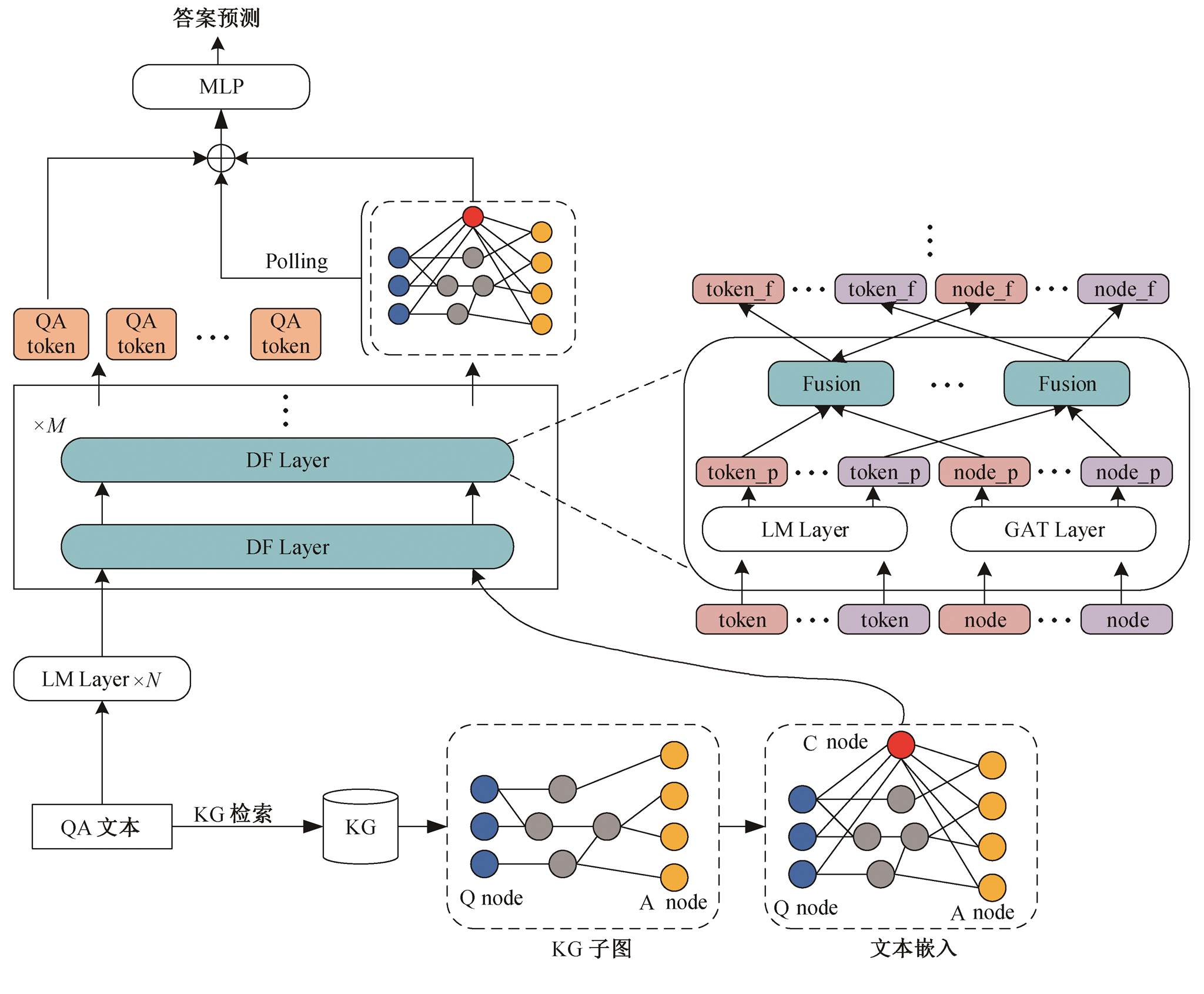
知识子图中的蓝色实体节点表示问题中提到的实体, 黄色实体节点表示答案中提到的实体, 红色节点表示问答文本节点
图1 DF-GNN模型的架构
Fig. 1 Model architecture of DF-GNN
2.2.1KG检索
对于每个 QA 上下文, 首先使用 SciSpacy[15]将(c, q, a)中识别出的实体链接到 G, 得到一组初始节点集合; 然后将初始节点集合中的每一个节点的两跳邻居添加进初始节点集, 得到检索节点集。
2.2.2KG去噪
根据实体链接置信度, 对检索节点集进行去噪处理, 方法如下: 1)对于从 c 和 q 识别出的实体, 抽取置信度高于 0.88(对实体链接统计分析可以得出, 当置信度高于 0.88 时, 链接实体的数量平均为 2 个)的实体作为最终链接实体; 2)对于从 a 识别出的实体, 首先将 a 作为一个实体, 并在 G 中检索, 若存在该实体, 则该实体即为最终链接实体, 否则进行去噪处理方式同第 1 步。两步操作后, 得到知识子图节点集{e1, …, eJ} (J 为节点数量), 然后检索连接知识子图节点集中任意两个节点的所有边, 形成Gsub。对于 Gsub 中的每个节点, 根据其对应的实体是从上下文 c或问题 q、答案 a 还是桥接路径连接而被分配对应类型。
为了表示 Gsub, 首先将知识子图节点集作为LM 的输入, 得到链接实体的初始节点嵌入![]()
![]() , 同时将整个 QA 文本 L 作为 Gsub 的头结点 eL, 其初始节点嵌入
, 同时将整个 QA 文本 L 作为 Gsub 的头结点 eL, 其初始节点嵌入![]() 被随机初始化; 然后在 GAT 的每一层中, 节点嵌入的当前表示
被随机初始化; 然后在 GAT 的每一层中, 节点嵌入的当前表示![]()
![]() 被馈送到当前层, 从而在图中的节点之间执行一轮信息传播, 并产生每个实体的预融合节点嵌入:
被馈送到当前层, 从而在图中的节点之间执行一轮信息传播, 并产生每个实体的预融合节点嵌入:
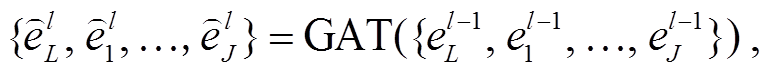 (2)
(2)
其中, ![]()
 表示节点的预融合嵌入, M 是GAT 层数, l 是当前所在层, GAT 对应图注意力网络。GAT 通过图中邻居之间的消息传递, 计算每个节点
表示节点的预融合嵌入, M 是GAT 层数, l 是当前所在层, GAT 对应图注意力网络。GAT 通过图中邻居之间的消息传递, 计算每个节点 的节点表示
的节点表示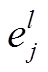 :
:
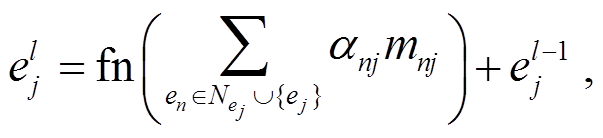 (3)
(3)
其中, Nej 表示任意节点 ej 的邻域,αnj表示传播消息的注意力权重, mnj表示邻域中某一邻居 en 传递给ej 的消息, fn是两层 MLP。
节点之间的关系嵌入 rnj 以及消息 mnj 通过下式计算:
rnj=fr(tnj, un, uj) , (4)
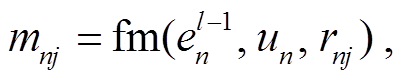 (5)
(5)
其中, un, uj∈{0, 1, 2}表示节点 n 和 j 的类型, tnj 是连接节点 en 和 ej 关系的关系嵌入表示, fr 是两层 MLP, fm 是线性变换。注意力权重 αnj 通过其重要性衡量每个邻居消息的贡献, 通过以下公式计算:
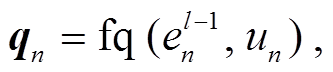 (6)
(6)
 (7)
(7)
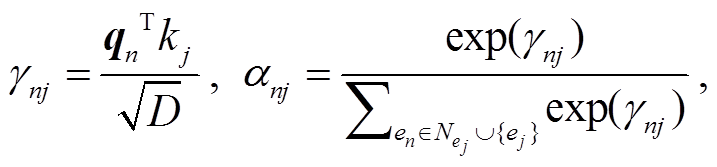 (8)
(8)
其中, fq 和 fk 是线性变换。
在第 l 个 DF 层中, 输入的 token 嵌入![]()
![]() 被馈送到额外的 LM 编码层中, 继续对文本的上下文进行编码:
被馈送到额外的 LM 编码层中, 继续对文本的上下文进行编码:
 (9)
(9)
其中, ![]()
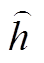 表示 token 的预融合嵌入。
表示 token 的预融合嵌入。
在 LM 层和 GAT 层分别更新 token 嵌入和节点嵌入后, 通过融合层 Fusion, 使两种模态通过对应的 token 和节点融合信息, 将对应的 token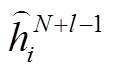 和节点
和节点![]() 的预融合嵌入连接起来, 通过 Fusion 层传递联合表示, 然后将输出的融合嵌入拆分为
的预融合嵌入连接起来, 通过 Fusion 层传递联合表示, 然后将输出的融合嵌入拆分为![]() 和
和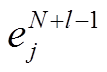 :
:
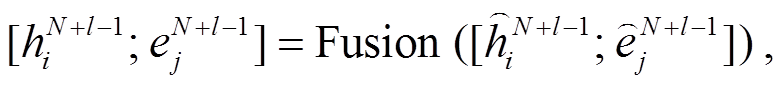 (10)
(10)
其中, hi与ej是一个 token-node 对, 即表示同一实体的文本嵌入和节点嵌入, Fusion 是两层 MLP。只有 token-node 对参与运算, 其余 token和节点保持不变, 但是它们会在各自模态传播的下一层(式(1)和(2))从交互 token-node 对中的 token 或节点中接收信息。因此, 两种模态的信息在多个 DF 层中直接交互, 语言表示与 KG 知识深度融合, 提升了每个实体对模型推理的影响程度。
对于 MCQA 任务, 给定语言上下文 L 和 Gsub, 根据上下文表示![]() 、从 Gsub 学习的 QA 文本节点表示
、从 Gsub 学习的 QA 文本节点表示![]() 以及最后一个 GAT 层的 Gsub 池化表示pGsub, 计算候选答案的分数:
以及最后一个 GAT 层的 Gsub 池化表示pGsub, 计算候选答案的分数:
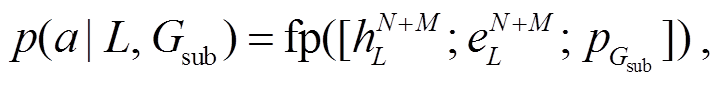 (11)
(11)
其中, fp表示两层 MLP。最后, 选择得分最高的候选答案为预测输出, 使用交叉熵损失函数来优化端到端模型。
本研究在生物医学领域公开数据集 MedQA-USMLE[16]和 MedMCQA[17]上评估 DF-GNN。Med-QA-USMLE 是一个 4 项多项选择题回答数据集, 这些问题来自美国医学执照考试(USMLE)的练习测试。该数据集包含 12723 个问题, 我们使用原始数据拆分方法[16]。MedMCQA 是一个选择题数据集, 数据来源于印度两所医学院入学考试(AIIMS 和NEET-PG)的模拟考试和过往考试, 训练集包含182822 个问题, 测试集包含 4183 个问题, 每个问题有 4 个选项。
我们遵循基线模型[5–7], 使用准确率得分(Acc)作为评估指标。
使用 BioLinkBERT[19]作为 DF-GNN 的预训练语言模型, 模型的超参数如表 1 所示。
使用生物医学领域的公开知识库 SemMed[11]作为外部知识源 G。该知识库是从整个 PubMed 引用集中提取的语义预测(三元组)的存储库, 语义预测的元素来自统一医学语言系统(UMLS)的知识源, SemMed 中的实体概念与 UMLS 对齐, 30 种常见预定义关系从 UMLS 的预定义关系中引出。
表1 模型和实验超参数设置
Table 1 Hyperparameter settings for models and experiments

类别超参数MedQA-USMLE 模型架构DF层数M3 LM层数N9 GAT注意头数2 GAT节点嵌入维度200 MLP隐藏层维度(DF 层除外)200 MLP隐藏层数1 DF层隐藏层维度400 正则化嵌入层、GNN层、全连接层Dropout rate0.2 LM参数学习率5×10–5 非LM参数学习率1×10–3 LM参数保持冻结的周期数0 优化优化器RAdam 学习率固定 批量大小128 周期数20 最大梯度规范(梯度剪切)1.0 数据最大节点数200 最大token数512
3.3.1微调预训练语言模型
为了研究使用 KG 作为外部知识源的效果, 我们将 DF-GNN 与原生预训练语言模型进行对比, 后者是知识不可知的, 我们选择 BioBERT[19], Sap-BERT[21]和 BioLinkBERT[20]进行对比。
3.3.2LM + KG模型
通过与现有的 LM+KG 方法进行比较, 来评估DF-GNN 利用知识图谱推理的能力。选择 QA-GNN[5], GreaseLM[6]以及 Dragon[9]进行比较。Gre-aseLM 是现有的性能最好的模型, Dragon 在 Grease-LM 的基础上对模型进行预训练, 取得了更好的效果。为了公平比较, 使用与本文模型相同的 LM 来初始化这些基线模型。
表 2 和 3 分别展示 MedQA-USMLE 和 Med- MCQA 数据集上的实验结果。我们不仅在 SemMed知识图谱以及去噪图谱上进行实验, 同时也利用去噪图谱改进对比模型的性能。我们观察到 DF-GNN的性能优于所有 LM 模型和 LM+KG 模型, 并且优于预训练模型 Dragon。除了 DF-GNN, MedQA-US-MLE 和 MedMCQA 上的 BioLinkBERT-large 和 Gre-aseLM 是最好的 LM 微调模型和 KG 增强模型, Dra-gon 是最好的预训练模型。在 MedQA-USMLE 数据集上, DF-GNN 相对于 BioLinkBERT-large 微调模型有 2.5%的改进, 相对于最佳模型 GreaseLM 有2%的改进, 相对于预训练模型 Dragon 有 1.6%的改进。在MedMCQA 数据集上, DF-GNN 相对于最佳模型 Gr-easeLM 有 1.7%的改进, 相对于预训练模型 Dragon有 0.9%的改进。在 MedQA-USMLE 和 MedMCQA数据集上, DF-GNN 的性能表现证明了统一实体表示方式、图谱去噪处理以及模态信息直接交互的有效性。
表2 MedQA-USMLE数据集上的模型效果对比
Table 2 Performance of baseline models on MedQA-USMLE

模型知识图谱准确率/% BioBERT-base[19]–34.1 BioBERT-large[19]–36.7 SapBERT[21]–37.2 BioLinkBERT-base[20]–40.0 BioLinkBERT-large[20]–44.6 +QA-GNN[5]UMLS+DrugBANK45.0 SemMed45.4 SemMed-denoise46.4 +GreaseLM[6]UMLS+DrugBANK45.1 SemMed45.6 SemMed-denoise46.6 +DF-GNN (本文方法)UMLS+DrugBANK45.4 SemMed45.8 SemMed-denoise47.1 +Dragon[9]UMLS+DrugBANK47.5 SemMed47.6 SemMed-denoise48.4 +pretrain+DF-GNN (本文方法)UMLS+DrugBANK48.0 SemMed48.6 SemMed-denoise49.1
说明: 粗体数字表示性能最优, 下同。
表3 MedMCQA数据集上的模型效果对比
Table 3 Performance of baseline models on MedMCQA

方法知识图谱准确率/% BioLinkBERT-large[20]–– +QA-GNN[5]UMLS+DrugBANK53.7 SemMed54.3 SemMed-denoise54.7 +GreaseLM[6]UMLS+DrugBANK53.9 SemMed54.4 SemMed-denoise54.8 +DF-GNN (本文方法)UMLS+DrugBANK54.5 SemMed54.9 SemMed-denoise55.6 +Dragon[9]UMLS+DrugBANK55.3 SemMed55.5 SemMed-denoise55.8 +pretrain+DF-GNN (本文方法)UMLS+DrugBANK55.6 SemMed55.9 SemMed-denoise56.2
4.2.1实体表示方法
不同于基线模型使用 UMLS+DrugBank(BankId +CUI)作为外部知识源, 我们使用 SemMed(CUI)作为外部知识源, 统一了文本和知识图谱的实体表示方式, 并在 MedQA-USMLE 和 MedMCQA 数据集上进行实验。如表 4 所示, 使用 CUI 统一实体表示后, 基线模型以及 DF-GNN 的性能均有所提高, 证明了统一实体表示的有效性。
4.2.2知识图谱去噪
表 4 中, 所有基线模型以及 DF-GNN 性能的提升并不明显, 我们认为是因为 SemMed 知识图谱中存在噪声, 导致大量无关实体影响模型的推理性能。我们对知识图谱去噪, 得到 Gsub, 如表 5 和 6 所示。去噪后, Gsub 问题实体和答案实体的数量明显下降, 所有基线模型以及 DF-GNN 的性能均有较大的提升。
4.2.3DF-GNN预训练方法选择
如表 7 所示, 我们遵从 Dragon[9]的方法, 在MedQA-USMLE 数据集上对 DF-GNN 进行同样的预训练, 在预训练目标上对比 MLM(掩码语言建模)、LinkPred(链接预测)和 MLM+LinkPred 的效果, 在 LinkPred 的头部选择上对比 DistMult, TransE和 RotatE 三种方法。与 Dragon[9]的结论一致, 在文本和 KG 上进行双向自监督任务, 有助于模型融合两种推理模式, 在预测头部的选择中, DistMult 的效果最优。
表4 MedQA-USMLE和MedMCQA上不同实体表示的效果对比
Table 4 Performance of different entity representations on MedQA-USMLE and MedMCQA

方法知识图谱准确率/%MedQA-USMLEMedMCQA BioLinkBERT-large[19]–44.6– +QA-GNN[5]UMLS+DrugBANKSemMed45.045.453.754.3 +GreaseLM[6]UMLS+DrugBANKSemMed45.145.653.954.4 +DF-GNN (本文方法)UMLS+DrugBANKSemMed45.445.854.554.9 +Dragon[9]UMLS+DrugBANKSemMed47.547.655.355.5 +pretrain+DF-GNN (本文方法)UMLS+DrugBANKSemMed48.048.655.655.9
表5 MedQA-USMLE和MedMCQA上知识图谱中实体数量比较
Table 5 Number of entities in knowledge subgraph on MedQA-USMLE and MedMCQA

数据集知识图谱MedQA-USMLEMedMCQAc和q数量(平均)a数量(平均)c和q数量(平均)a数量(平均) trainSemMed74.524.419.483.90 devSemMed73.924.4710.224.24 testSemMed75.924.379.363.95 trainSemMed-denoise26.721.573.381.35 devSemMed-denoise26.601.563.651.49 testSemMed-denoise27.244.563.331.36
表6 MedQA-USMLE和MedMCQA上去噪知识图谱性能比较
Table 6 Performance of denoise knowledge graph on MedQA-USMLE and MedMCQA

模型知识图谱准确率/%MedQA-USMLEMedMCQA BioLinkBERT-large[19]–44.6– +QA-GNN[5]SemMed45.454.3 SemMed-denoise46.454.7 +GreaseLM[6]SemMed45.654.4 SemMed-denoise46.654.8 +DF-GNN (本文方法)SemMed45.854.9 SemMed-denoise47.155.6 +Dragon[9]SemMed47.655.5 SemMed-denoise48.455.8 +pretrain+DF-GNN (本文方法)SemMed48.655.9 SemMed-denoise49.156.2
表7 DF-GNN预训练方法选择
Table 7 Pretraining design choices of DF-GNN

消融类型消融方法准确率/% 预训练目标MLM + LinkPred (final)49.1 MLM only48.2 LinkPred only47.9 LinkPred headDistMult (final)49.1 TransE47.9 RotatE48.0
4.3.1推理结果可视化
本研究的目的是通过提取 GAT 引起的节点到节点的注意力权重来展示 DF-GNN 的推理过程, 我们通过 Gsub 的注意力邻接矩阵, 挑选注意力权重高于给定阈值的边和对应头尾结点, 进行可视化展示。图 2 为 DF-GNN 在单跳推理和多跳推理问题的两个示例。可以看到, 通过文本和知识图谱之间的联合推理, DF-GNN 在单跳和多跳问题中都可以找到关键实体, 从而推理出正确答案。
4.3.2定性分析
如图 3 所示, 我们通过 MedQA-USMLE 数据集的一个示例, 对比 DF-GNN 与 Dragon模型对问答文本各实体的注意力权重。在示例中, DF-GNN 正确地预测了答案是 C “轮状病毒”, 而 Dragon 做出错误预测, 即 D “脊髓灰质炎病毒”。对于这两个模型, 我们观察通过 Gsub 提取的节点的注意力权重可以发现, DF-GNN 模型重点关注在“基因”, 并对与该实体相连的“基因重组”“双链RNA”和“病毒重组”更关注; Dragon 模型重点关注“病毒”以及相连的“病毒颗粒”。我们认为, Dragon 在头节点的交互信息被平均到每个实体, 降低了“基因”对于模型推理的重要程度, DF-GNN 的直接交互方式提升了“基因”对模型推理的影响, 因此能够预测正确答案。
本文针对生物医学领域问答存在的问题, 提出一种新的模型 DF-GNN, 通过统一文本和知识图谱的实体表示、对知识子图去噪处理以及改进文本与知识图谱的交互方式, 将文本实体与对应的子图实体直接交互, 使两个模态的信息深度融合, 提升每个实体对模型推理的影响程度。在生物医学领域数据集上的实验结果表明, DF-GNN 模型优于微调 LM基线[19–21]以及现有的最佳 LM+KG 模型[5–6]和预训练模型。同时, 通过提取 GAT 引起的节点到节点的注意力权重来展示 DF-GNN 的推理过程, 利用知识图谱的路径信息对模型推理提供可解释性。
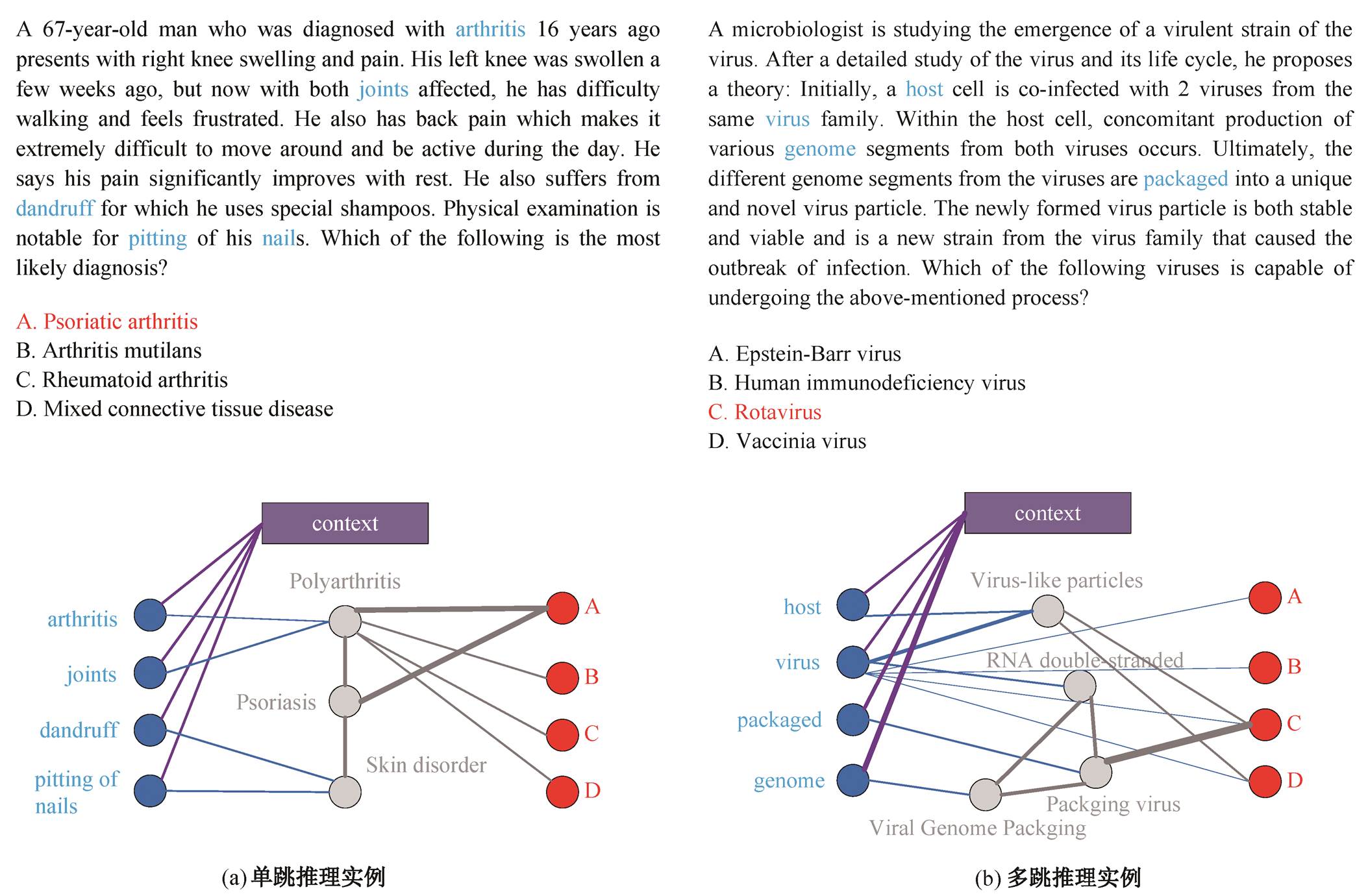
图2 DF-GNN推理结果展示
Fig. 2 Reasoning result of DF-GNN
图3 DF-GNN与Dragon的推理结果比较
Fig. 3 Comparison of reasoning results between DF-GNN and Dragon
参考文献
[1] Jin Qiao, Yuan Zheng, Xiong Guangzhi, et al. Bio-medical question answering: a survey of approaches and challenges. ACM Computing Surveys, 2022, 55 (2): 1–36
[2] Gu Yu, Tinn R, Cheng Hao, et al. Domain-specific language model pretraining for biomedical natural lan-guage processing. ACM Transactions on Computing for Healthcare, 2022, 3(1): 1–23
[3] Liu Fangyu, Shareghi E, Meng Zaiqiao, et al. Self-alignment pretraining for biomedical entity represen-tations [EB/OL]. (2020–10–22)[2023–05–20]. https:// arxiv.org/abs/2010.11784
[4] Mutabazi E, Ni J, Tang Guangyi, et al. A review on medical textual question answering systems based on deep learning approaches. Applied Sciences, 2021, 11 (12): 54–56
[5] Yasunaga M, Ren Hongyu, Bosselut A, et al. QA-GNN: reasoning with language models and knowledge graphs for question answering [EB/OL]. (2021–04–13)[2023–05–20]. https://arxiv.org/abs/2104.06378
[6] Zhang Xikun, Bosselut A, Yasunaga M, et al. Grea-selm: graph reasoning enhanced language models for question answering [EB/OL]. (2022–02–21)[2023–05–20]. https://arxiv.org/abs/2201.08860
[7] Chen Zheng, Kordjamshidi P. Dynamic relevance graph network for knowledge-aware question answe-ring [EB/OL]. (2022–09–20)[2023–05–20]. https://ar xiv.org/abs/2209.09947
[8] Bommasani R, Hudson D A, Adeli E, et al. On the opportunities and risks of foundation models [EB/OL]. (2021–08–16)[2023–05–20]. https://arxiv.org/abs/2108. 07258
[9] Yasunaga M, Bosselut A, Ren Hongyu, et al. Deep bidirectional language-knowledge graph pretraining. Advances in Neural Information Processing Systems, 2022, 35: 37309–37323
[10] Bodenreider O. The unified medical language system (UMLS): integrating biomedical terminology. Nucleic acids research, 2004, 32(suppl 1): D267–D270
[11] Kilicoglu H, Shin D, Fiszman M, et al. SemMedDB: a PubMed-scale repository of biomedical semantic pre-dications. Bioinformatics, 2012, 28(23): 3158–3160
[12] Wishart D S, Knox C, Guo A C, et al. DrugBank: a knowledgebase for drugs, drug actions and drug tar-gets. Nucleic Acids Research, 2008, 36(suppl 1): D901 –D906
[13] Johnson A E W, Pollard T J, Shen L, et al. MIMIC-III, a freely accessible critical care database. Scientific Data, 2016, 3(1): 1–9
[14] Haveliwala T H. Topic-sensitive pagerank // Procee-dings of the 11th International Conference on World Wide Web. Honolulu, 2002: 517–526
[15] Neumann M, King D, Beltagy I, et al. ScispaCy: fast and robust models for biomedical natural language pro-cessing [EB/OL]. (2019–02–20)[2023–05–20]. https:// arxiv.org/abs/1902.07669
[16] Jin D, Pan E, Oufattole N, et al. What disease does this patient have? a large-scale open domain question an-swering dataset from medical exams. Applied Scien-ces, 2021, 11(14): 6421–6422
[17] Pal A, Umapathi L K, Sankarasubbu M. Medmcqa: A large-scale multi-subject multi-choice dataset for medical domain question answering // Conference on Health, Inference, and Learning. New Orleans, 2022: 248–260
[18] Lin B Y, Chen X, Chen J, et al. KagNet: knowledge-aware graph networks for commonsense reasoning [EB/OL]. (2019–09–04)[2023–05–20]. https://arxiv. org/abs/1909.02151
[19] Lee J, Yoon W, Kim S, et al. BioBERT: a pre-trained biomedical language representation model for biome-dical text mining. Bioinformatics, 2020, 36(4): 1234–1240
[20] Yasunaga M, Leskovec J, Liang P. LinkBERT: pretrai-ning language models with document links [EB/OL]. (2022–05–29)[2023–05–20]. https://arxiv.org/abs/2203. 15827
[21] Beltagy I, Lo K, Cohan A. SciBERT: a pretrained language model for scientific text [EB/OL]. (2019–05–26)[2023–05–20]. https://arxiv.org/abs/1903.10676
Interpretable Biomedical Reasoning via Deep Fusion of Knowledge Graph and Pre-trained Language Models
Abstract Joint inference based on pre-trained language model (LM) and knowledge graph (KG) has not achieved better results in the biomedical domain due to its diverse terminology representation, semantic ambiguity and the presence of large amount of noise in the knowledge graph. This paper proposes an interpretable inference method DF-GNN for biomedical field, which unifies the entity representation of text and knowledge graph, denoises the subgraph constructed by a large biomedical knowledge base, and further improves the information interaction mode of text and subgraph entities by increasing the direct interaction between corresponding text and subgraph nodes, so that the information of the two modes can be deeply integrated. At the same time, the path information of the knowledge graph is used to provide interpretability for the model reasoning process. The test results on the public dataset MedQA-USMLE and MedMCQA show that DF-GNN can more reliably leverage structured knowledge for reasoning and provide explanatory properties than existing biomedical domain joint inference models.
Key words biomedical domain; pre-trained language model; knowledge graph; joint reasoning