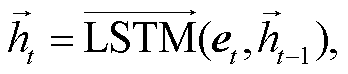 (2)
(2)
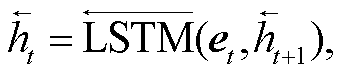 (3)
(3)
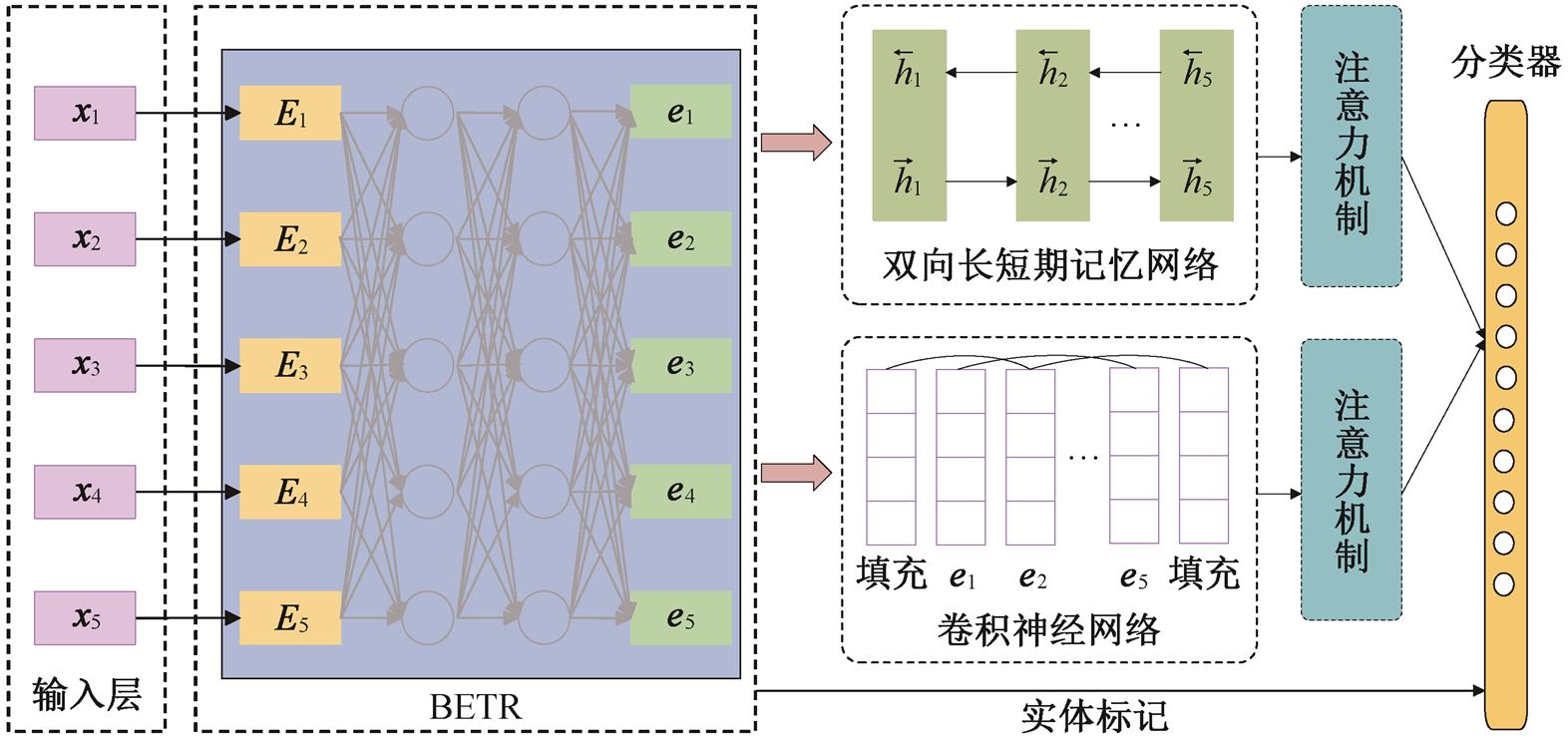
图1 本文模型的总体结构
Fig. 1 Overall structure diagram of the proposed model
北京大学学报(自然科学版) 第59卷 第1期 2023年1月
Acta Scientiarum Naturalium Universitatis Pekinensis, Vol. 59, No. 1 (Jan. 2023)
doi: 10.13209/j.0479-8023.2022.065
国家自然科学基金(61876031)和国家科技创新2030—“新一代人工智能”重大项目(2020AAA08000)资助
收稿日期: 2022‒05‒09;
修回日期: 2022‒07‒26
摘要 医疗文本具有实体密度高、句式冗长等特点, 简单的神经网络方法不能很好地捕获其语义特征, 因此提出一种基于预训练模型的混合神经网络方法。首先使用预训练模型获取动态词向量, 并提取实体标记特征; 然后通过双向长短期记忆网络获取医疗文本的上下文特征, 同时使用卷积神经网络获取文本的局部特征; 再使用注意力机制对序列特征进行加权, 获取文本全局语义特征; 最后将实体标记特征与全局语义特征融合, 并通过分类器得到抽取结果。在医疗领域数据集上的实体关系抽取实验结果表明, 新提出的混合神经网络模型的性能比主流模型均有提升, 说明这种多特征融合的方式可以提升实体关系抽取的效果。
关键词 医疗文本; 关系抽取; 混合神经网络; 预训练模型
随着医疗信息化的推进, 医疗领域产生大量非结构化的文本, 如医学文献和临床记录等。这些文本包含丰富的医疗信息, 如药物和疾病等医疗实体以及临床表现和药物治疗等实体关系。在非结构化的医疗文本中挖掘有用信息, 构建高水平的医疗知识库, 能够促进医疗信息化的发展, 也是自然语言处理(natural language processing, NLP)领域重要的研究方向。
实体关系抽取是信息抽取[1]的重要任务之一, 旨在从非结构化的文本中抽取出不同实体之间的语义关系, 从而提取有用的信息, 这是构建知识图谱时一个非常重要的过程[2]。在 1998 年的信息理解会议(message understanding conference, MCU)上首次提出实体关系抽取任务, 经过多年的发展, 通用领域的关系抽取任务已经较好地完成。医疗领域的实体关系抽取发展较晚, 早期的生物医疗关系任务有蛋白质‒蛋白质关系抽取(protein-protein interaction, PPI)[3]、化合物‒疾病关系抽取(chemical-disease re-lation, CDR)[4]以及药物‒药物关系抽取(drug-drug interaction, DDI)[5]等。后来, 医疗领域的实体关系抽取逐渐成为研究热点。
早期医疗领域的实体关系抽取方法均基于词典和规则层面, 需要由医疗领域的专家制定专业的医疗词典和医疗知识库, 再设计制定规则模板来进行医疗实体关系的抽取[6]。基于词典和规则的方法有一定的效果, 但是需要医疗专家的专业知识, 其准确率依赖于词典和规则的质量, 在大规模医疗文本数据集上的泛化能力和效果很差。
随着深度学习技术的发展, 神经网络方法被用于医疗实体关系抽取, 减少了对人工的依赖, 可以稳定并准确地从医疗文本中学习特征。卷积神经网络(convolution neural network, CNN)[7]可以获取文本的局部特征, 但忽略了文本的长距离依赖。循环神经网络(recurrent neural network, RNN)[8]可以获取文本的上下文语义特征, 但不能提取局部信息, 且忽略了句法和语义层面的特征。注意力机制(atten-tion mechanism, ATT)[9]可以加权序列特征, 增强模型捕获文本特征的能力。Google 团队 2018 年发布以 Transformers 为基础的预训练语言模型(bidirec-tional encoder representations from transformers, BE-RT)[10], 能够获取结合上下文语境的动态词向量, 在自然语言处理领域取得良好的效果。
不同于通用领域的文本, 医疗领域的文本中存在大量分布密集的专业领域实体, 实体之间关系交错复杂, 简单的神经网络方法无法有效地获取文本中的语义特征。如在“胰腺癌首次治疗 4 个月后, 上腹部超声检查显示胰腺肿块与肝转移。”这个医疗文本实例中存在 3 种类型(疾病、检查和症状)的 4个实体(胰腺癌、上腹部超声检查、胰腺肿块和肝转移), 并存在(胰腺癌, 影像学检查, 上腹部超声检查)、(胰腺癌, 临床表现, 胰腺肿块)和(胰腺癌, 临床表现, 肝转移)3 组关系。医疗领域文本中常见类似的实体密度高、句式冗长、实体关系交错互联的现象。
针对上述问题, 本文提出基于预训练模型和混合神经网络的医疗实体关系抽取模型。该模型采用预训练语言模型 BERT 动态地获取文本的词向量, 并提取实体标记(entity tagging)特征, 用双向长短期记忆网络(bidirectional long-short term memory, BiL-STM)获取医疗文本的全局上下文语义, 用 CNN 对医疗文本的局部信息特征进行抽象提取和表示, 最后通过注意力机制加权, 结合实体标记特征, 对文本关系进行分类。模型采用 BiLSTM 和 CNN 并行获取上下文语义, 兼具两类神经网络的优点, 兼顾全局和局部特征, 可以有效地减少医疗领域实体密集和句式冗长对关系抽取结果的影响。
早期的实体关系抽取大多在通用领域进行, 主要有基于词典的方法、基于规则的方法和基于模式匹配的方法。Miller 等[11]采用对实体信息词汇化的, 概率分布与上下文无关的语法解析器来生成规则, 进行实体关系抽取。基于词典和规则的方法依赖于词典和规则的质量, 并且迁移难度大, 无法应用到其他领域。传统的机器学习方法中有基于特征向量(feature-based)的方法, 通过自然语言处理工具提取文本中的重要特征(如依存关系和词块等), 进而描述实体之间的关系, 再采用机器学习模型(如最大熵和条件随机场)进行实体关系抽取。Zelenko 等[12]通过浅层句法分析设计树核函数, Culotta 等[13]通过依存句法捕获对象间的相似性来构造树核函数, Kim 等[14]采用基于特征的线性核函数方法抽取药物之间的关系。
随着深度学习技术的发展, 神经网络方法在通用领域的实体关系抽取中也有所突破。经典的实体关系抽取有基于 CNN 的方法、基于 RNN 的方法和基于 LSTM[15]的方法。基于 CNN 的方法通过卷积核对输入文本进行卷积操作, 编码文本信息, 提取文本的局部特征。Zeng 等[16]提出采用卷积神经网络来提取词汇和句子特征, 将这两级特征拼接作为特征向量进行关系抽取, 解决了预处理提取特征可能造成错误传播的问题。基于 RNN 的方法通过RNN 处理单元内部的前馈连接和反馈连接, 可以学习文本序列的上下文依赖特征。Socher 等[17]使用RNN 获取语句的内部特征和相邻短语信息特征进行关系抽取。基于 LSTM 的方法通过门控机制来提取文本的语义特征, 解决了 RNN 易出现梯度消失以及梯度爆炸的问题。注意力机制可以对输入特征进行加权, 通常结合神经网络进行关系抽取。闫雄等[18]将注意力机制与 CNN 相结合进行关系抽取, 使用注意力机制获取原始词向量之间的相互关系, 弥补了 CNN 提取特征能力不足的问题。
深度学习方法在医疗领域的实体关系抽取中也取得良好的效果。Liu 等[19]将 Text-CNN 与生物医学领域预训练词向量和位置向量相结合, 进行药物之间关系的提取, 缓解了生物医学文本中实体对分布密集的问题。关鹏举等[20]将 BiLSTM 网络的输出进行平均池化处理, 然后使用 Softmax 在临床医疗文本上进行关系分类, 取得良好的效果。Wei 等[21]将 BERT 预训练模型用于临床关系抽取, 也取得良好的效果, 证明了 BERT 在医疗关系抽取任务中的有效性。
本文提出的基于预训练模型与混合神经网络相结合的医疗实体关系抽取模型总体结构如图 1 所示, 分为 5 个步骤。
1)文本表示: 对于输入的医疗文本序列, 通过预训练模型 BERT 获取包含上下文特征的字向量, 作为混合神经网络的输入。
2)上下文信息建模: 采用双向的 LSTM, 获取文本中结合上下文信息的长期依赖特征。
3)局部特征建模: 对获得的字向量序列进行卷积和池化, 提取文本的局部特征。
4)特征加权: 利用注意力机制对序列重新加权, 给予较重要的特征向量较大的权重。
5)关系预测: 融合多种特征, 并且使用 Soft-Max 分类器计算每个关系的置信度, 进行关系类别 预测。
为获取输入序列文本的位置特征, 突出实体特征与实体关系抽取任务的相关性, 本文模型用
使用 BERT 获取文本的词向量, 将长度为 n的文本序列表示为 X = (x1, x2, …, xn)。在文本序列首尾分别添加“[CLS]”和“[SEP]”标志, 则 BERT 输出的文本序列向量可以表示为
V = BERT([CLS], x1, x2, …, xn, [ SEP])
= (e1, e2, …, en), (1)
其中, xi表示输入序列的第 i个文字, V为输入序列 X的向量化表示。
医疗文本的句式冗长问题导致提及实体在句子中的分布较为分散, 采用 BiLSTM 可以提取文本的上下文信息和深层语义信息。BiLSTM 对序列特征进行前向和后向两次 LSTM 训练优化, 捕获前后字词之间的语义关系, 增强语义信息, 保证提取特征的全局性和完整性, 缓解句式冗长的问题。作为一种特殊的 RNN 模型, LSTM 可以很好地提取文本序列信息, 其门控机制解决了用 RNN 提取长期依赖时梯度爆炸和梯度消失的问题。
在医疗关系抽取中, 头实体和尾实体的顺序不一定是前后顺序, 且序列后面的特征可能对序列前面的特征有影响。因此, 我们将前向 LSTM 和后向LSTM 拼接为 BiLSTM, 用来获取序列的上下文依赖信息, 提高模型对序列长期依赖特征的捕获能力。对于 BERT 文本表示层输出的序列特征 V=(e1, e2, …, en)使用 LSTM 单元进行处理, 计算过程如下:
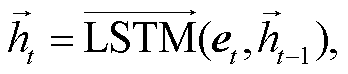 (2)
(2)
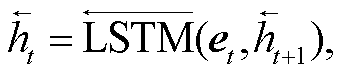 (3)
(3)

图1 本文模型的总体结构
Fig. 1 Overall structure diagram of the proposed model
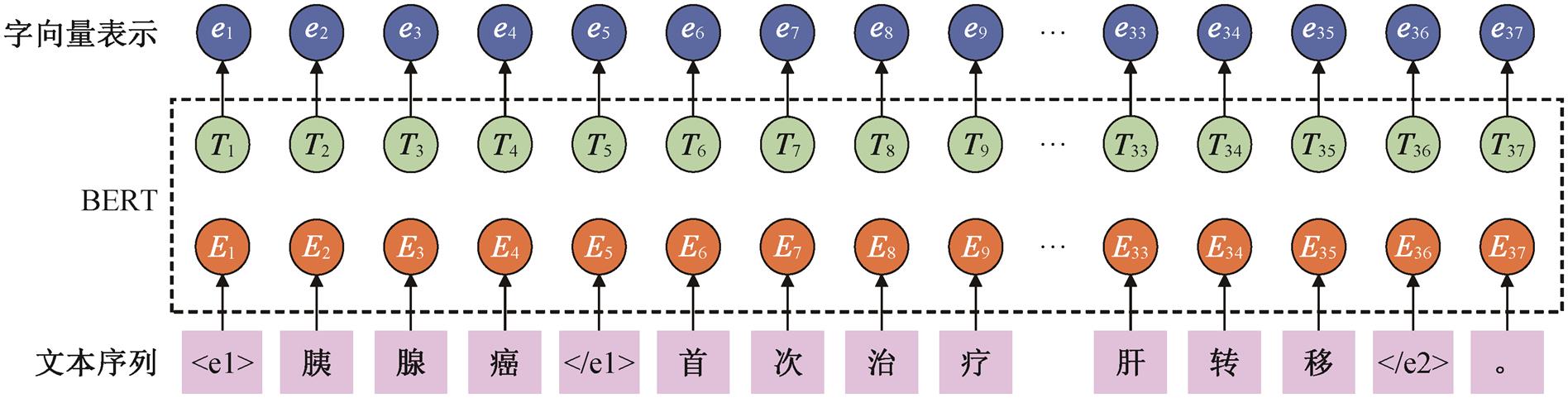
图2 文本向量化流程
Fig. 2 Text vectorization flow
其中, 输入向量为 et, 前向 LSTM 的隐藏状态为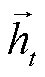 , 后向 LSTM 的隐藏状态为
, 后向 LSTM 的隐藏状态为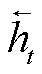 , 将两者拼接得到 BiL-STM 输出的表示为
, 将两者拼接得到 BiL-STM 输出的表示为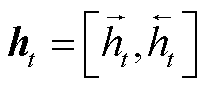 。对于隐藏单元数为u的 LSTM, BiLSTM 的输出定义为 H:
。对于隐藏单元数为u的 LSTM, BiLSTM 的输出定义为 H:
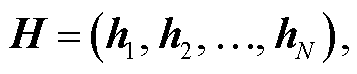 (4)
(4)
其中, N为隐藏单元个数, 大小为 2u。
在实体关系抽取任务中, 文本中每个实体的特征对模型预测最后的关系标签都非常重要, 因此提取文本局部的特征, 即不同字符、不同实体的特征非常必要, 而 CNN 是提取及合并局部特征的有效方法。医疗文本中存在较多的专业领域词汇, 关系抽取模型需要具有识别此类词汇的能力。与词级特征相比, 字符级特征表示有助于在关系抽取任务中识别未知术语。通过字符级特征表示, 可以有效地解决词语不规范问题。本文使用 CNN 提取字符级别的局部特征, 对 BERT 文本表示层输出的序列特征 V=(e1, e2, …, en)进行卷积操作, 提取特征序列中每个实体的局部特征。卷积特征 p的卷积操作计算过程如下:
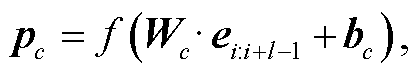 (5)
(5)
其中, f为 Relu 激活函数, e为输入的向量特征, i表示输入序列的第 I个特征, c为卷积核的数量, Wc表示卷积核的可训练权重参数, l为卷积核的长度, b为偏置参数。在进行卷积操作时, 在矩阵的边缘, 卷积核的长度可能会超出矩阵范围, 因此需要进行填充操作, 使矩阵的边缘可以作为卷积操作的中心, 获取更全面的局部特征。对卷积操作得到的特征向量, 需要进行降维, 常用的方式有最大池化和平均池化等。最大池化的过程就是提取卷积特征向量中固定范围内的最大值, 作为池化后的特征表示, 对于卷积特征 p, 其池化特征 c可以由下式表示:
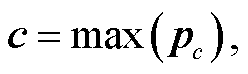 (6)
(6)
则用 CNN 提取序列局部向量后的特征向量可以表示为 S = (c1, c2, …, cn)。
医疗文本中存在医疗实体分布密集的问题, 对于提及实体的关系分类, 每个实体的重要性不同。注意力机制能关注对任务结果影响较大的特征, 考虑输入特征与结果的相关性。
对于输入长度为 n的特征序列 X = [x1, x2, …, xn], 有注意力概率分布 αi。使用 αi对输入特征进行加权求和, 计算公式如下:
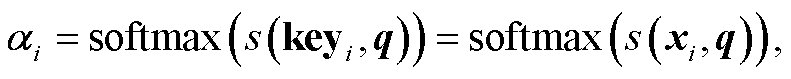 (7)
(7)
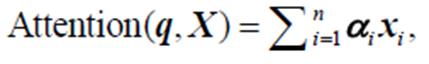 (8)
(8)
其中, s(xi, q)为注意力评分机制。注意力评分机制通常有加性模型、点积模型、缩放点积模型和双线性模型。本文采用点积模型, 计算公式如下:
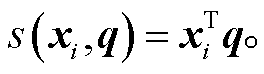 (9)
(9)
对于长度为 n的特征序列 X = [x1, x2, …, xn], 引入注意力机制后的输出为 r, 计算公式如下:
 (10)
(10)
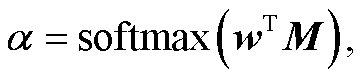 (11)
(11)
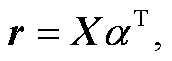 (12)
(12)
其中, w为可训练的权重参数。注意力层的输出结果 h*为特征加权求和的结果, 计算公式如下:
h*= sum(r)。 (13)
在关系预测层提取 BERT 文本表示层中实体标记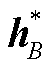 和 CNN-ATT 的输出
和 CNN-ATT 的输出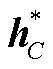 进行拼接, 结果为
进行拼接, 结果为
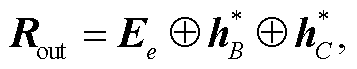 (14)
(14)
其中, ⊕表示向量拼接。使用 softmax 预测 Rout对应输入句子 X的关系类别标签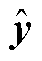 , 计算 X属于每类标签的条件概率
, 计算 X属于每类标签的条件概率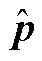 , 将最大的
, 将最大的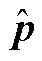 值作为 X的预测类别标签
值作为 X的预测类别标签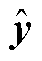 , 计算公式如下:
, 计算公式如下:
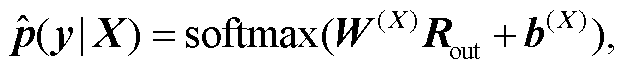 (15)
(15)
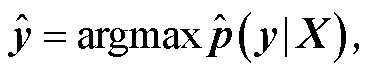 (16)
(16)
其中, W (X)为权重参数, b(X)为偏置参数。损失函数为多分类交叉熵。计算真实关系标签与预测关系标签的交叉熵, 作为损失函数, 计算公式如下:
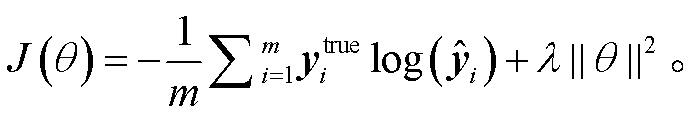 (17)
(17)
其中, m为真实关系类别标签数, 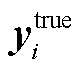 为当前句子X的真实标签, λ||θ||2为 L2 正则化项, λ为 L2 正则化的超参数。本文还选择 AdamW作为模型的优化器, 并在模型中加入 Dropout。
为当前句子X的真实标签, λ||θ||2为 L2 正则化项, λ为 L2 正则化的超参数。本文还选择 AdamW作为模型的优化器, 并在模型中加入 Dropout。
我们分别在中、英文医学文本实体关系抽取数据集上进行实验。
中文数据集来自 CHIP2020 的评测任务 2 (http: //www.cips-chip.org.cn/2020/eval2), 由郑州大学自然语言处理实验室和北京大学计算语言学教育部重点实验室联合构建, 包含儿科训练语料和百种常见疾病训练语料。儿科训练语料包含 518种儿科疾病; 百种常见疾病训练语料包含 109 种常见疾病, 其中包含 44 种关系类型, 其数量如表 1 所示。经去除停用词和文本字符正则化处理, 得到训练数据 43624条, 测试数据 10613 条。
英文医学数据集来自 SemEval2013 task9 的 DDI医学数据集, 由标注了药物与药物之间相互作用的文档组成, 其中 Medline 数据集由 Medline 和 Pub-Med 的文章摘要组成, 相互作用药物对中包含 232对正例和 1555 对负例。训练集包含 142 篇文档, 共有 1301 个句子和 1787 对相互作用药物对, 按照 8:2的比例划分, 得到训练数据 1430 条, 验证数据 375条。数据集定义的 4 种药物交互标签如表 2 所示。
本文将准确率 P(precision)、召回率 R(recall)和F1 值作为医疗实体关系抽取任务的评价指标。设ri为关系类别集合中的关系类型, 其 P, R和 F1 值的计算公式如下:
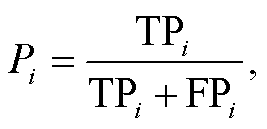 (18)
(18)
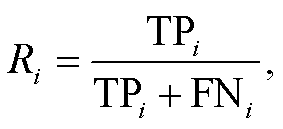 (19)
(19)
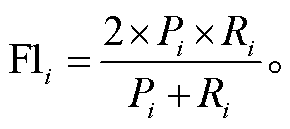 (20)
(20)
其中, TPi表示预测关系类型和真实关系类型均为ri的关系类型数量, FPi表示预测关系类型为 ri但真实关系类型不为 ri的关系类型数量, FNi表示预测关系类型不为 ri但真实关系类别为 ri的关系类型数量, TPi+FPi表示预测关系类型为 ri的总数量, TPi+FNi表示真实关系类型为 ri的总数量。
表1 CHIP2020数据集关系类型及数量[22]
Table 1 CHIP2020 dataset relation types and quantity[22]
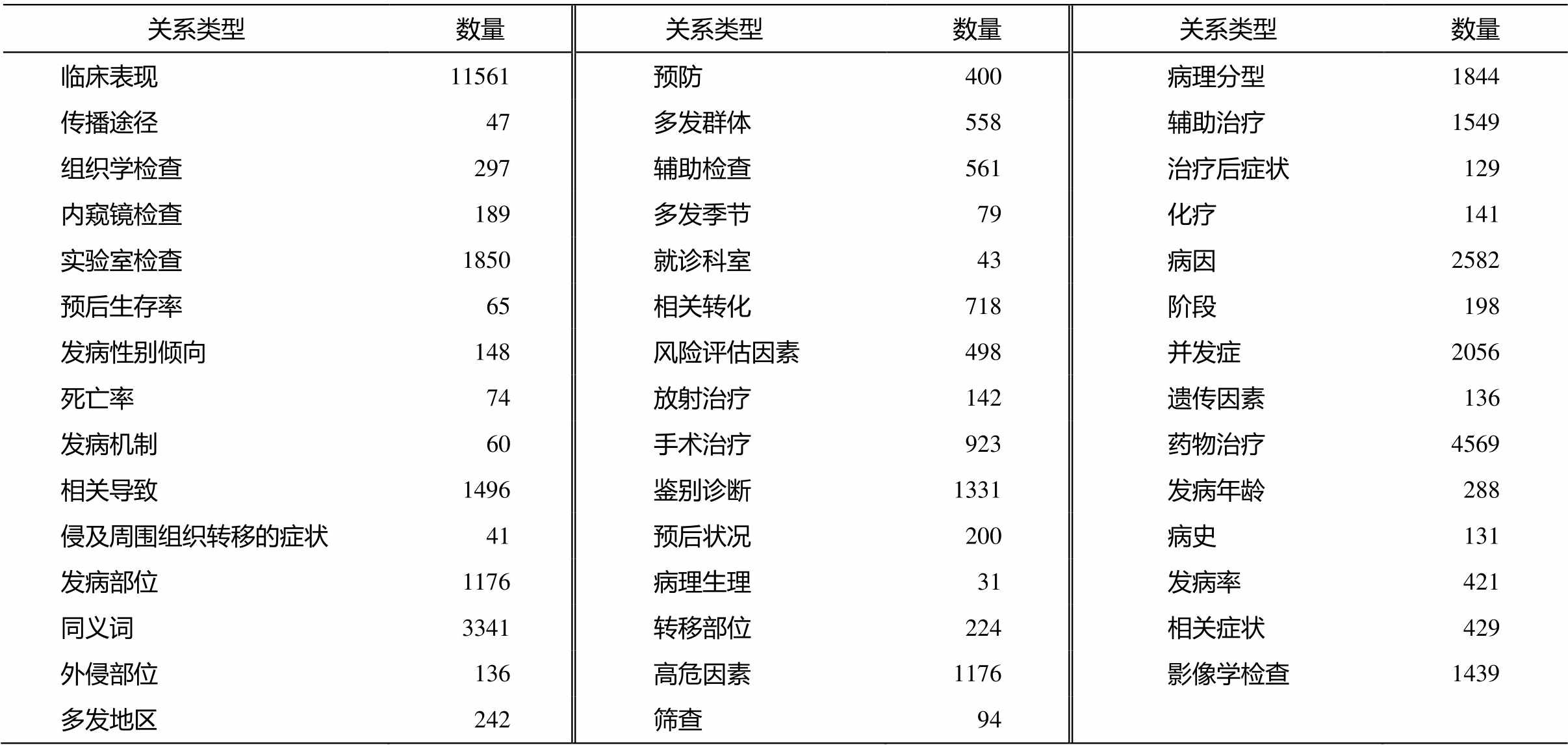
关系类型数量关系类型数量关系类型数量 临床表现11561预防 400病理分型1844 传播途径47多发群体 558辅助治疗1549 组织学检查297辅助检查 561治疗后症状 129 内窥镜检查189多发季节 79化疗 141 实验室检查1850就诊科室 43病因2582 预后生存率65相关转化 718阶段 198 发病性别倾向148风险评估因素 498并发症2056 死亡率74放射治疗 142遗传因素 136 发病机制60手术治疗 923药物治疗4569 相关导致1496鉴别诊断1331发病年龄 288 侵及周围组织转移的症状41预后状况 200病史 131 发病部位1176病理生理 31发病率 421 同义词3341转移部位 224相关症状 429 外侵部位136高危因素1176影像学检查1439 多发地区242筛查 94
表2 DDI数据集药物交互标签[23]
Table 2 Drug interaction labels in the DDI dataset[23]

标签含义数量 Mechanism描述药物相互作用的药物代谢动力学机制62 Effect描述药物相互作用的效果152 Advice描述有关同时使用两种药物的建议意见8 Interaction说明两种药物相互作用但没有提供有关作用的详细信息10
在 CHIP2020 数据集上使用权重平均(weighted average)的评价方式, 计算方法如下: 将 Pi, Ri和 F1i分别与各关系类型的占比相乘, 再对所有关系类型的数值求和, 得到最终结果。
在 DDI 数据集上使用微平均(micro average)的评价方式, 计算方法如下: 在分类结果中, 对各关系类型的 TPi, FPi和 FNi求平均值, 再计算总的 Pi, Ri和 F1i, 得到最终结果。
本文中, 设置实验模型的迭代轮数为 50。如果模型的指标在 10 轮之内没有得到优化, 则停止模型的训练。其他超参数设置如表 3 所示。
表3 超参数设置
Table 3 Hyperparameter settings
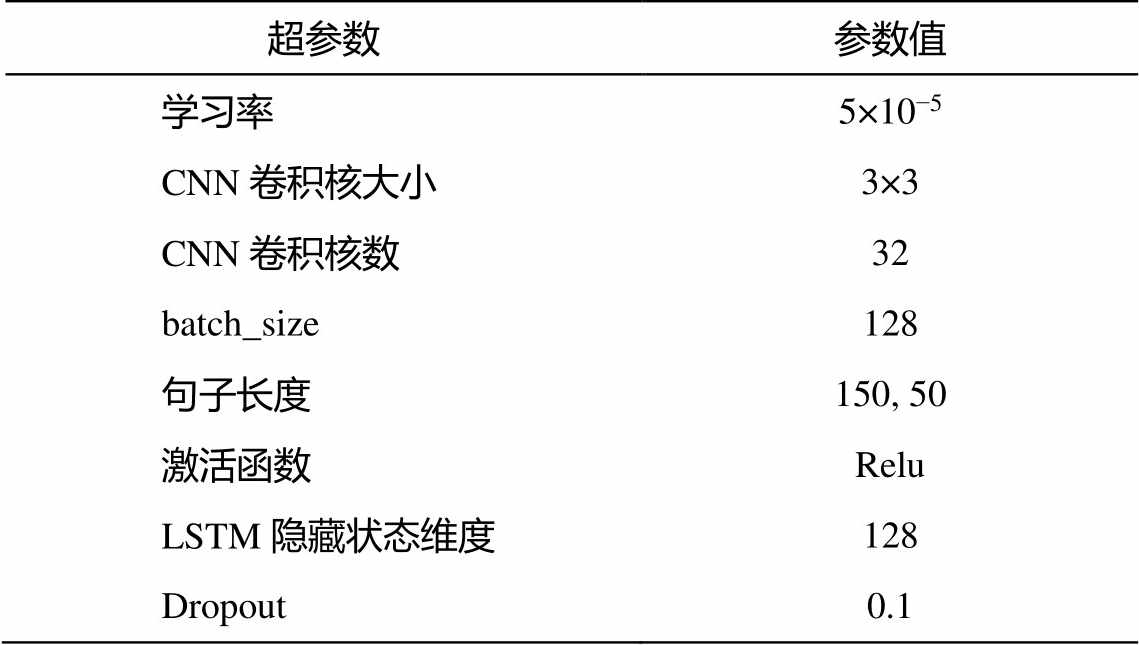
超参数参数值 学习率5×10−5 CNN卷积核大小3×3 CNN卷积核数32 batch_size128 句子长度150, 50 激活函数Relu LSTM隐藏状态维度128 Dropout0.1
说明: CHIP2020数据集的句子长度为150, DDI数据集的句子长度为50。
为评估本文模型的有效性, 将本文模型与医疗领域先进的实体关系抽取基准模型进行对比。
在 CHIP2020 数据集上进行实验时, 与下述基准模型进行对比, 结果见表 4。
表4 CHIP2020 数据集实验结果(%)
Table 4 Experimental results of CHIP2020 dataset (%)
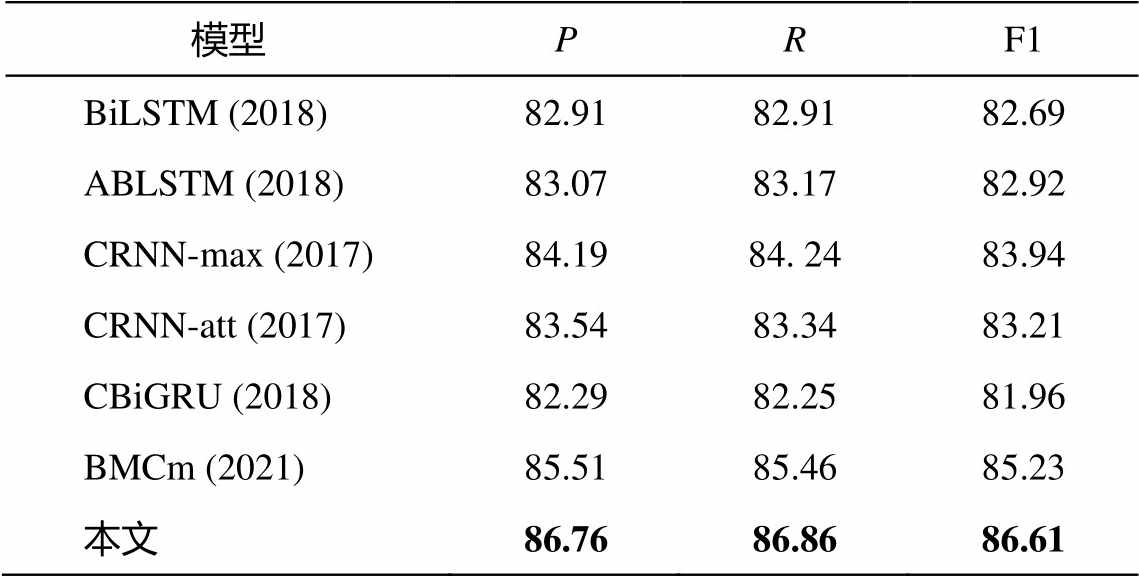
模型PRF1 BiLSTM (2018)82.9182.9182.69 ABLSTM (2018)83.0783.1782.92 CRNN-max (2017)84.1984. 2483.94 CRNN-att (2017)83.5483.3483.21 CBiGRU (2018)82.2982.2581.96 BMCm (2021)85.5185.4685.23 本文86.7686.8686.61
说明: 粗体数字表示最好的结果, 下同。
BiLSTM 和 ABLSTM[24]: 使用双向 LSTM 网络提取文本的上下文信息, 进行实体关系抽取。
CRNN-max 和 CRNN-att[25]: 结合 BiLSTM 与CNN, 分别提取上下文信息和局部特征, 然后进行最大池化和注意力池化, 再进行实体关系抽取。
CBiGRU[26]: 使用 CNN 和双向 GRU 提取局部特征和上下文信息, 进行实体关系抽取。
BMCm[27]: 使用 BiLSTM 来提取上下文语义信息, 并结合多通道注意力机制和 CNN 进行实体关系抽取。
实验结果显示, 在中文医疗实体关系抽取任务中, 本文模型的性能比基准模型好。本文模型能够利用 BiLSTM 提取医疗文本中的实体相关长期依赖关系, CNN 能够提取文本中实体相关的局部特征, 注意力机制能够针对不同特征进行加权来减弱噪声的影响。将不同特征进行融合, 可以有效地提高医疗实体关系抽取任务的性能。在对比的基线模型中, BiLSTM 和 ABLSTM 的性能一般, 因为该模型只利用上下文信息, 缺少局部特征的辅助, 降低了模型对医疗实体关系抽取的性能; CRNN-att 使用注意力池化对最终的特征进行加权, 忽略了模型误差在 BiLSTM 与 CNN 之间的传播, 降低了模型区分不同关系类型的性能; BMCm 模型使用 BiLSTM, CNN 和注意力机制, 但更侧重上下文依赖特征的提取。实验结果证明本文模型在网络结构上具有一定的优越性。
我们在 DDI 数据集上进行实验时, 与下述基准模型进行对比, 预训练模型 BERT 采用医疗领域的SciBERT[28]。除句子长度外, 模型的超参数均与在CHIP2020 中文数据集上的实验相同。表 5 展示实验结果中 4 种交互类型的 F1 值以及总体微平均的P, R和 F1 值。
PM-BLSTM[29]: 基于位置感知的深度多任务学习方法, 使用 BiLSTM 对医学文本进行编码。
RHCNN[30]: 获取文本中实体的语义嵌入和位置嵌入, 使用循环混合卷积神经网络获取特征, 抽取 DDI。
AGGCN[31]: 使用基于注意力机制的图卷积网络, 结合基于循环网络的编码器进行 DDI 抽取。
MEAA[32]: 基于多实体感知以及实体信息的DDI 提取模型, 采用 BioBERT 和 BiGRU 获取文本的特征。
DDMS[33]: 将药物的描述信息和药物的分子结构信息作为额外的辅助信息, 使用 sciBERT 获取文本向量表示, 使用图神经网络编码分子结构信息进行 DDI 抽取。
实验结果显示, 在英文医学实体关系抽取任务中, 本文所提模型依然有较好的性能, 交互类型中Effect 和 Interaction 取得最佳性能, 总体准确率也最高, 证明本文模型在英文医学关系抽取任务中能发挥较好的作用。与使用句法依存树信息的 AGGCN模型以及使用额外辅助信息的 DDMS 模型相比, 本文模型的总体性能超越 AGGCN, F1 值提高 6.69%,4 种交互类型中有 3 种的性能超越 AGGCN; 本文模型的总体性能逼近 DDMS, 交互类型中 Effect 和Interaction 的 F1 值超越 DDMS, 总体准确率超过DDMS, 证明本文模型不使用额外的外部信息就能达到较好的性能。
表5 DDI数据集实验结果(%)
Table 5 Experimental results of DDI dataset (%)
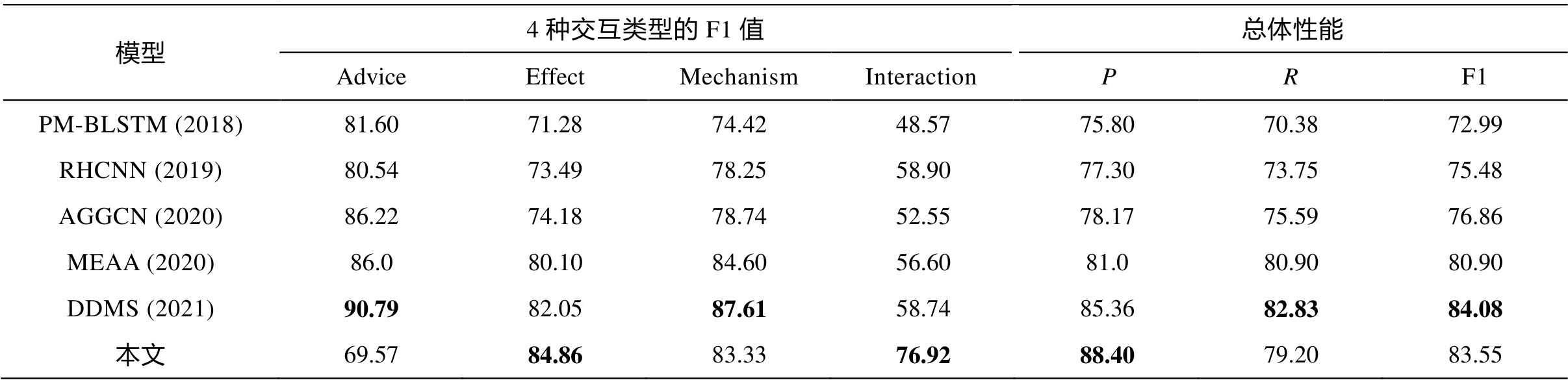
模型4种交互类型的F1值总体性能 AdviceEffectMechanismInteractionPRF1 PM-BLSTM (2018)81.6071.2874.4248.5775.8070.3872.99 RHCNN (2019)80.5473.4978.2558.9077.3073.7575.48 AGGCN (2020)86.2274.1878.7452.5578.1775.5976.86 MEAA (2020)86.080.1084.6056.6081.080.9080.90 DDMS (2021)90.7982.0587.6158.7485.3682.8384.08 本文69.5784.8683.3376.9288.4079.2083.55
为验证本文模型中各部分的作用, 进行各个模块的消融实验, 结果如表 6 所示。
通过移除本文模型中的上下文信息来建模层(BiLSTM), 验证其在医学关系抽取中的有效性。移除 BiLSTM 后, 在 CHIP2020 和 DDI 两个数据集上的 F1 值分别下降 2.4%和 1.42%, 表明移除 BiLSTM会降低模型预测实体关系的性能。使用 BiLSTM 提取文本的上下文信息, 能获取医学文本的长期依赖特征, 有助于模型去理解医学文本中的实体关系。在 DDI 数据集的实验结果中, 去除 BiLSTM 后, 交互类型中 Mechanism 和 Interaction 的 F1 值并未下降, 表明针对不同的交互类型, 不同的特征信息有不同的增益效果, 甚至会带来负面影响。
为验证本文模型中局部特征在医学关系抽取任务中的有效性, 我们在医疗关系数据集上进行局部特征提取层(CNN)的消融实验。移除 CNN 后, 模型预测医学实体关系的性能明显下降, 在 CHIP2020和 DDI 这两个数据集上的 F1 值分别降低 1.04%和1.23%, 表明在预测医学实体关系时, CNN能够提取实体的局部特征。这些局部特征能够帮助模型理解实体之间的关系, 有助于模型预测医学实体之间的关系。
注意力机制(ATT)能够对输入的特征进行加权处理, 使得模型能够对不同的特征给予不同的关注, 减少对预测关系类型作用较小的特征的影响。为验证本文模型中 ATT 的作用, 我们在不同的数据集上进行 ATT 的消融实验。移除 ATT 后, 在 CHIP2020和 DDI 两个数据集上的 F1 值分别下降 1.81%和1.36%。实验结果表明, 注意力机制可以针对不同的特征进行加权, 减少特征中噪声的影响, 提高模型对医学实体关系预测的性能。
同时去除 BiLSTM 和 ATT, 或同时去除 CNN 和ATT 后, 模型的性能进一步下降, 表明本文模型的所有组件对模型都有积极的贡献。
实体标记能够标记实体对在文本中的位置, 将实体的位置特征注入模型中。为验证实体标记特征在本文模型中的有效性, 我们进行实体标记特征的消融实验。移除实体标记特征后, 在 CHIP2020 和DDI 两个数据集上的 F1 值分别下降 4.39%和 1.37%, 证明实体标记特征能够将实体位置信息添加到模型中, 提升模型的性能。
从医学实体关系抽取数据集的测试集中选取部分实例, 将其在注意力层的权重进行可视化。图 3显示, 注意力权重可以很好地集中到提及实体附近; 描述实体之间关系的字词也有一定的权重; 在文本中添加的实体标识符可以标识实体的位置, 在注意力层中标识符也被分配较高的权重; 使用预训练模型 BERT 处理的文本被添加特殊的标识符, 标识符中的“[CLS]”蕴含文本整体的语义特征, “[SEP]”标识出文本句子的结束位置, 这两个标识符在注意力层也被分配部分权重。
图 4 显示本文模型在医学关系抽取测试数据集DDI 上的混淆矩阵, 从中可以看到以下错误: 1)4 种交互类型中, Mechanism, Effect 和 Advice 这 3 种经常被错误地划分划到无类别的负面实例(Negati-ve)类型; 2) Interaction 的实例经常被错误地划分到Effect 类型。
表6 不同数据集的消融实验结果(%)
Table 6 Results of ablation experiments in different data sets(%)
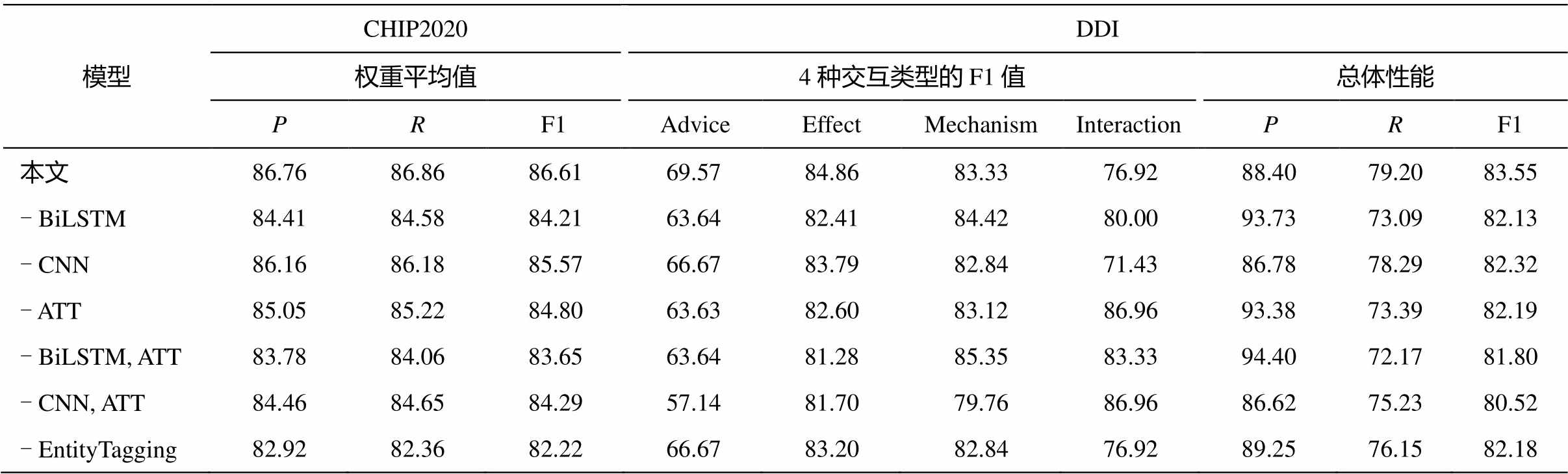
模型CHIP2020DDI权重平均值4种交互类型的F1值总体性能PRF1AdviceEffectMechanismInteractionPRF1 本文86.7686.8686.6169.5784.8683.3376.9288.4079.2083.55 - BiLSTM84.4184.5884.2163.6482.4184.4280.0093.7373.0982.13 - CNN86.1686.1885.5766.6783.7982.8471.4386.7878.2982.32 - ATT85.0585.2284.8063.63 82.6083.12 86.96 93.38 73.39 82.19 - BiLSTM, ATT83.7884.0683.6563.6481.2885.3583.3394.4072.1781.80 - CNN, ATT84.4684.6584.2957.1481.7079.7686.9686.6275.2380.52 - EntityTagging82.9282.3682.2266.6783.2082.8476.9289.2576.1582.18

颜色越深, 注意力权重越大
图3 注意力层权重的可视化
Fig. 3 Visualization of attentional layer weights
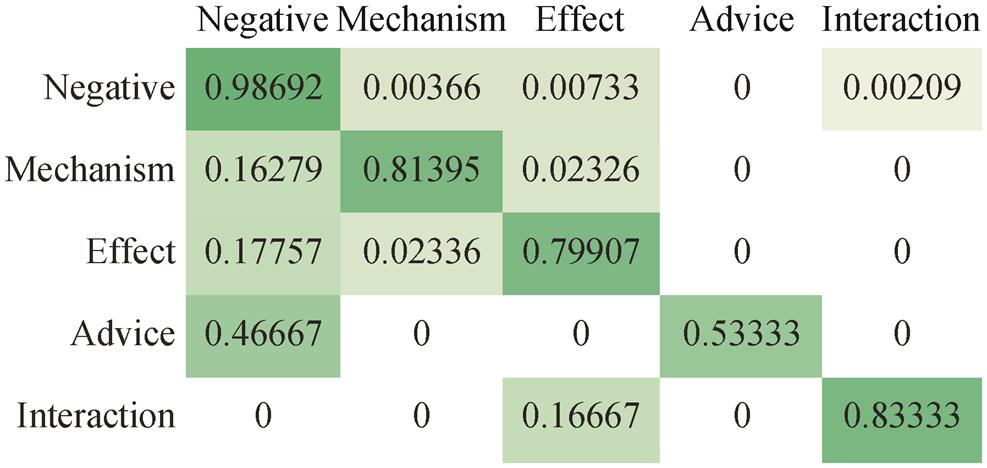
归一化数值; 颜色越深, 占比越大
图4 DDI数据集上的混淆矩阵
Fig. 4 Confusion matrix on DDI dataset
第一类错误主要由 DDI 关系抽取数据集中数据类型分布不平衡造成。DDI 训练数据集中共存在1787 个样本, 无类别的负例为 1555 条。数据标签数量的不平衡, 会造成将小数量的实例分类为大数量的实例。第一类错误的存在导致模型在进行关系抽取时, Mechanism, Effect 和 Advice 这 3 种类型的分类结果会得到较多的假负例(false negative, FN), 而假正例(false positive, FP)较少。例如, 在 Advice类型的测试结果中, FP 的占比为 0, FN 的占比为0.46667, 准确率为 1, 召回率为 0.5333。因此, DDI数据集上全部交互类型的微平均评价指标中, 准确率较高, 召回率较低。
第二类错误产生的主要原因是 Interaction 类型的数量较少, 且在部分实例中具有与 Effect 类型相似的语义。例如, 实例“Treatment with antidep-ressant drugs can directly interfere with blood glucoselevels or may interact with hypoglycemic agents .”为 Interaction 类型, 实例“Similarly, diazepam dec-reased the antinociceptive effect of metamizole (onlyin the tail-flick test) and indomethacin .”为 Effect 类型, 这两个实例都描述药物之间的关系, 且拥有相同的语义关系, 这两种交互类型文本之间语义的相似导致第二类错误分类。
本文提出一种面向医疗实体关系抽取任务的混合神经网模型, 使用预训练 BERT 获取文本的动态词向量, 解决静态词向量的一词多义问题。利用CNN 捕获句子的局部特征信息, 使用 BiLSTM 获取句子的上下文信息和长期依赖特征, 再分别经过加权的注意力机制获取全局的语义特征, 实现对医疗文本深层语义特征的获取。在中、英文实体关系抽取数据集上的对比试验和消融实验证明了模型的有效性。
后续工作中考虑将外部医学知识作为辅助信息添加到模型中, 以便获取更丰富的医学知识来辅助模型理解医疗文本。
参考文献
[1]Grishman R. Information extraction: techniques and challenges // Information Extraction, International Summer School. Frascati: Springer, 1997: 10‒27
[2]Liu Qiao, Li Yang, Duan Hong, et al. Knowledge graph construction techniques. Computer Research and Development, 2016, 53(3): 582‒600
[3]Pyysalo S, Airola A, Heimonen J, et al. Comparative analysis of five protein-protein interaction corpora. BMC Bioinformatics, 2008, 9(3): S3‒S6
[4]Wei C, Peng Y, Leaman R, et al. Assessing the state of the art in biomedical relation extraction: overview of the BioCreative V chemical-disease relation (CDR) task. Database: the Journal of Biological Databases and Curation, 2016, 64: baw032
[5]Segura-Bedmar I, Martínez P, Herrero-Zazo M. Les-sons learnt from the DDIExtraction-2013 shared task. Journal of Biomedical Informatics, 2014, 51: 152‒ 164
[6]Blaschke C, Valencia A. The frame-based module of the SUISEKI information extraction system. IEEE Intelligent Systems, 2002, 17(2): 14‒20
[7]LeCun Y, Bottou L, Bengio Y, et al. Gradient-based learning applied to document recognition. Procee-dings of the IEEE, 1998, 86(11): 2278‒2324
[8]Elman J L. Finding structure in time. Cognitive Sci-ence, 1990, 14(2): 179‒211
[9]Cho K, Merrienboer B, Gulcehre C, et al. Learning phrase representations using RNN Encoder-Decoder for statistical machine translation // Proceedings of the 2014 Conference on Empirical Methods in Natural Language Processing (EMNLP). Doha: Association for Computational Linguistics, 2014: 1724‒1734
[10]Devlin J, Chang M, Lee K, et al. BERT: pre-training of deep bidirectional transformers for language un-derstanding // Proceedings of the 2019 Conference of the North American Chapter of the Association for Computational Linguistics: Human Language Tech-nologies. Minneapolis, 2019: 4171‒4186
[11]Miller S, Fox H, Ramshaw L, et al. A novel use of statistical parsing to extract information from text // Proceedings of the 1st North American Chapter of the Association for Computational Linguistics Confern-ce. Seattle, 2000: 226–233
[12]Zelenko D, Aone C, Richardella A. Kernel methods for relation extraction. J Mach Learn Res, 2003, 3: 1083–1106
[13]Culotta A, Sorensen J S. Dependency tree kernels for relation extraction // Proceedings of the 42nd Annual Meeting of the Association for Computational Lingui-tics. Barcelona: ACL, 2004: 423‒429
[14]Kim S, Liu H, Yeganova L, et al. Extracting drug-drug interactions from literature using a rich feature-based linear kernel approach. J Biomed Informatics, 2015, 55: 23‒30
[15]Hochreiter S, Schmidhuber J. Long short-term me-mory. Neural Comput, 1997, 9(8): 1735‒1780
[16]Zeng Daojian, Liu Kang, Lai Siwei, et al. Relation classification via convolutional deep neural network // 25th International Conference on Computational Lin-guistics. Dublin: ACL, 2014: 2335‒2344
[17]Socher R, Huval B, Manning C D, et al. Seman- tic compositionality through recursive matrix-vector spaces // Proceedings of the 2012 Joint Conference on Empirical Methods in Natural Language Processing and Computational Natural Language Learning. Jeju Island: ACL, 2012: 1201‒1211
[18]闫雄, 段跃兴, 张泽华. 采用自注意力机制和CNN融合的实体关系抽取. 计算机工程与科学, 2020, 42(11): 2059‒2066
[19]Liu Shengyu, Tang Buzhou, Chen Qingcai, et al. Drug-drug interaction extraction via convolutional neural networks. [EB/OL]. (2016‒01‒31) [2021‒12‒ 0]. https://www.hindawi.com/journals/cmmm/2016/69 18381/
[20]关鹏举, 曹春萍. 基于BLSTM的临床文本实体关系抽取. 软件, 2019, 40(5): 159‒162
[21]Wei Qiang, Ji Zongcheng, Si Yuqi, et al. Relation extraction from clinical narratives using pre-trained language models // AMIA Annual Symposium procee-dings. Washington, DC, 2019: 1236–1245
[22]Guan Tongfeng, Zan Hongying, Zhou Xiabing, et al. CMeIE: construction and evaluation of chinese med-cal information extraction dataset // Natural Language Processing and Chinese Computing, 9th CCF Interna-ional Conference (NLPCC 2020). Zhengzhou: Spri-ner, 2020: 270‒282
[23]Segura-Bedmar I, Martinez P, Herrero-Zazo M. SemEval-2013 task 9 : extraction of drug-drug inte-racions from biomedical texts (DDIExtraction 2013) // Proceedings of the 7th International Workshop on Semantic Evaluation. Atlanta: The Association for Computer Linguistics, 2013: 341‒350
[24]Sahu S K, Anand A. Drug-drug interaction extraction from biomedical texts using long short-term memory network. J Biomed Informatics, 2018, 86: 15‒24
[25]Raj D, Sahu S, Anand A. Learning local and global contexts using a convolutional recurrent network model for relation classification in biomedical tex // Proceedings of the 21st Conference on Computational Natural Language Learning (CoNLL 2017). Vanco-ver: Association for Computational Linguistics, 2017: 311‒321
[26]He Bin, Guan Yi, Dai Rui. Convolutional gated recurrent units for medical relation classification // IEEE International Conference on Bioinformatics and Biomedicine (BIBM 2018). Madrid: IEEE Computer Society, 2018: 646‒650
[27]张世豪, 杜圣东, 贾真, 等. 基于深度神经网络和自注意力机制的医学实体关系抽取. 计算机科学, 2021, 48(10): 77‒84
[28]Beltagy I, Lo Ke, Cohan A. SciBERT: a pretrained language model for scientific text // Proceedings of the 2019 Conference on Empirical Methods in Na-tural Language Processing and the 9th International Joint Conference on Natural Language Processing 9EMNLP-IJCNLP 2019). Hong Kong: Association for Computational Linguistics, 2019: 3613‒3618
[29]Zhou Deyu, Miao Lei, He Yulan. Position-aware deep multi-task learning for drug-drug interaction extrac-tion. Artif Intell Medicine, 2018, 87: 1‒8
[30]Sun Xia, Dong Ke, Ma Long, et al. Drug-drug inte-raction extraction via recurrent hybrid convolutio- nal neural networks with an improved focal loss [EB/OL]. (2019‒01‒08) [2021‒12‒15]. https://www. mdpi.com/1099-4300/21/1/37
[31]Park C, Park J, Park S. AGCN: attention-based graph convolutional networks for drug-drug interaction ex-traction. Expert Syst Appl, 2020, 159: 113538
[32]Zhu Yu, Li Lishuang, Lu Hongbin, et al. Extracting drug-drug interactions from texts with BioBERT and multiple entity-aware attentions. J Biomed Informa-tics, 2020, 106: 103451
[33]Asada M, Miwa M, Sasaki Y. Using drug descriptions and molecular structures for drug-drug interaction extraction from literature. Bioinform, 2021, 37(12): 1739‒1746
Medical Entity Relation Extraction Based on Pre-trained Model and Hybrid Neural Network
Abstract Medical text has high entity density and verbose sentence structure, which makes the simple neural network methods unable to capture its semantic features. Therefore, a hybrid neural network method based on pre-trained model is proposed. Firstly, a pre-trained model is used to obtain the dynamic word vector and the entity tagging features are extracted. Secondly, the contextual features of the medical text are obtained through a bidirectional long and short-term memory network. Simultaneously, the local features of the text are obtained using the convolutional neural network. Then the global semantic features of the text are obtained by weighting the sequence features through the attention mechanism. Finally, the entity tagging features are fused with the global semantic features and the extraction results are obtained through the classifier. The experimental results of entity relation extraction on the medical domain dataset show that the performance of the proposed hybrid neural network model is improved compared with the mainstream models, which indicates that this multi-feature fusion method can improve the effect of entity relation extraction.
Key words medical text; relation extraction; hybrid neural network; pre-trained model