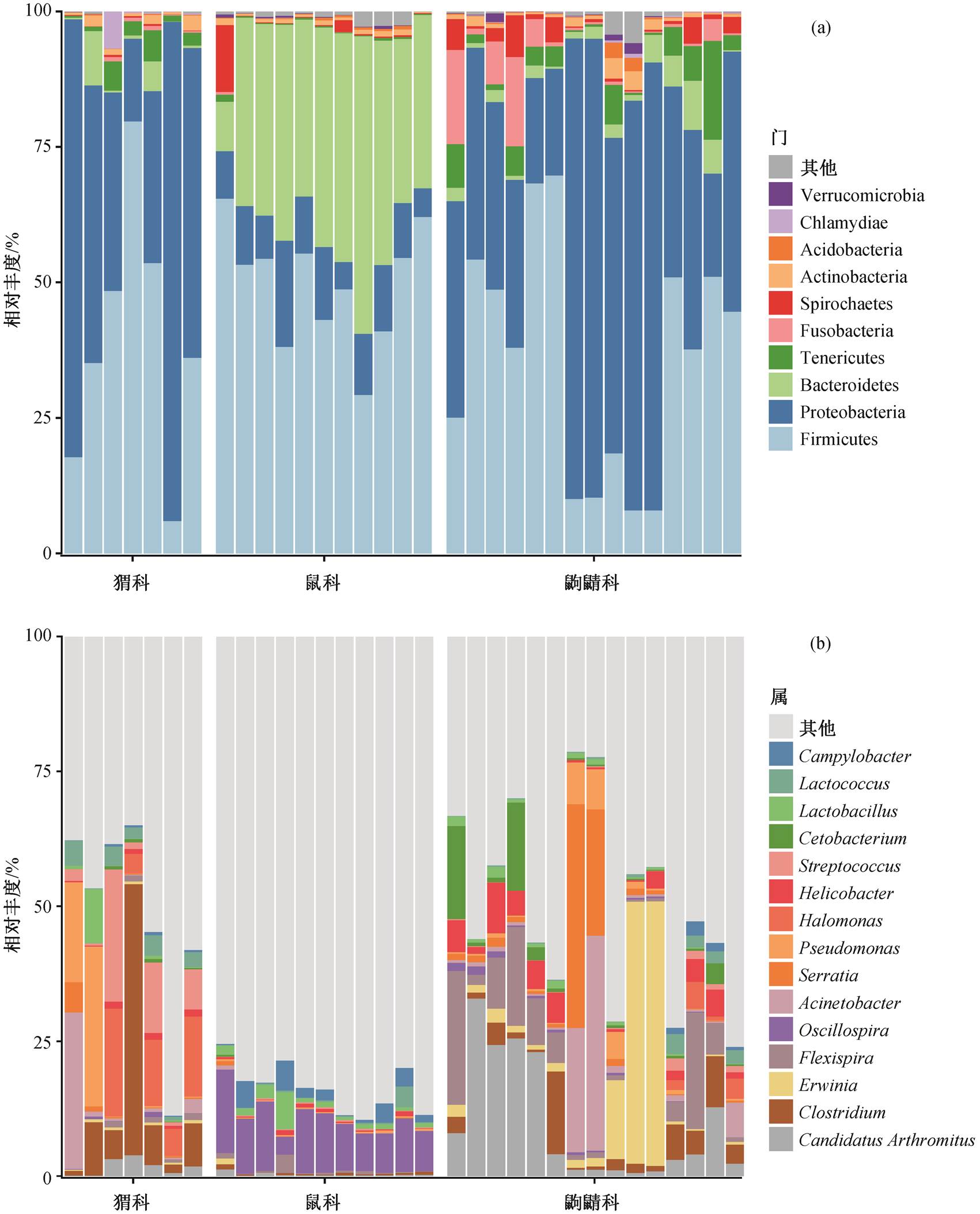
图1 肠道微生物在各样本中门水平(a)和属水平(b)的相对丰度
Fig. 1 Composition of the gut microbiota in the all samples at phylum (a) and genus (b) level
摘要 以生活在同一生境, 但具有不同进化关系和不同食性的野生哺乳类动物(鼠科、猬科和鼩鼱科)为研究对象, 通过 16S rRNA 基因扩增子测序, 分析和比较其肠道菌群。共识别出 5378 个操作分类单位(OTUs), 主要隶属 Firmicutes (40.55%), Proteobacteria (34.60%)和Bacteroidetes (13.67%)。Firmicutes 和 Bacteroidetes 是鼠科的优势菌门, Proteobacteria 是猬科和鼩鼱科的优势菌门。多样性分析表明, 鼠科、猬科与鼩鼱科的肠道微生物多样性及群落结构具有显著性差异; LEfSe 分析表明, 鼠科中存在更多与复杂碳水化合物发酵相关的细菌, 猬科和鼩鼱科中含较多氨基酸发酵菌; 益生菌(如 Lactobacillus 和 Lactococcus 等)共存于这 3 类野生小型哺乳类动物中, 起调控宿主健康的作用。研究结果揭示, 宿主食性与进化关系影响着野生小型哺乳动物肠道菌群结构, 而肠道菌群可能在多种方面对宿主起益生作用。
关键词 野生小型哺乳动物; 肠道菌群; 16S rRNA基因扩增子测序; 宿主饮食
肠道微生物不仅在保持肠道健康、促进肠道发育中扮演重要角色[1], 也影响着宿主的消化[2]、免疫[3]、发育[4]和行为特征[5]等。近年来, 越来越多的研究表明, 动物在适应逆境或新环境时, 除基因组发挥主要作用外, 其与肠道微生物组的长期协同进化对这一适应过程也起着促进作用[6]。肠道菌群可以促进野生动物适应特定饮食的生境[7], 并有助于在寒冷环境中维持动物的正常体温[8], 甚至可以帮助野生动物防御病原体感染[9]。肠道菌群的研究已经逐渐应用到野生动物保护领域[10], 了解动物肠道微生物群落组成和动态调控, 有助于理解宿主与肠道微生物复合体如何适应环境变化的响应机制, 对进一步开展流行病学及野生动物保护方面的研究具有十分重要的意义[11]。
生物学家对野生小型哺乳类动物的研究主要通过生态学特性、行为特征、功能基因分析以及转录组分析来研究其动物起源、系统发育、种群遗传和生物地理学特征等[12], 对其肠道微生物的研究集中在野生小家鼠[13–14]、野生木鼠[15]以及海南多种鼠类[16]上, 目前关于其他野生小型哺乳类动物肠道微生物的研究寥寥无几。随着肠道菌群的重要性逐渐为人所知以及分子测序技术的快速发展, 以 16S/ 18S rRNA 基因或 ITS 区域为目标序列的菌群多样性组成测序分析结果为全面解析野生动物生理生化特性提出新见解。
鼠科是起源最古老的鼠科类群, 该类动物分布广泛, 可以引起并传播人兽共患病, 多为小型的植食性动物, 盲肠较为发达[17]。猬科在我国是稀有物种, 属于杂食性动物, 取食节肢动物等无脊椎动物, 也取食果实种子[18]。鼩鼱科是哺乳动物第四大科, 我国鼩鼱科物种十分丰富[19], 大多以动物为食, 有些物种还会以植物的茎叶以及果实为食[20]。猬科和鼩鼱科由于缺乏盲肠, 肠道相对较短, 具有迅速排空食物的能力[21]。目前, 在物种进化过程中, 鼠科、猬科和鼩鼱科的肠道微生物适应性演化尚未得到揭示, 而肠道微生物对宿主有着非常重要的生物学意义。因此, 本研究选取鼠科(啮齿目, Rodentia)、猬科(食虫目或劳亚食虫目, Eulipotyphla)以及鼩鼱科(食虫目或劳亚食虫目, Eulipotyphla)为研究对象, 采用 16S rRNA 基因扩增子测序技术, 分析处于同一生境的鼠科、猬科和鼩鼱科的肠道微生物菌群多样性、组成与差异, 以期有助于理解宿主进化、饮食与肠道微生物之间的关联。
鼠科(Muridae, Rodentia)、猬科(Erinaceidae, Eulipotyphla)和鼩鼱科(Soricidae, Eulipotyphla)样品采集于云南哀牢山生态站。该生态站位于哀牢山国家级自然保护区北段的云南省景东县境内, 地理坐标为 101°02′E, 24°32′N, 海拔 2450m, 地处西南季风气候区, 属亚热带山地气候, 全年无夏, 春秋相连[22]。
采用谢尔曼(Sherman)陷阱法和皮特法尔(Shrew)陷阱法[23], 放置燕麦或黄粉虫, 于秋季连续一周夜间进行诱捕, 捕获后的野生小型哺乳类动物立即进行解剖, 取其肠道及内容物置于乙醇中, 提取 DNA 之前在−20°C 条件下保存。
在通风橱中风干样品, 以便去除酒精, 取 100~ 200mg 肠道内容物, 利用 DNeasy Powersoil 试剂盒(QIAGEN, Gernamy)提取总 DNA, 然后用 Qubit(In-vitrogen, USA)和凝胶电泳检查基因组 DNA 的质量和浓度。用引物 515F(GTGYCAGCMGCCGCGGT AA)和 806R (GGACTACNVGGGTWTCTAAT)扩增16S rRNA 基因的 V4 可变区。使用 QIAquick PCR纯化试剂盒(QIAGEN, Germany)纯化扩增子, 并在Illumina HiSeq 平台上测序。
将数据导入 QIIME2, 使用 DADA2 插件, 根据质量图对序列进行过滤和修剪, 聚类分配成操作分类单位(OTU)[24]。使用 R 软件的 LULU 包, 去除不正确的 OTU 序列, 并将相似序列合并[25], 重新导入QIIME2, 选择 mafft 比对序列命令, 创建比对的序列, 并除去高度可变的位置[26], 利用 FASTTREE 生成系统发育树[27]。利用 classify-sklearn 命令, 将 OTU比对到预先经过训练的 Greengenes 13_8[28]参考集上, 进行序列物种分类注释[29]。在 R 版本 3.6.3 中使用 microbiome 包, 计算 α 多样性指数(Chao1 指数和 Shannon 指数)[30], 采用 Wilcoxon 秩和检验方法, 分析 α 多样性指数在不同样品组之间是否具有显著性差异[31]。使用 vegan 包 ordinate 函数, 基于 OTU计算 β 多样性指数(方法包括非度量多维标定法(non-metric multidimensional scaling, NMDS)和主坐标分析法(principal co-ordinates analysis, PCoA), 距离计算采用 Bray-Curtis 相异度和 weighted UniFrac 距离)[32], 通过 adonis 函数中的置换多元方差分析(PERMANOVA)方法, 检验 3 个物种科的肠道微生物组成之间是否存在显著差异, 通过 ANOSIM (an-alysis of similarities, 相似性分析)检验组间差异与组内差异的关系[33]。采用 ampvis2 包, 计算核心微生物 Venn 图(相对丰度超过 0.1%且在不同组样品中共现率不小于 75%称为“核心 OTU”)[34]。采用 LEfSe分析肠道微生物在 3 种野生小型哺乳类动物中的差异物种[35]。使用 PICRUSt2 软件进行 KEGG Orthology(KO)预测[36], 并折叠至第二水平代谢通路上, 然后将结果导入元基因组概况统计分析(STAMP)软件中进行统计分析。使用非参数检验分析组与组之间差异代谢通路, 并对 p 值进行 Benjamini-Hochberg FDR多重检验, 以便控制假阳性[37]。
共测定 33 个样品, 其中包含 11 个鼠科样品、7个猬科样品和 15 个鼩鼱科样品。通过 16S rRNA 基因扩增子测序, 共获得 3196024 条序列(平均每个样品中有 96849 条序列), 聚类为 5378 个 OTUs (去除叶绿体与线粒体), 注释到 43 门 116 纲 200 目 257 科447 属 217 种。随着测序深度不断加深, 所有样品的稀释曲线趋于平缓(附录 1)(访问 http://xbna.pku. edu.cn 查看附录), 说明本次实验的数据量可靠。
不同宿主的肠道菌群结构和组成分析如图 1 所示, 平均相对丰度排前 5 位的门为 Firmicutes (厚壁菌门, 40.55%)、Proteobacteria (变形菌门, 34.60%)、Bacteroidetes (拟杆菌门, 13.67%)、Cyanobacteria (蓝菌门, 2.68%)和 Tenericutes (软壁菌门, 2.56%), 平均相对丰度超过 1%的门有 8 个。在属水平上, 注释到的相对丰度较高的菌为 Candidatus Arthromitus(Firmicutes, 4.72%)、Erwinia (Proteobacteria, 3.96%)、Flexispira (Firmicutes, 3.51%)、Oscillospira (Firmicut-es, 3.45%)和 Acinetobacter (Proteobacteria, 3.40%)等, 其中相对丰度超过 0.1%的属有 54 个, 主要来自Firmicutes 和 Proteobacteria。结果显示, Firmicutes, Bacteroidetes 和 Proteobacteria 是鼠科、猬科和鼩鼱科的主要优势菌门。在鼠科中, Firmicutes (49.46%)和 Bacteroidetes (35.70%)的相对丰度较高, 两者相对丰度之和超过 85%, 其中 Oscillospira, Ruminoco-ccus 和 Coprococcus 等是鼠科中 Firmicutes 门的重要组成属, Bacteroidetes 门中主要注释到的属为 Pre-votella 和 Bacteroides, 在 Bacteroidetes 中存在大量未分类到属的物种, 并且相对丰富度较高, 说明野生小型哺乳类动物肠道中的 Bacteroidetes 可能存在现存数据库中未收录的新属。在猬科和鼩鼱科中, Proteobacteria 的相对丰度较高, 分别为 44.96%和47.45%, 其次是 Firmicutes (36.15%和 36.07%)。虽然在门水平猬科与鼩鼱科的组成相似, 但是属水平的组成有差异, 猬科中的 Proteobacteria 门以 Acine-tobacter 和 Pseudomonas 为主, Firmicutes 门以 Clos-tridium 和 Streptococcus 为主; 而 Erwinia 以及 Flexi-spira 是鼩鼱科中 Proteobacteria 门的重要组成部分, Firmicutes 门以 Candidatus Arthromitus 属为主。除此之外, Cyanobacteria (11.42%)门在猬科中的相对丰度较高, 鼩鼱科中 Tenericutes (4.31%)和 Fusobac-teria (3.76%)相对丰度较高。上述结果说明, 不同科宿主的肠道菌群组成存在一定的差异。
本研究发现, Firmicutes 是野生小型哺乳动物的主要分类群, 其次是 Proteobacteria 和 Bacteroidetes, 与 Suzuki 等[14]关于哺乳动物肠道细菌组成的研究结果一致。来自 Firmicutes 和 Bacteroidetes 的细菌负责多种作用, 包括代谢产物的产生、免疫系统成熟、血管生成和脂肪储存。Proteobacteria 与能量积累有关[38], 编码多种功能, 其中最突出的包括蛋白质和氨基酸代谢, 碳水化合物代谢以及辅因子、维生素、辅基、色素代谢[39]。我们的发现符合目前人们的认识, 即哺乳动物的肠道中含有高度受限的细菌门, 以便适应胃肠道环境。Firmicutes 与碳水化合物、多糖和脂肪酸的分解有关, 它们被用做宿主的能源[40]。许多 Firmicutes 和 Bacteroidetes 的分离物被证明可以发酵分解植物的细胞壁成分[41], 已有研究发现 Proteobacteria 在狗猫等肉食性动物胃肠道中的丰度较高, 在健康的猫和狗的胃肠道中维持氧稳态[39]。因此, 植食性的鼠科中含有较多的Firmicutes 和 Bacteroidetes, 可能用于促进宿主消化复杂碳水化合物, 而 Proteobacteria 可能有利于以肉食性为主的猬科和鼩鼱科的肠道稳态。
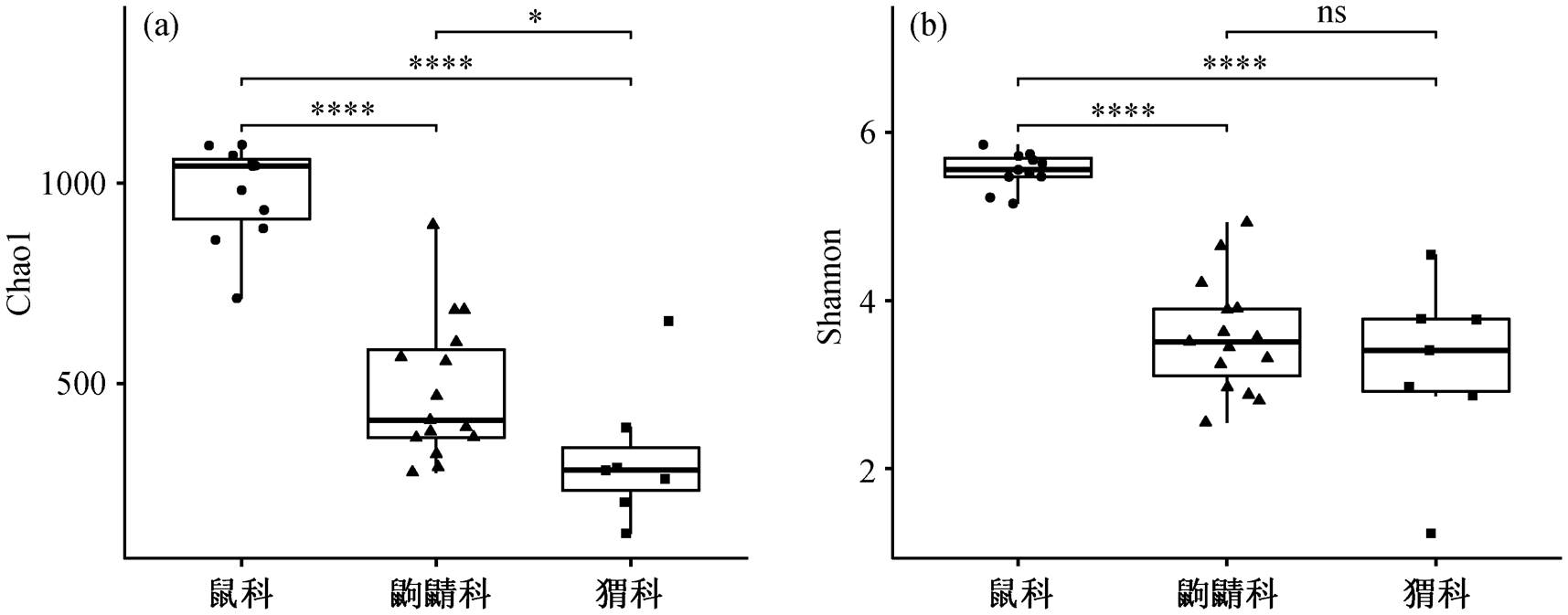
肠道菌群多样性分析主要包括 α 多样性和 β 多样性。Chao1 指数反映菌群的丰富度, 其值越高表明菌群物种的丰富度越高; Shannon 指数反映菌群的多样性, 其值越高表明菌群的多样性越高。如图2 所示, 鼠科的 Chao1 指数和 Shannon 指数均高于猬科和鼩鼱科, 猬科的丰富度和多样性均最低。Wilcoxon 检验表明鼠科与猬科、鼩鼱科之间存在显著性差异, Chao1 指数显示猬科与鼩鼱科之间存在差异, 而 Shannon 指数没有显著性差异, 说明鼠科具有更高的肠道菌群丰富度和多样性, 其次是鼩鼱科, 而猬科与鼩鼱科之间有一定的丰富度差异, 但没有多样性差异。Klimenko 等[42]发现 α 多样性和部分微生物丰度与长期的水果或蔬菜饮食成正比, Nelson 等[43]认为摄入植物中的复杂碳水化合物(如纤维素和淀粉)是导致草食动物肠道细菌群落丰富的驱动力。在哺乳动物的肠道中, 植食性动物的细菌丰富度最高, 其次是杂食动物和肉食性动物[44]。本研究同样发现植食性的鼠科肠道菌群具有较高的 α 多样性。

图1 肠道微生物在各样本中门水平(a)和属水平(b)的相对丰度
Fig. 1 Composition of the gut microbiota in the all samples at phylum (a) and genus (b) level
*p<0.5, ****p <0.001
图2 基于 OTU 构造的 α 多样性(Chao1 指数(a)和 Shannon 指数(b))
Fig. 2 Alpha diversity based on OTUs (Chao1 index (a) and Shannon index (b))
为进一步探究宿主与肠道菌群组分的关系, 我们通过计算 Bray-curits 距离和 Unweighted UniFrac距离, 采用主坐标分析(PCoA)和非度量多维尺度分析(NMDS)方法研究鼠科、猬科和鼩鼱科肠道菌群的 β 多样性(图 3)。PCoA 图显示, PCo1 作为第一主坐标轴解释样品中所有差异的 26.2%, PCo2 解释样品中所有差异的 14.1%, 这 3 个不同科的样本中, 鼠科所有样品间距离更近, 单独聚为一簇, 而猬科与鼩鼱科的样品较为分散。PERMANOVA 分析发现, 不同物种的野生哺乳动物肠道菌群之间存在显著差异(Bray-curits 距离: F = 7.11, R2 = 0.31, p <0.001; weighted UniFrac 距离: F = 14.46, R2 = 0.49, p<0.001)。对组与组之间进行两两比较发现, 鼠科与猬科之间的组成差异解释度最大, 其次为鼠科与鼩鼱科(附录 2), 说明鼠科、猬科与鼩鼱科的肠道菌群组成有显著差异, 猬科与鼩鼱科之间的组成更为相似。母华强等[18]的研究表明, 猬科和鼩鼱科属于杂食性动物, 其菌群会根据食物资源差异而做出相应的调整。David 等[45]发现饮食能够在短期内改变肠道微生物组成, 可能造成个体之间肠道菌群的差异较大, 因此猬科与鼩鼱科的样品较为分散。另外, 我们通过相似性分析 (analysis of similarities, ANOSIM)发现, 各样品组间差异大于组内差异, 说明虽然存在样品组内个体差异, 但其影响较小(R2 = 0.9187, p < 0.001)。
LEfSe 分析通过在组间寻找具有统计学差异的生物标记物来识别组间不同物种丰度的特征及其关联的类别。我们使用 LEfSe 对鼠科、猬科和鼩鼱科的肠道微生物差异性进行分析。本研究中共得到288 个生物标记物在这 3 个组间具有显著差异。如图 4 所示, 在门水平上, 主要有 Proteobacteria, Bacteroidetes, Tenericutes 和 Fusobacteria 等在 3 组的相对丰度显著不同。鼠科的肠道菌群以高碳水化合物发酵细菌为主导, 例如 Prevotella, Lactoba-cillus, Ruminococcus, Coprococcus, Dorea, Butyri-cicoccus, Oscillospira 和 Bilophila 等, 这些属主要来自 Bacteroidetes 门、Ruminococcaceae 科以及 Lach-nospiraceae 科, 其中 Bacteroidetes 门的物种几乎都在鼠科中丰度较高, 尤其是 S24-7 科[46]。研究表明Prevotella, Oscillospira 以及 Ruminococcus 与高纤维饮食有关, 具有降解纤维素和抗性淀粉等复杂碳水化合物的能力, 是降解纤维的细菌[47–50]。在鼠科中相对丰度显著较高的这些属具有通过发酵碳水化合物来产生 SCFA(短链脂肪酸)的能力。在猬科中, Clostridium, Turicibacter, Achromobacter, St-reptococcus 和 Actinetobacter 等具有统计学差异; 在鼩鼱科中, Faecalibacterium, Phascolarctobacterium, Cetobacterium, Erwinia, Helicobacter 和 Fusobacteri-um 等具有统计学差异, 这些属主要属于 Proteobac-teria, Tenericutes 和 Fusobacteria 门。猬科和鼩鼱科中某些细菌的丰度变化可能反映动物摄入的蛋白质增加, 碳水化合物和纤维摄入减少。例如, 猬科中具有较高丰度的 Clostridium, 该属是主要的蛋白水解细菌, 能够利用蛋白质产生更多的 SCFA[51], 并且在消耗较多蛋白质和脂肪的人和牛中被观察到[52]。Fusobacterium 在狗和海洋肉食动物中都具有较高的相对丰度, 通过发酵氨基酸产生各种有机酸(如乙酸和丁酸等)[53]。不同目(啮齿目和食虫目)之间肠道微生物组成差异较大, 属于同一目的猬科和鼩鼱科, 虽然有相似的饮食习惯, 但具有不同的进化关系, 其肠道微生物在属水平上仍有一定的差异。通过随机森林法和非参数法检验不同宿主科的差异菌属, 其结果具有一致性(附录 3), 说明饮食可能对哺乳动物肠道菌群有重要影响, 但宿主遗传学在塑造宿主肠道菌群的系统发育规模上仍然具有重要作用。
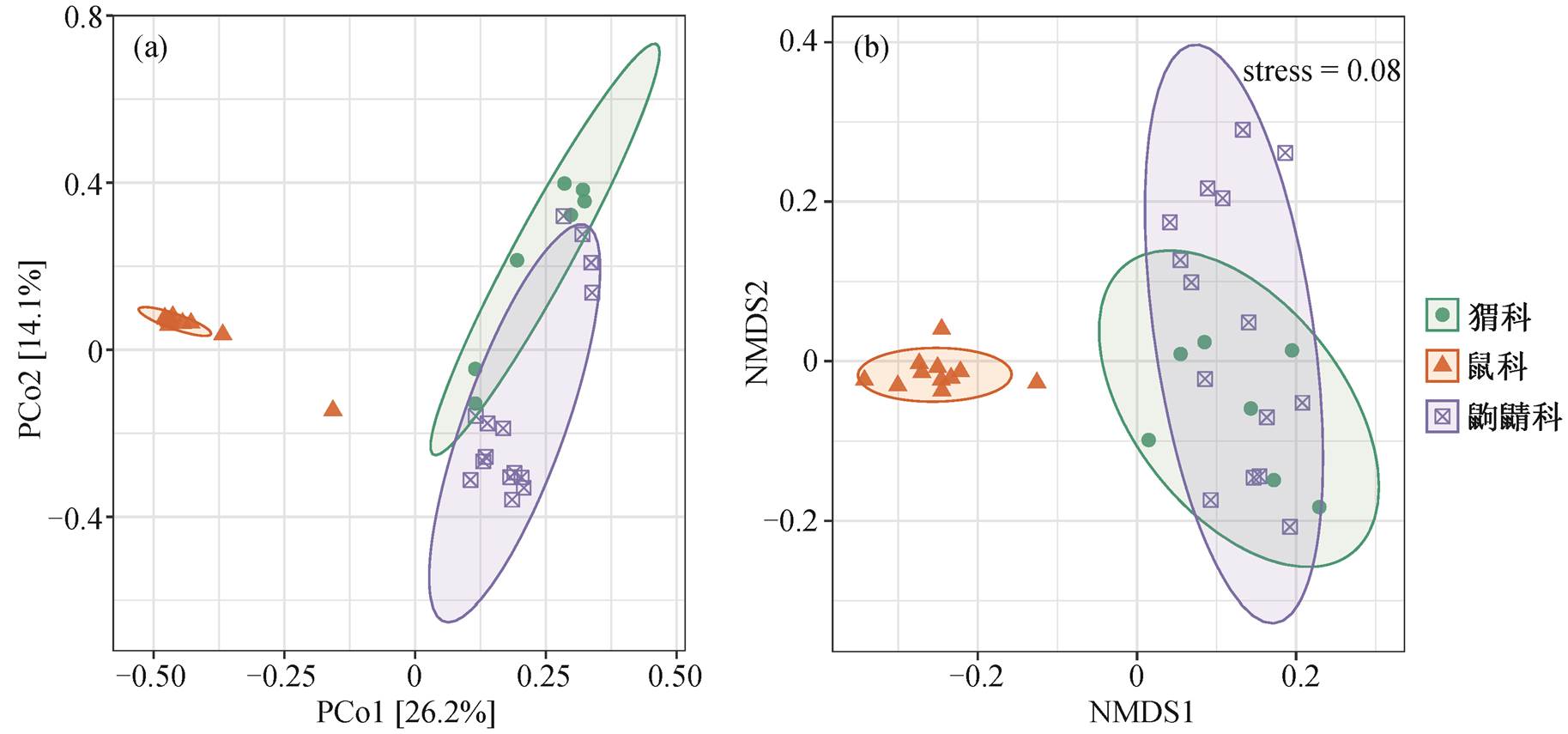
(a)基于Bray-curits距离的主坐标分析; (b)基于加权距离的非度量标定法
图3 基于OTU的物种组成结构分析
Fig. 3 Non-metric multidimensional scaling analysis plots based on OTUs
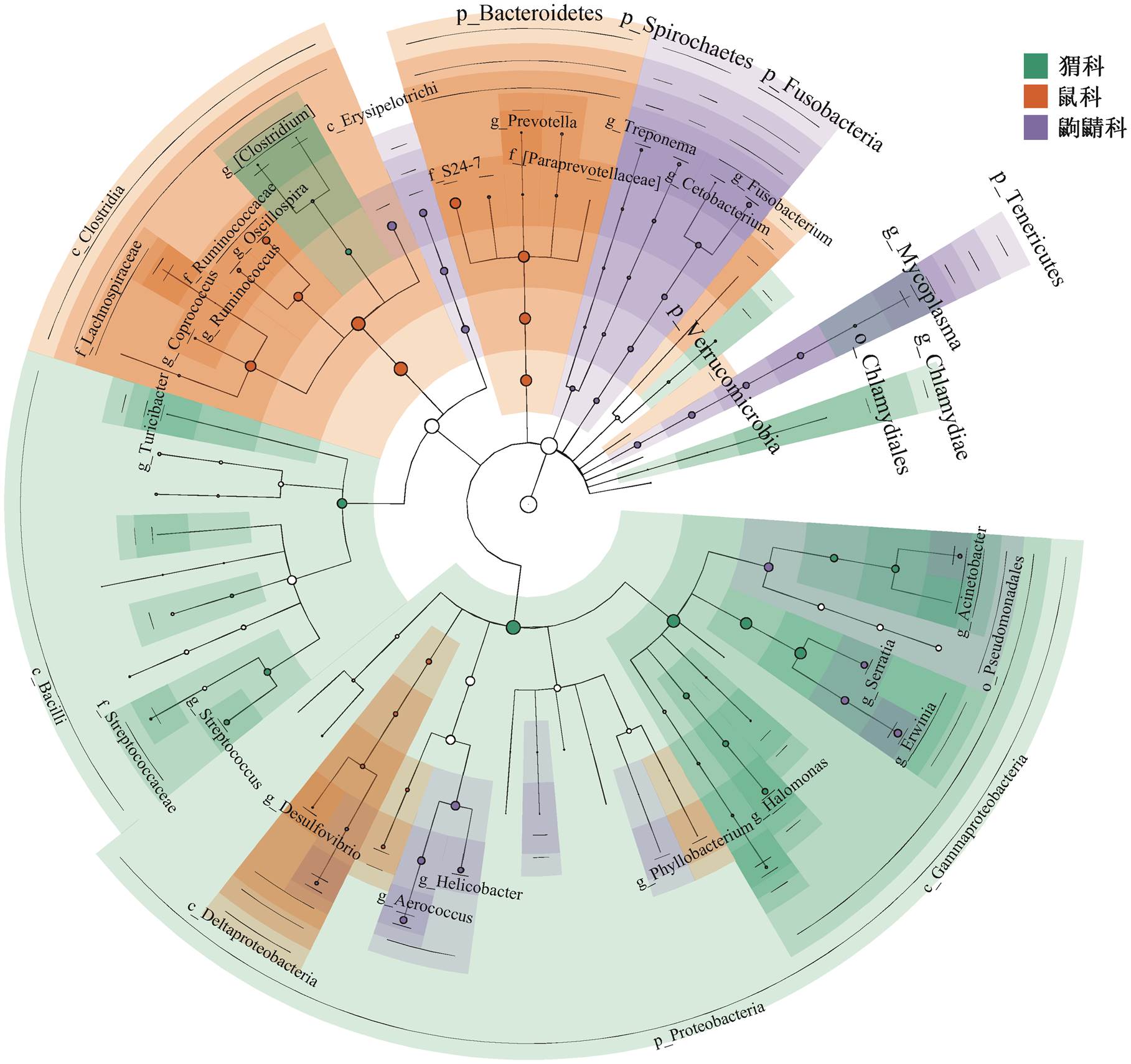
图4 通过线性判别分析效应大小确定的3个宿主科组中的差异菌
Fig. 4 Differences in bacterial taxa among three family host determined by linear discriminative analysis of effect size (LEfSe)
总而言之, 上述分析表明这 3 个科的野生小型哺乳类动物肠道菌群有较高的物种特异性, 宿主饮食对塑造肠道微生物有重要的影响。在饮食相似、栖息环境相同的情况下, 同一目的物种肠道微生物也有一定的差异, 说明宿主遗传对肠道微生物的影响不可避免。
对 3 个科之间进行特有和共有 OTU 分析, 结果如图 5 所示, 鼠科、猬科和鼩鼱科共有 10 个 OTU, 占全部 OTU 序列的 10.4%。鼠科含有最多的特有核心 OUT(为 581), 相对丰度为 25.6%, 而猬科和鼩鼱科含有较少的特有的核心 OTU, 进一步证明鼠科中具有较高的物种丰富度和多样性。这与 α 多样性分析中鼠科多样性显著高于猬科和鼩鼱科一致。猬科与鼩鼱科之间的共有 OTU 多于猬科与鼠科, 说明同一目的物种具有更多的相似菌群, 与 β 多样性中猬科与鼩鼱科的肠道菌群结构更相似的结果一致。3 个科共有 OUT, 主要注释分类为 Serratia(2.53%), Enterococcus (0.65%),Lactococcus (0.53%), Lactobacillus (0.49%)以及 Clostridium (0.28%)共 5个属。Lactobacillus, Lactococcus 和 Enterococcus 是潜在的肠道益生菌[54–55], 影响宿主的健康。Sted-man 等[56]发现 Lactobacillus 作为肠道共生菌, 也显示出作为野生生物益生菌的有益特性, 可以减少野生动植物物种传染病。共有 OTU 的功能作用表明, 益生菌可能广泛存在于野生哺乳类动物肠道中, 并对宿主的健康发挥着不可估量的调控作用。
通过 PICRUSt2, 总共预测到 40 个第二水平的代谢通路, 其中膜运输、碳水化合物代谢、氨基酸代谢、复制与修复以及能量代谢等是这 3 个科中肠道微生物主要预测到的代谢通路(附录 4)。其中, 碳水化合物和氨基酸代谢在鼠科中的相对丰度与猬科和鼩鼱科具有显著差异, 由图 6 和 7 可见, 代谢淀粉和蔗糖的 1,6-葡萄糖苷寡糖和 β-果糖呋喃糖苷酶(K01182, K01193)在鼠科中的丰度显著高于猬科和鼩鼱科。鼠科中的肠道微生物似乎可以从更广泛的多糖中获取能量, 而异生物素的生物降解和代谢在猬科与鼩鼱科中得到富集。食虫目对异生素的降解和代谢被认为是有毒或外来化合物暴露导致的微生物群功能性反应[57]。同时, 猬科和鼩鼱科中富含与氨基酸代谢相关的酶, 如精氨酸脱羧酶(K01585)和鸟氨酸脱羧酶(K01581)等, 富含动物蛋白饮食的猬科和鼩鼱科, 可以通过氨基酸代谢为宿主提供更多的能量。这些结果表明, 宿主饮食的差异使得肠道微生物通过调控其代谢途径来适应不同的宿主环境。
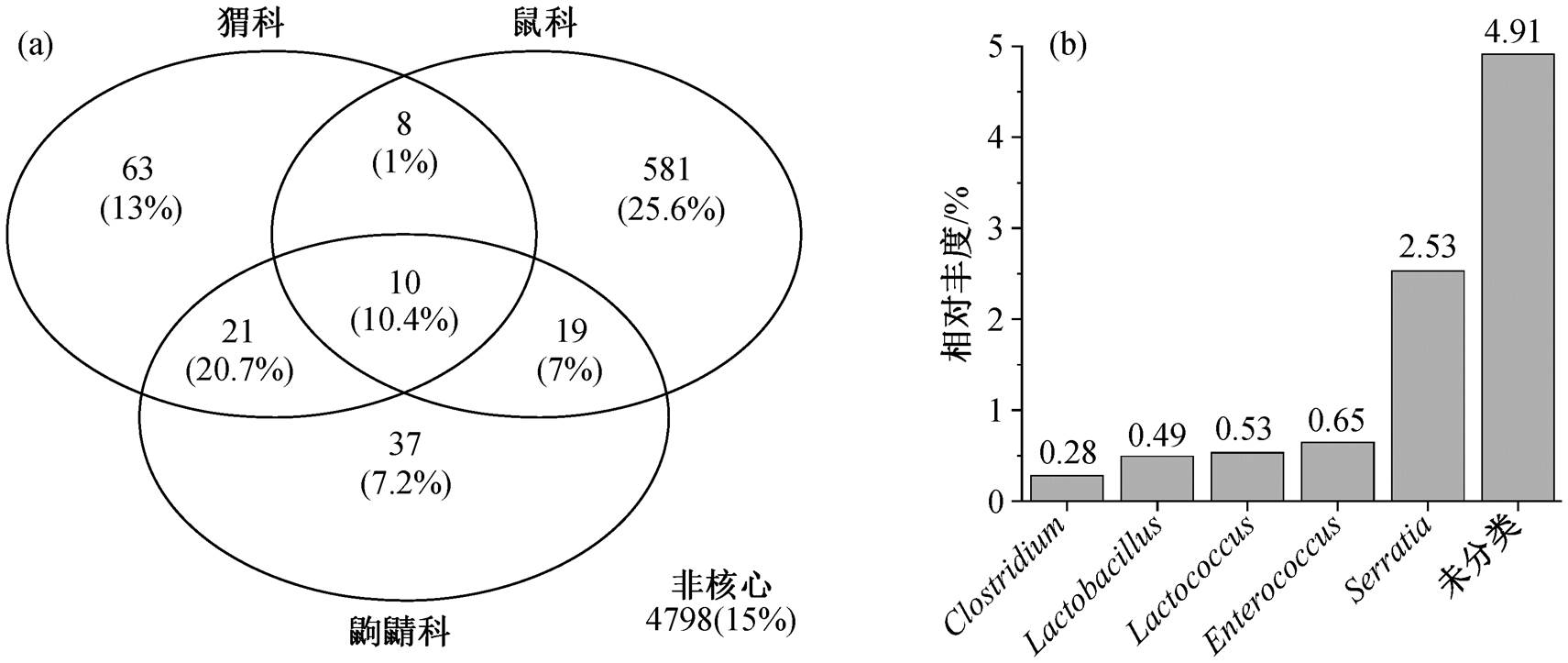
图5 3个科间特有与共有核心微生物数量(a)以及共有OTU在所有样品中的相对丰度(b)
Fig. 5 Number of shared and unique OTUs (a) and core bacterial relative abundance of all samples (b) of three family host groups
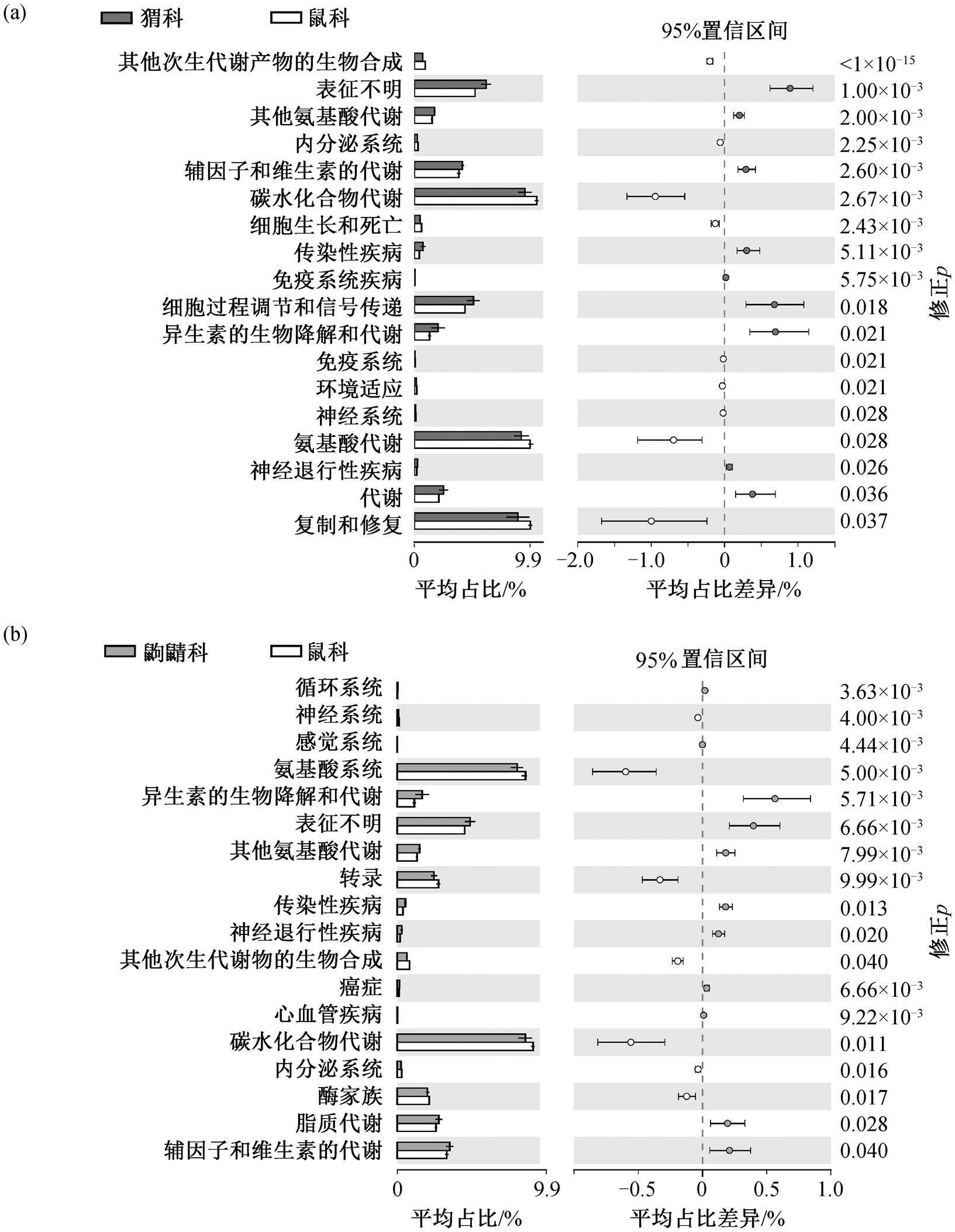
图6 鼠科与猬科(a)、鼠科与鼩鼱科(b)的第二水平代谢通路差异
Fig. 6 Statistical difference of second-level metabolic pathways: muridae host group vs erinaceidae host group (a) and muridae host group vs soricidae host group (b)
本研究基于 16S rRNA 基因扩增子测序技术, 分析鼠科、猬科和鼩鼱科的肠道微生物组成及多样性, 得出以下结论。
1)鼠科、猬科和鼩鼱科的肠道菌群组成由Firmicutes, Bacteroidetes 和 Proteobacteria 主导。鼠科以 Firmicutes 和 Bacteroidetes 为主, 猬科和鼩鼱科以 Proteobacteria 为主。鼠科的肠道菌群多样性高于猬科和鼩鼱科, 且 3 个野生物种科的肠道菌群结构具有显著差异。
2)鼠科肠道中 Prevotella, Oscillospira 及 Ru-minococcus 等纤维素降解菌属丰度显著高于猬科和鼩鼱科, 鼠科中碳水化合物代谢途径较为丰富。猬科和鼩鼱科中蛋白水解细菌(如 Clostridium 和Fuso-bacterium)丰度较高, 表明特定的内源性因素(包括其营养水平)对肠道微生物的影响较大, 肠道微生物会根据宿主的饮食习惯改变其组成, 适应肠道环境的变化。
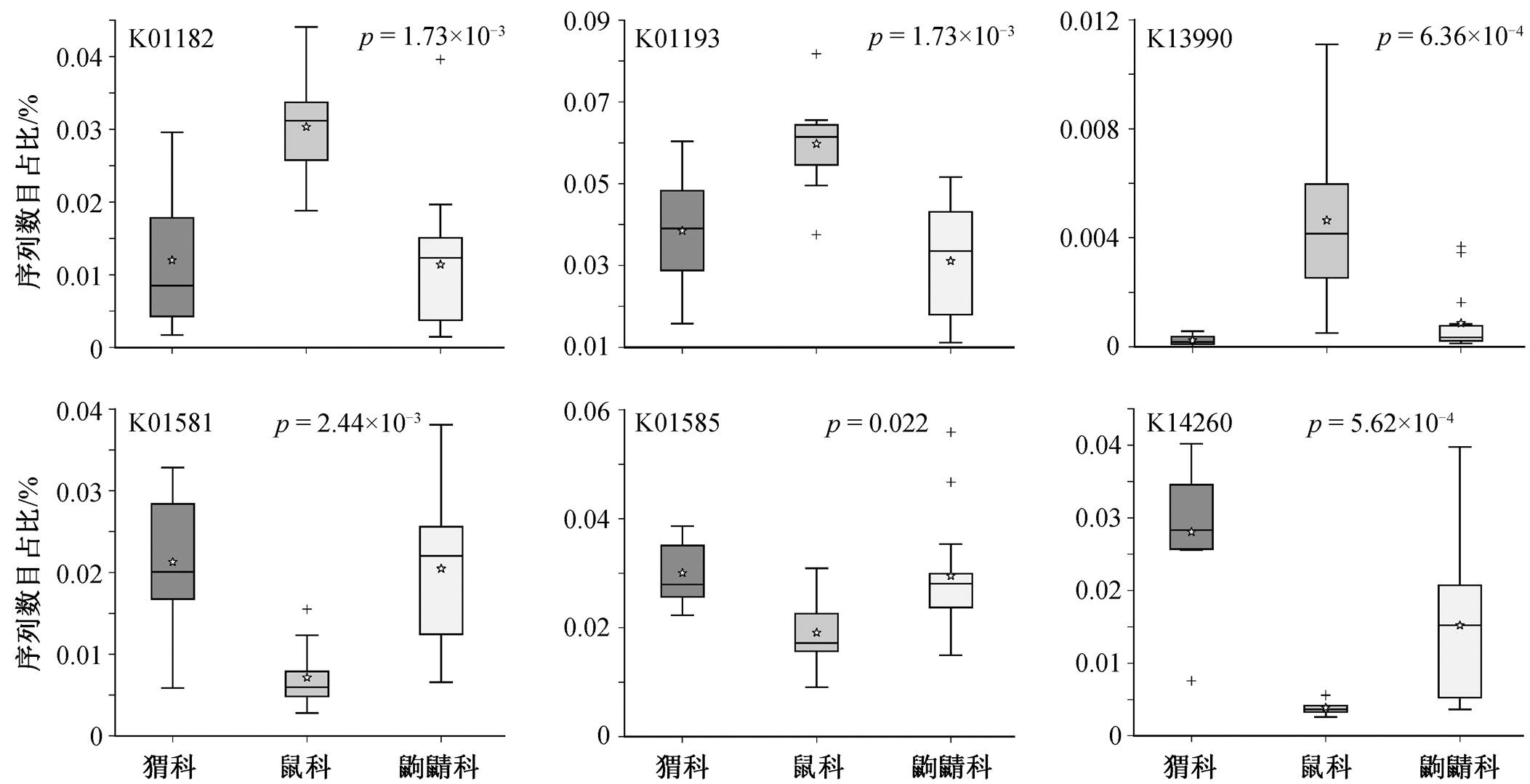
图7 鼠科、猬科和鼩鼱科三组之间的差异KO基因
Fig. 7 Statistical difference of KO genes among muridae, erinaceidae and soricidae host groups
3)具有抗病能力的益生菌(如 Lactobacillus 和Lactococcus 等)共存于这 3 类野生小型哺乳类动物中, 可能降低野生小型哺乳类动物感染疾病的风险。
参考文献
[1] Ley R E, Peterson D A, Gordon J I. Ecological and evolutionary forces shaping microbial diversity in the human intestine. Cell, 2006, 124(4): 837–848
[2] Hooper L V, Midtvedt T, Gordon J I. How host-micro-bial interactions shape the nutrient environment of the mammalian intestine. Annual Review of Nutrition, 2002, 22(1): 283–307
[3] Round J L, Mazmanian S K. The gut microbiota shapes intestinal immune responses during health and disease. Nature Reviews Immunology, 2009, 9(5): 313–323
[4] Fraune S, Bosch T C. Why bacteria matter in animal development and evolution. Bioessays, 2010, 32(7): 571–580
[5] Heijtz R D, Wang S, Anuar F, et al. Normal gut micro-biota modulates brain development and behavior. Proceedings of the National Academy of Sciences, 2011, 108(7): 3047–3052
[6] 张亮, 徐艳春, 李晓平, 等. 肠道微生态系统及其与宿主的协同进化. 现代生物医学进展, 2010, 10 (1): 168–172
[7] Rosshart S P, Herz J, Vassallo B G, et al. Laboratory mice born to wild mice have natural microbiota and model human immune responses. Science, 2019, 365: eaaw4361
[8] Youngblut N D, Reischer G H, Walters W, et al. Host diet and evolutionary history explain different aspects of gut microbiome diversity among vertebrate clades. Nature Communications, 2019, 10(1): 1–15
[9] Rosshart S P, Vassallo B G, Angeletti D, et al. Wild mouse gut microbiota promotes host fitness and im-proves disease resistance. Cell, 2017, 171(5): 1015–1028
[10] West A G, Waite D W, Deines P, et al. The micro-biome in threatened species conservation. Biological Conservation, 2019, 229: 85–98
[11] Guo W, Ren K, Ning R, et al. Fecal microbiota tran-splantation provides new insight into wildlife conser-vation. Global Ecology and Conservation, 2020, 24: e01234
[12] 王琳琳. 中国劳亚食虫类哺乳动物六物种转录组的初步分析[D]. 广州: 南方医科大学, 2020
[13] Suzuki T A, Phifer-Rixey M, Mack K L, et al. Host genetic determinants of the gut microbiota of wild mice. Molecular Ecology, 2019, 28(13): 3197–3207
[14] Suzuki T A, Martins F M, Nachman M W. Altitudinal variation of the gut microbiota in wild house mice. Mol Ecol, 2019, 28(9): 2378–2390
[15] Maurice C F, Knowles S C, Ladau J, et al. Marked seasonal variation in the wild mouse gut microbiota. The ISME Journal, 2015, 9(11): 2423–2434
[16] 周洁, 周晓君, 轩断断, 等. 海南岛中部山区鼠类肠道菌群多样性研究. 中国媒介生物学及控制杂志, 2020, 31(5): 596–601
[17] Romanenko S, Perelman P, Trifonov V, et al. Chromo-somal evolution in Rodentia. Heredity, 2012, 108(1): 4–16
[18] 母华强, 张泽钧, 张明春, 等. 四川宝兴中国鼩猬的一些生物学资料. 四川动物, 2011, 30(1): 94–96
[19] 周婧晶, 丘银彬, 万韬, 等. 鼩鼱科三物种转录组初步分析. 兽类学报, 2021, 41(2): 173–181
[20] 聂永刚, 胡锦矗, 陈锋华. 微尾鼩的食性与防治初探. 皖西学院学报, 2006(2):73–75.
[21] Douady C J, Douzery E J P. Molecular estimation of eulipotyphlan divergence times and the evolution of “Insectivora”. Molecular Phylogenetics Evolution, 2003, 28(2): 285–296
[22] 吴德林, 罗成昌. 人类活动对云南哀牢山小型兽类群落结构的影响. 动物学研究, 1993, 14(1): 35–41
[23] Wiener J G, Smith M H. Relative efficiencies of four small mammal traps. Journal of Mammalogy, 1972, 53(4): 868–873
[24] Callahan B J, McMurdie P J, Rosen M J, et al. DADA2: high-resolution sample inference from Illu-mina amplicon data. Nature Methods, 2016, 13(7): 581–583
[25] Frøslev T G, Kjøller R, Bruun H H, et al. Algorithm for post-clustering curation of DNA amplicon data yields reliable biodiversity estimates. Nature Commu-nications, 2017, 8(1): 1–11
[26] Katoh K, Standley D M. MAFFT multiple sequence alignment software version 7: improvements in performance and usability. Molecular Biology and Evolution, 2013, 30(4): 772–780
[27] Price M N, Dehal P S, Arkin A P. FastTree: computing large minimum evolution trees with profiles instead of a distance matrix. Molecular Biology and Evolu-tion, 2009, 26(7): 1641–1650
[28] McDonald D, Price M N, Goodrich J, et al. An im-proved Greengenes taxonomy with explicit ranks for ecological and evolutionary analyses of bacteria and archaea. The ISME Journal, 2012, 6(3): 610–618
[29] Leung T, Poulin R. Parasitism, commensalism, and mutualism: exploring the many shades of symbioses. Vie et Milieu, 2008, 58(2): 107–115
[30] McMurdie P J, Holmes S. phyloseq: an R package for reproducible interactive analysis and graphics of mic-robiome census data. PLoS One, 2013, 8(4): e61217
[31] Cuzick J A. Wilcoxon-type test for trend. Statistics in Medicine, 1985, 4(1): 87–90
[32] Oksanen J. Constrained ordination: tutorial with R and vegan. R-packace Vegan, 2012, 1(10): 1–10
[33] Anderson M J. Permutational multivariate analysis of variance. Department of Statistics, 2005, 26: 32–46
[34] Andersen K S, Kirkegaard R H, Karst S M, et al. ampvis2: an R package to analyse and visualise 16S rRNA amplicon data. BioRxiv, 2018: 299537
[35] Paulson J N, Stine O C, Bravo H C, et al. Differential abundance analysis for microbial marker-gene sur-veys. Nature Methods, 2013, 10(12): 1200–1202
[36] Douglas G M, Maffei V J, Zaneveld J R, et al. PICRUSt2 for prediction of metagenome functions. Nature Biotechnology, 2020, 38(6): 685–688
[37] Parks D H, Tyson G W, Hugenholtz P, et al. STAMP: statistical analysis of taxonomic and functional pro-files. Bioinformatics, 2014, 30(21): 3123–3124
[38] Chevalier C, Stojanović O, Colin D J, et al. Gut microbiota orchestrates energy homeostasis during cold. Cell, 2015, 163(6): 1360–1374
[39] Moon C D, Young W, Maclean P H, et al. Metage-nomic insights into the roles of Proteobacteria in the gastrointestinal microbiomes of healthy dogs and cats. Microbiologyopen, 2018, 7(5): e00677
[40] Flint H J, Bayer E A, Rincon M T, et al. Poly-saccharide utilization by gut bacteria: potential for new insights from genomic analysis. Nature Reviews Microbiology, 2008, 6(2): 121–131
[41] Rxowland I, Gibson G, Heinken A, et al. Gut mic-robiota functions: metabolism of nutrients and other food components. European Journal of Nutrition, 2018, 57(1): 1–24
[42] Klimenko N S, Tyakht A V, Popenko A S, et al. Microbiome responses to an uncontrolled short-term diet intervention in the frame of the citizen science project. Nutrients, 2018, 10(5): 576
[43] Nelson T M, Rogers T L, Brown M V. The gut bacterial community of mammals from marine and terrestrial habitats. PLoS One, 2013, 8(12): e83655
[44] Ley R E, Hamady M, Lozupone C, et al. Evolution of mammals and their gut microbes. Science, 2008, 320: 1647–1651
[45] David L A, Maurice C F, Carmody R N, et al. Diet rapidly and reproducibly alters the human gut micro-biome. Nature, 2014, 505: 559–563
[46] Knowles S C L, Eccles R M, Baltrūnaitė L. Species identity dominates over environment in shaping the microbiota of small mammals. Ecology Letters, 2019, 22(5): 826–837
[47] Reichardt N, Duncan S H, Young P, et al. Phylo-genetic distribution of three pathways for propionate production within the human gut microbiota. The ISME Journal, 2014, 8(6): 1323–1335
[48] Shah H N, Collins D M. Prevotella, a new genus to include Bacteroides melaninogenicus and related species formerly classified in the genus Bacteroides. International Journal of Systematic and Evolutionary Microbiology, 1990, 40(2): 205–208
[49] Ou J, Carbonero F, Zoetendal E G, et al. Diet, microbiota, and microbial metabolites in colon cancer risk in rural Africans and African Americans. The American Journal of Clinical Nutrition, 2013, 98(1): 111–120
[50] Tyakht A V, Kostryukova E S, Popenko A S, et al. Human gut microbiota community structures in urban and rural populations in Russia. Nature Communi-cations, 2013, 4(1): 1–9
[51] Scott K P, Gratz S W, Sheridan P O, et al. The influ-ence of diet on the gut microbiota. Pharmacological Research, 2013, 69(1): 52–60
[52] Lau S K, Teng J L, Chiu T H, et al. Differential microbial communities of omnivorous and herbivo-rous cattle in southern China. Computational Struc-tural Biotechnology Journal, 2018, 16: 54–60
[53] O’Connor A, Quizon P M, Albright J E, et al. Res-ponsiveness of cardiometabolic-related microbiota to diet is influenced by host genetics. Mammalian Ge-nome, 2014, 25(11): 583–599
[54] Fernandez B, Hammami R, Savard P, et al. Pedio-coccus acidilactici UL 5 and Lactococcus lactis ATCC 11454 are able to survive and express their bacter-iocin genes under simulated gastrointestinal condi-tions. Journal of Applied Microbiology, 2014, 116(3): 677–688
[55] Vimont A, Fernandez B, Hammami R, et al. Bacter-iocin-producing Enterococcus faecium LCW 44: a high potential probiotic candidate from raw camel milk. Frontiers in Microbiology, 2017, 8: 865
[56] Stedman A, van Vliet A, Chambers M, et al. Gut com-mensal bacteria show beneficial properties as wildlife probiotics. Annals of the New York Academy of Sciences, 2020, 1467(1): 112–132
[57] Rampelli S, Schnorr S L, Consolandi C, et al. Meta-genome sequencing of the Hadza hunter-gatherer gut microbiota. Current Biology, 2015, 25(13): 1682–1693
Diversity Analysis of Intestine Microbiota of Yunnan Small Wildlife Mammals Based on 16S rRNA Gene Amplicon Sequencing Technology
Abstract This study takes two wild mammals living in the same environment, but with different evolutionary relationships and different dietary habits (Apodemus, Erinaceidae and Soricidae) as the research objects, using the 16S rRNA gene amplicon sequencing technology to analyze and compare the gut microbiota of these mammals. 5378 operational taxonomic units (OTU) are identified, mainly affiliated with Firmicutes (40.55%), Proteobacteria (34.60%), and Bacteroidetes (13.67%). Firmicutes and Bacteroidetes are the dominant phyla of the Apodemus. Proteobacteria is the dominant phylum of Erinaceidae and Soricidae. The results of diversity analyses show that the gut microbiota diversity and community composition are significantly different among Apodemus, Erinaceidae and Soricidae. LEfSe analysis shows that there are more bacteria related to complex carbohydrate fermentation in the Apodemus, while the Erinaceidae and Soricidae contains higher amino acid fermentation bacteria. Probiotics, such as Lactobacillus and Lactococcus, coexist in these three wild small mammals to regulate host health. This study reveals that phylogeny and feeding habits of the host have a profound effect on the shaping gut microbes. The gut microbiota may also play a probiotic effect on the host in many aspects.
Key words wild small mammals; gut microbiota; 16S rRNA gene amplicon sequencing; host diet
doi: 10.13209/j.0479-8023.2022.002
国家自然科学基金(51709005)资助
收稿日期: 2021-03-31;
修回日期: 2021-05-28