图1 异质信息网络中元路径示例
Fig.1 Examples of meta-paths in heterogeneous information network
摘要 提出一种融合元路径信息的图神经网络模型, 用于预测药物‒靶标相互作用(GMDTI)。首先根据 8 个数据集中的药物、靶标、疾病和副作用数据以及它们之间的 8 种作用关系, 构建药物‒靶标异质信息网络(HIN); 然后定义两条元路径来捕获药物‒靶标 HIN 中的不同子结构信息和不同节点间隐藏的语义信息, 并应用图神经网络的方法聚合节点的一阶邻居信息和元路径中节点间的语义信息; 最后利用端到端的学习方法完成 DTIs 预测。该方法同时考虑药物‒靶标 HIN 的结构特性和元路径语义信息, 有助于学习到更多潜在的药物‒靶标作用关系。实验结果表明, GMDTI 的预测准确率高于所有基线模型, AUC 达到 98.6%, AUPR 达到94.5%。同时通过调整数据的稀疏度和降噪实验, 证明 GMDTI 具备优于所有基线模型的鲁棒性。
关键词 药物‒靶标相互作用预测; 图神经网络; 异质信息网络; 元路径; 特征表示
药物‒靶标相互作用(drug-target interactions, DTIs)预测是药物研发的关键步骤。DTIs 预测指通过药物和靶标的结构特征以及已知的药物与靶标之间的关系、药物与药物之间的关系等信息, 挖掘目前尚未发现的潜在的药物-靶标相互作用关系。通过识别尚未发现的 DTIs, 可以探索已知药物的新用途。在新药物的研发过程中准确地预测 DTIs, 可以帮助研究人员快速地筛选出有效的候选药物, 降低研发成本, 减少研制的盲目性, 因此预测 DTIs 是新药物研发工作中极为重要的基础任务[1-2]。
传统的 DTIs 预测方法主要有两种: 基于配体的方法[3]和分子对接模拟法[4]。基于配体的方法利用相似的分子通常会与相似的靶标相结合的思想, 通过比较新的配体与已知的靶标配体来预测 DTIs。目前, 大多数基于配体的方法都是针对一个靶标建立的, 使其只能针对一个靶标的分子活性做预测, 推广使用受到限制。分子对接模拟法利用靶标的三维结构进行模拟[4-6], 当靶标的三维结构不可用时, 这类方法失效。此外,对接模拟通常需要很长的时间, 效率较低。
近年来, 随着人工智能技术在生物医疗领域的深度应用, 越来越多的研究人员致力于使用机器学习的计算方法来预测 DTIs, 可以很好地克服传统DTIs 预测方法只能针对单个靶标做预测以及预测精确度不高、效率低的问题。
基于机器学习的 DTIs 预测方法可分为基于矩阵相似度计算的和基于异质信息网络(heterogene-ous information network, HIN)的两大类。
基于矩阵相似度计算的方法是通过不同的矩阵相似性度量方法来计算药物与靶标之间的相似性, 从而进行 DTIs 预测, 主要包含二分图局部方法和矩阵分解方法。Bleakley 等[7]提出二分图局部模型, 首次利用有监督机器学习方法进行 DTIs 预测, 将药物-靶标相互作用预测问题转换成二分类问题, 将药物的化学结构和靶标的序列结构作为输入特征, 分别训练药物和靶标的局部模型, 因此 SVM分类器可以针对药物和靶标生成两个独立的预测结果, 基于这两个独立预测结果的平均值, 计算药物-靶标的最终预测结果。基于矩阵分解的相似度度量方法则将 DTIs 预测任务视为寻找缺失相互作用矩阵的补全问题, 例如 Zheng 等[8]提出 MSCMF 模型, 通过加权平均方案来整合多个数据源的信息, 从而获得对应的药物和靶标相似度矩阵, 然后使用这些相似度矩阵来正则化给定的 DTIs 网络的矩阵分解操作。
基于矩阵相似度计算的预测方法没有考虑网络的拓扑结构, 也没有区分网络中药物与靶标之间关系的异质性, 所以会损失网络中节点之间的交互语义信息, 导致无法进行更准确的 DTIs 预测。因此, 基于 HIN 的方法被用于 DTIs 预测。为了集成异构数据源中的各种信息, Luo 等[9]提出 DTINet 预测方法, 从所构建的药物-靶标 HIN 中自动学习药物和靶标的低维特征向量(该特征向量可以准确地解释网络中节点的拓扑结构的特性), 然后运用归纳矩阵, 在学到的特征基础上完成 DTIs 预测。
由于 DTINet 将特征学习与任务分离, 所以学习到的特征表示不一定是 DTIs 预测任务中的最优表示。为了解决特征学习与任务分离的问题, Wan等[10]创建一个新的框架 NeoDTI, 使用图神经网络邻居信息聚合[11]的方法, 通过聚合节点的一阶邻居信息来提取药物和靶标的复杂隐藏特征, 并从中学习节点含有网络拓扑结构的特征表示, 取得出色的预测结果。此外, 为了聚合 HIN 中节点的高阶信息, Liu 等[12]等提出 GADTI 模型, 通过将 GCN[13]与随机游走相结合, 使信息聚合的范围从一阶扩展到多阶, 相当于增加了卷积的感受野, 实现更远距离的信息传递。
基于 HIN 的 DTIs 预测方法优势在于可以整合不同类型节点之间的交互信息和节点间隐藏的语义信息, 但带来一个新的问题: 如何有效地表示异质信息网络节点间隐含的语义信息?
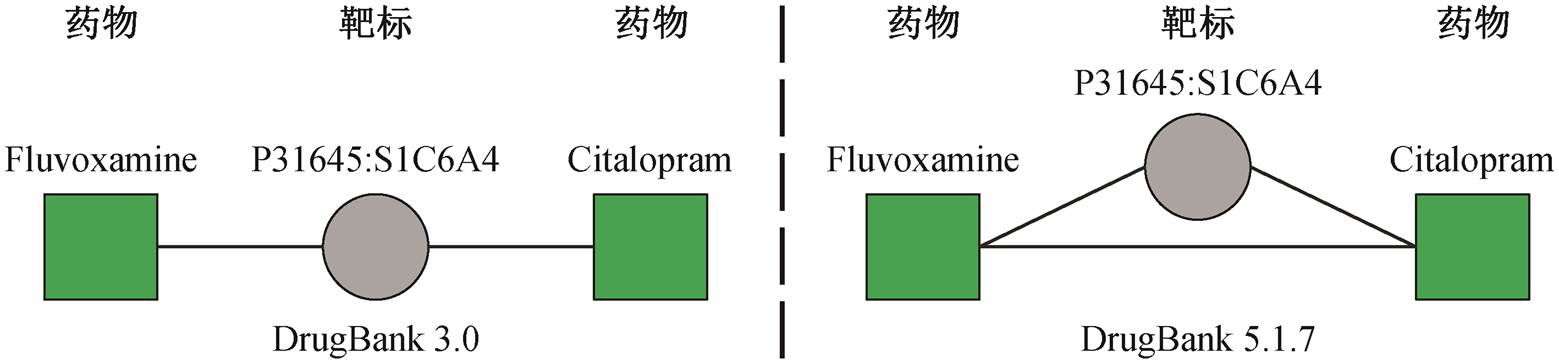
元路径[14]可以指定对象的连接序列, 获取网络的子结构, 并捕获源节点与目标节点间的语义, 广泛地运用于基于 HIN 的数据挖掘问题中[15]。在药物-靶标HIN 中, 同样可以利用元路径来抽取网络的子结构, 并捕获源节点和目标节点间的语义信息。如图 1 所示, 在 DrugBank 3.0 版数据库[16]中, 氟伏沙明与西酞普兰没有任何联系, 但二者可以通过添加药物-靶标-药物这条元路径发生关联。在最新的 Drug Bank 5.1.7 版数据库中, 的确更新了氟伏沙明与西酞普兰之间的联系, 表示联合使用氟伏沙明和西酞普兰可以提高血清浓度, 说明可以通过元路径捕获有利于提高预测效果的语义信息。
为了解决既有方法没有利用 HIN 的子结构信息以及节点间隐藏的语义信息这一问题, 本文提出一种融合元路径信息的图神经网络模型, 用于预测药物-靶标相互作用的方法(graph neural network with meta-path information for drug-target interaction pre-diction model, GMDTI)。在 NeoDTI 模型的基础上, 加入两条不同的元路径来捕获药物-靶标 HIN 中不同类型的网络子结构和源节点与目标节点间的语义信息, 同时考虑药物、靶标、疾病与副作用节点的一阶邻居信息, 然后运用图神经网络提取 HIN中每个节点的特征, 最后根据所提取节点的特征进行DTIs 预测。

图1 异质信息网络中元路径示例
Fig.1 Examples of meta-paths in heterogeneous information network
定义 1 药物-靶标 HIN。给定一个图 G = (V, E), V 代表节点集, E 代表边集。节点集合中每个节点 v 属于对象集合 O 中的一种对象类型, 其中O={药物, 靶标, 疾病, 副作用}; 边集合中的每条边属于关系类型集合 R 中的一种关系类型, 其中R={药物-药物-相互作用, 药物-药物-结构相似度, 药物-靶标-相互作用, 药物-疾病-相互联系, 药物-副作用-相互联系, 靶标-靶标-相互作用, 靶标-靶标-结构相似性, 靶标-疾病-相互联系}。
定义 2 元路径。一条元路径 Φ 被定义为
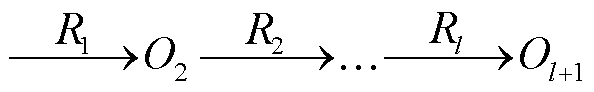 。此路径描述对象 O1到 Ol+1 之间的复合关系 R=R1°R2°…°Rl, 其中°表示关系上的合成运算符。此外, 定义 Φo 为从节点类型为 o(o∈O)出发的元路径集合。
。此路径描述对象 O1到 Ol+1 之间的复合关系 R=R1°R2°…°Rl, 其中°表示关系上的合成运算符。此外, 定义 Φo 为从节点类型为 o(o∈O)出发的元路径集合。
定义 3 基于元路径的邻居。给定一个节点 v和一条元路径 Φ, 节点 v 基于元路径 Φ 的邻居集合为NvΦ。
如图 2 所示, 本文提出的 GMDTI 模型具有以下特点: 1)使用 8 个独立的与药物和靶标相关的数据集来构建药物-靶标 HIN, 该网络由 4 种类型的节点和 8 种类型的边构成, 不同类型的节点由不同类型的边连接, 相同类型的节点可以由多种类型的边连接; 2)针对药物-靶标 HIN 中的所有节点, 使用低维向量进行随机初始化表示, 然后通过其一阶邻居信息来更新每个节点的特征表示; 3)基于已构建的药物-靶标 HIN, 设计两条包含不同语义信息的元路径, 根据元路径找到药物和靶标节点基于元路径的邻居; 4)通过聚合药物和靶标节点基于元路径的邻居信息, 更新药物和靶标节点的特征表示; 5)通过以上步骤学到的节点特征表示重构初始的药物-靶标 HIN, 旨在最小化初始网络与重构网络之间的差异, 并且利用重构的药物-靶标网络进行 DTIs 的预测。
通过聚合节点的一阶邻居信息, 可以让模型学习到 HIN 的整体结构信息。GMDTI 使用图神经网络, 整合来自每个节点的邻居信息。给定一个药物-靶标 HIN, 随机初始化节点向量表示函数 g0: V→Rd 将每个节点 v (v∈V)映射到 d 维的向量表示 g0(v), 边权重映射函数f: E→R 将每条边 e (e∈E)映射到其边权重 f (e)上, 每个节点的邻居信息聚合运算公式为
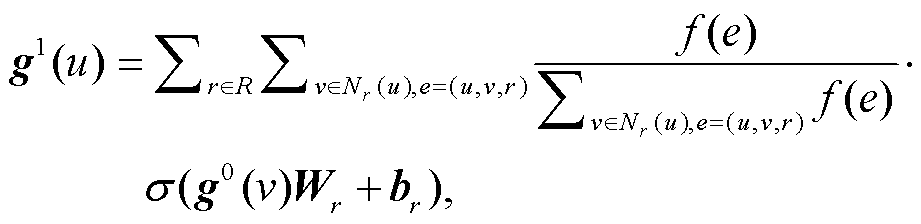 (1)
(1)其中, e=(u, v, r)表示节点 u∈V 和节点 v∈V 通过关系 r∈R 相连接的边, Nr(u) = {v, u ≠ v, u∈V, v∈V, (u, v, r)∈E}表示节点 u 通过关系 r∈R 与节点 v 相连接的节点集合, σ(·)表示非线性激活函数, Wr∈Rd×d是权重矩阵, br∈Rd是偏置项, 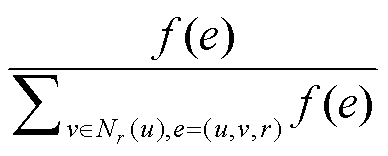 是归一化函数。
是归一化函数。
我们选择药物-靶标-药物和靶标-药物-靶标两条元路径, 药物-靶标-药物路径指不同药物对同一靶标的关联, 靶标-药物-靶标路径指不同靶标对同一药物的关联。通过这两条元路径, 可以获取路径中包含的语义信息, 并且让模型学习到药物-靶标HIN 不同的子结构信息, 进行更精确的 DTIs 预测。
通过元路径 Փ 找到的连接边为 eՓ∈E, 通过边权重映射函数 f: E→R, 将这些边映射到其边权重f (eՓ)上, 节点基于元路径的邻居信息聚合操作运算公式为
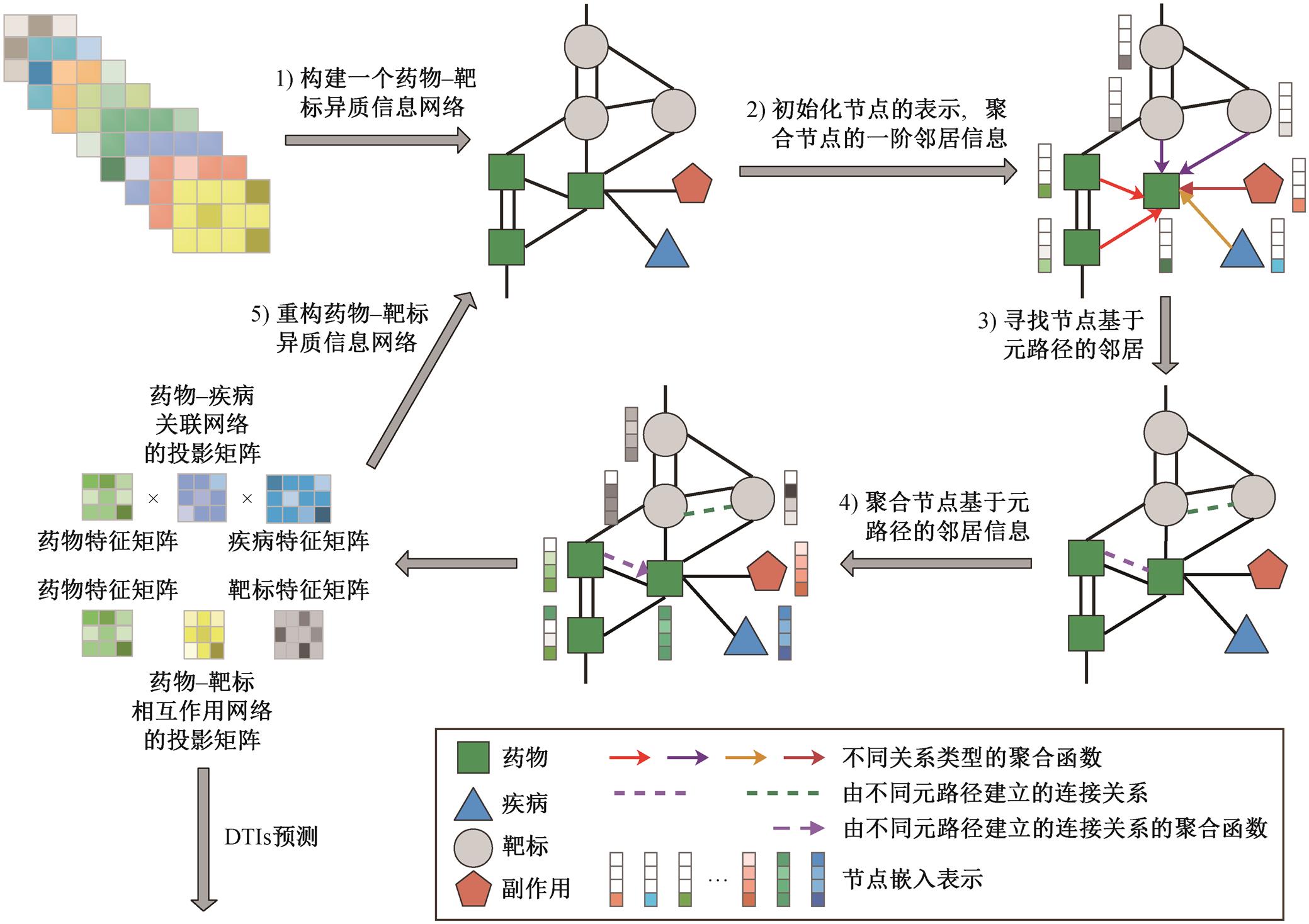
图2 GMDTI模型框架
Fig.2 Overall architecture of GMDTI
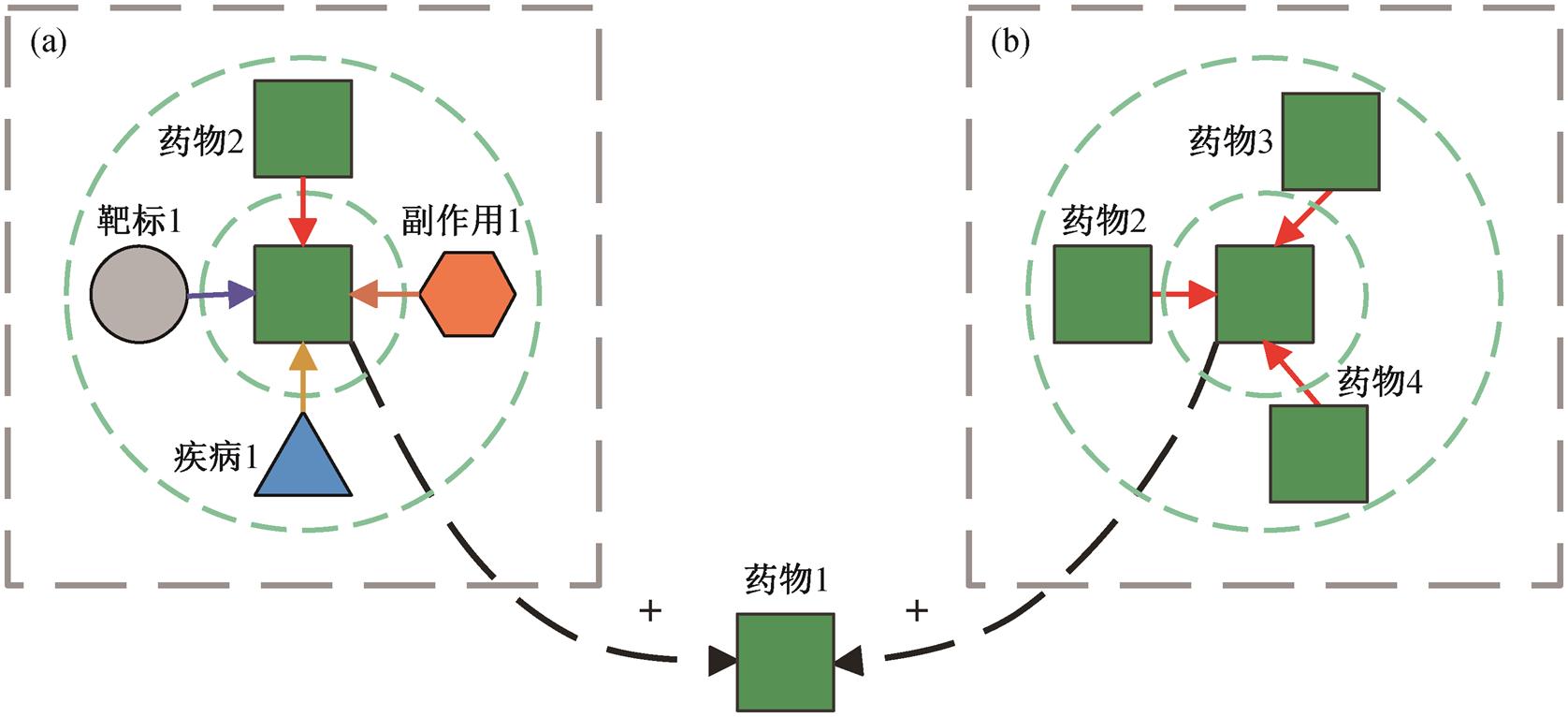
(a)药物 1 节点一阶邻居信息聚合操作; (b)药物 1 节点基于药物‒靶标‒药物元路径的邻居信息聚合操作。不同颜色的箭头表示不同类型边的聚合操作
图3 节点的一阶邻居信息和基于元路径的邻居信息聚合操作流程
Fig.3 Explanation of neighborhood information aggregation and neighborhood information based on drug-target-drug meta-paths aggregation
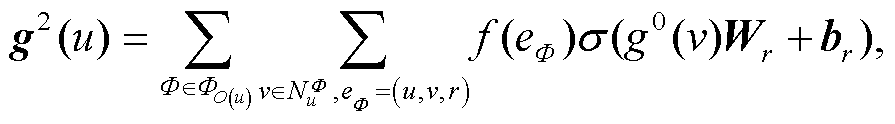 (2)
(2)其中, eՓ=(u, v, r)表示节点 u 和 v 通过元路径 Փ 相连, 且该连接边类型为 r; NuΦ 表示节点 u 基于元路径 Փ的邻居节点集合; ՓO(u)表示从节点类型为 O(u)出发的元路径集合; Wr∈Rd×d是权重矩阵; br∈Rd是偏置项。图 3 展示药物节点的一阶邻居信息聚合和基于药物-靶标-药物元路径的邻居信息聚合操作流程。
为了充分利用药物-靶标 HIN 中的整体结构信息、局部结构信息以及节点间的语义信息, 对于药物和靶标节点 u (u∈O{药物, 靶标}), 用 3 种向量进行聚合表示, 即节点的初始向量表示 g0(u)、一阶邻居信息的向量表示 g1(u)和基于元路径邻居信息的向量表示 g2(u)。对于非药物和靶标节点 u′ (u′∈O{疾病, 副作用}), 仅由节点的初始向量表示 g0(u′)和一阶邻居信息的向量表示 g1(u′)构成。将节点的几种向量表示相加, 再经过单层神经网络和 L2 正则化来更新所有节点的向量表示。节点 u 和 u′的最终向量表示运算公式如下:
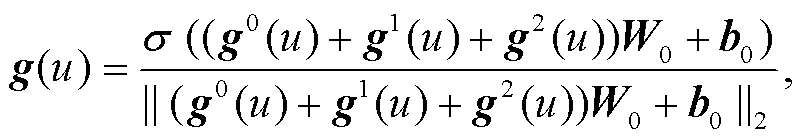 (3)
(3)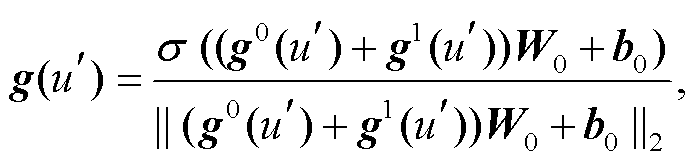 (4)
(4)
其中, W0∈Rd×d是权重矩阵, b0是偏置项。
给定节点 u 的最终嵌入表示 g(u), 训练神经网络, 以便最小化重构矩阵与初始矩阵之间的损失。损失函数定义为
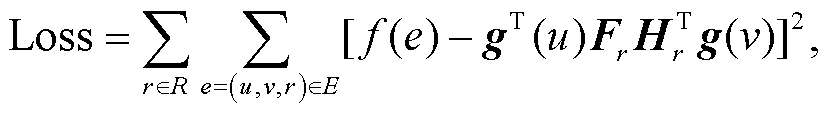 (5)
(5)其中, Fr∈Rd×k和 Hr∈Rd×k是关于类型为 r 边的特定投影矩阵, 这两个投影矩阵的内积应尽可能地还原原始边权重 f (e)。如果边类型 r 是对称的, 例如{药物-药物-相互作用, 靶标-靶标-相互作用, 靶标-靶标-序列相似性}, 则设 Fr=Hr 来增强这种对称性。
考虑到所有操作都是可微的和次可微的, 可以通过执行梯度下降, 以端到端的方式训练参数。训练后, 重构的药物-靶标矩阵可用于预测每个 DTI的得分。重构的药物-靶标相互作用矩阵 M 可以定义为以下形式:
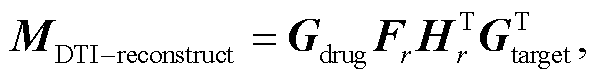 (6)
(6)其中, Gdrug 和 Gtarget 分别是药物和靶标的特征矩阵。
我们采用文献[10]中的数据集。该数据集包含8 个独立的关系矩阵: 药物-药物相互作用矩阵、药物-靶标相互作用矩阵、药物-疾病关联矩阵、药物-药物结构相似度矩阵、药物-副作用关联矩阵、靶标-靶标相互作用矩阵、靶标-靶标序列相似度矩阵以及靶标-疾病关联矩阵。除药物结构相似性和靶标序列相似性矩阵的边是非负实值权重外, 其他所有矩阵均具为二进制边权重(有已知的相互作用或联系边权重为 1, 否则为 0)。另外, 我们通过药物-靶标-药物和靶标-药物-靶标这两条元路径, 提取药物-药物元路径矩阵和靶标-靶标元路径矩阵。实验数据集中包含 708 种药物、1512 种靶标、1923条药物-靶标相互作用边(DTI)、13558 条由药物-靶标-药物元路径建立的连接边以及 4268 条由靶标-药物-靶标元路径建立的连接边。
本文以 AUC (area under the receiver operating characteristic curve)和 AUPR (area under the precision- recall curve)为评价指标, 对实验结果进行度量。AUC 适用于各类正负样本相对平衡的数据。在正负样本高度不平衡的情况下, AUPR 比 AUC 更敏感,更加适用于评价模型在不平衡样本情况下的链接预测能力。
可以把 DTIs 预测任务视为一个二分类问题, 将其中已知的药物-靶标相互作用对作为正样例, 未知的药物-靶标相互作用对作为负样例。为了模拟现实中 DTIs 数据稀疏的情况, 首先采样所有的正样例, 然后对负样例对进行随机采样, 负样列对的数量为正样例对的 10 倍。接下来, 采用 10 折交叉验证来验证模型的性能。在每一折中, 随机选取数据集中 90%的正负样例对作为训练集来训练模型参数, 剩余 10%的数据作为测试集来测试模型的性能。实验中与以下 6 种基线方法进行对比: 1)BLM-NII[17], 基于邻居相互作用谱的局部二分图模型; 2)HNM[18], 多层异质信息网络模型, 能捕获疾病、药物和靶标之间的相互关系和内部联系; 3)MSCMF, 多相似度矩阵分解模型, 用矩阵分解方法将药物和靶标矩阵规范化, 能够集成多种相似矩阵; 4)DTI-Net, 一种网络集成方法, 能集成异构数据源中的各种信息, 学习节点包含 HIN 拓扑结构的低维特征向量; 5)NeoDTI, 采用图神经网络的方法, 能够集成多种信息源数据, 并自动学习节点保留网络拓扑结构的向量表示; 6)GADTI, 采用图神经网络和重启随机游走方法, 聚合节点的多阶邻居信息, 实现更远距离的信息传递。
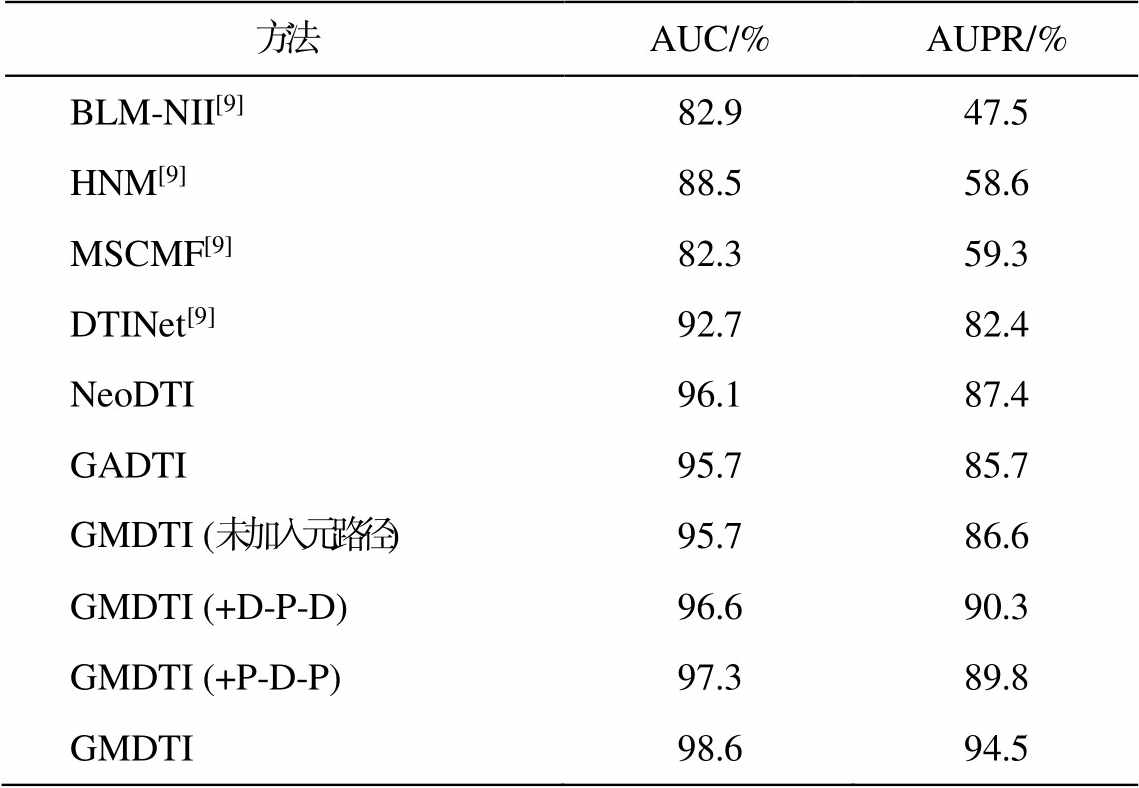
表 1 给出本文方法和基线方法在数据集上的性能表现, 其中每个实验结果均为 10 折交叉验证所得。可以看出, 在没有加入元路径的情况下, GMDTI能够基本上准确地预测 DTIs, 其 AUC 几乎优于所有基线方法, AUPR 与表现最好的基线方法NeoDTI仅相差 0.8%。基于异质结构网络的 GADTI 是通过重启随机游走的方法获取节点的高阶邻居信息, 所以很容易捕获与当前节点相关性弱的高阶节点信息, 进而削弱相关性强的一阶邻居信息的影响, 导致效果没有 NeoDTI 方法好。其他基线方法, 由于没有利用药物-靶标 HIN 的拓扑结构信息和隐藏的语义信息, 仅利用少量与药物和靶标相关的数据, 或仅利用简单的矩阵分解方法, 不能处理矩阵内冗余的信息, 所以预测效果均不佳。
从表 1 还可以看出, 在 GMDTI 中加入药物-靶标-药物元路径或靶标-药物-靶标元路径, AUC 和AUPR 均优于所有基线方法, 并且与没有加入元路径的 GMDTI 相比, AUC 分别提高 0.9%和 1.6%, AUPR 分别提高 3.7%和 3.2%, 说明加入特定的元路径有助于模型学习到 HIN 特定的子结构, 从而提高DTIs 预测能力。在 GMDTI 中同时加入两条元路径后, 与只加入一条元路径相比, AUPR 至少提升4.2%。这是因为同时加入两条元路径时, 模型能学习到更多样的子结构, 捕获更丰富的语义信息, 从而更准确地预测DTIs。
表1 不同方法性能比较
Table 1 Comparison of different methods
方法AUC/%AUPR/% BLM-NII[9]82.947.5 HNM[9]88.558.6 MSCMF[9]82.359.3 DTINet[9]92.782.4 NeoDTI96.187.4 GADTI95.785.7 GMDTI (未加入元路径)95.786.6 GMDTI (+D-P-D)96.690.3 GMDTI (+P-D-P)97.389.8 GMDTI98.694.5
说明: +D-P-D表示只加入药物-靶标-药物元路径, +P-D-P表示只加入靶标-药物-靶标元路径。
由于 DTIs 的实际数据较为稀疏, 所以通过逐步增加负样本比例的方式模拟实际情况, 以便观察GMDTI 的性能表现。由于 NeoDTI 和 GADTI 与本文所提方法思路上较为相似, 并且基础实验结果比其他基线方法表现好, 因此后续实验中仅与 Neo-DTI 和 GADTI 两种方法进行比较。
如表 2 所示, 随着负样本比例逐步增加, NeoDTI, GADTI 和 GMDTI 的 AUC 均没有大的波动, 但三者的 AUPR 都明显下降, 说明负样本的数量会对模型的预测性能产生影响, 准确地选择对 DTIs 预测任务有利的负样本数量非常重要。相比于 NeoDTI 和GMDTI, GMDTI 的 AUPR 仍具有较大的优势。这是因为, 融入 HIN 的子结构信息和语义信息有利于模型在不平衡数据条件下探寻更全面的网络信息, 避免因网络节点的邻居过少而学不到更好的节点特征表示。这也证明 GMDTI 在稀疏 DTIs 网络中具有较好的表现能力。
另外, 实验结果显示 GADTI 的 AUC 和 AUPR优于 NeoDTI, 说明在不平衡数据集中, GADTI 通过融合节点的高阶邻居信息, 有助于提高 DTIs 预测能力。但是, 由于 GADTI 是通过重启随机游走的方法获得节点的高阶邻居信息, 容易捕获到与节点相关度较弱的“噪声”节点信息, 所以使得预测效果远不如GMDTI。
表2 逐步增加负样本比例的模型性能比较(%)
Table 2 Performance of different models in the case of gradually increasing negative samples (%)
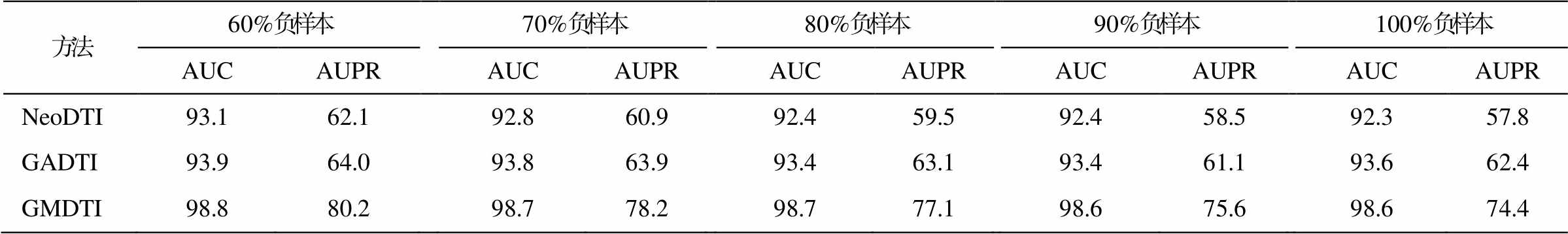
方法60%负样本70%负样本80%负样本90%负样本100%负样本 AUCAUPRAUCAUPRAUCAUPRAUCAUPRAUCAUPR NeoDTI93.162.192.860.992.459.592.458.592.357.8 GADTI93.964.093.863.993.463.193.461.193.662.4 GMDTI98.880.298.778.298.777.198.675.698.674.4
表3 模型鲁棒性实验(%)
Table 3 Robustness of GMDTI (%)
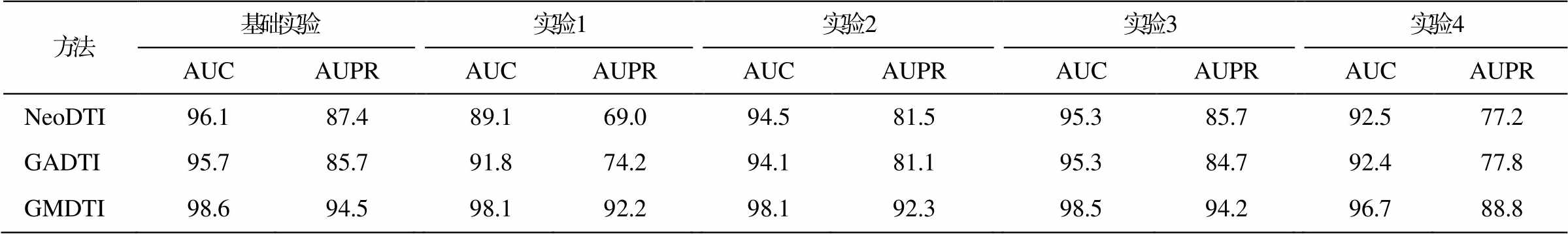
方法基础实验实验1实验2实验3实验4 AUCAUPRAUCAUPRAUCAUPRAUCAUPRAUCAUPR NeoDTI96.187.489.169.094.581.595.385.792.577.2 GADTI95.785.791.874.294.181.195.384.792.477.8 GMDTI98.694.598.192.298.192.398.594.296.788.8
说明: 基础实验的结果来源于表1。
数据集中可能包含“冗余的”DTI (即同一种靶标与一种以上类似的药物连接)。这种情况下, 药物靶标网络中冗余的 DTI 边可能造成 DTIs 预测性能的假性提升。为了证明本文所提模型的鲁棒性, 我们进行 4 种类型的 10 倍交叉验证实验。实验 1: 移除具有相似药物结构(两种药物化学结构的相似度>60%)或具有相似靶标结构(两种靶标序列的相似度>40%)的 DTI; 实验 2: 移除具有相似药物相互作用(Jaccard 相似度>60%)的 DTI; 实验 3: 移除具有相似副作用(Jaccard 相似度>60%)的 DTI; 实验 4: 移除与类似疾病相关的药物或靶标(即 Jaccard 相似度>60%)的 DTI。
实验结果如表 3 所示, 可以看出在去除“冗余DTI”数据后, 所有预测方法的性能均有所下降, 但GMDTI 的 AUC 和 AUPR 优于 NeoDTI 和 GADTI, 并且 AUPR 远高于 NeoDTI 和 GADTI。与去除“冗余 DTI”数据前的实验结果相比, GMDTI 模型的性能没有明显下降, 说明本文提出的模型在去除“冗余 DTI”数据的情况下仍然具有较好的预测性能, 鲁棒性较强。
为了充分利用 HIN 的子结构信息和节点间的语义信息, 本文设计药物-靶标-药物以及靶标-药物-靶标两条不同的元路径, 并提出一种新的模型GMDTI 来聚合 HIN 中节点的一阶邻居信息和元路径的语义信息。利用图神经网络, 更好地学习药物和靶标复杂的隐藏特征, 并通过端到端的方式, 同时优化特征提取过程和 DTIs 预测任务。实验结果表明, 与几个基线模型相比, GMDTI 具有更好的DTIs预测性能。
在加入所有负样本的实验中, GMDTI 的 AUC比基线模型至少提高 5.0%, AUPR 至少提高 12.0%, 证明利用元路径来捕获药物-靶标 HIN 中隐含的语义信息和子结构信息, 可以在稀疏网络中更好地预测 DTIs。
去除“冗余 DTI”数据后, GMDTI 模型的性能没有明显下降, 且结果远好于基线方法, 证明 GMDTI模型具有较强的鲁棒性。
本文方法目前仅使用二阶长度的元路径, 没有考虑更远距离的元路径。未来工作中将考虑利用不同类型、不同长度的元路径, 进一步提高模型的DTIs 预测性能。此外, 药物和靶标具有丰富的文本信息, 探索这些文本信息对 DTIs 预测的作用也是未来的研究工作之一。
参考文献
[1]Chen R, Liu X, Jin S, et al.Machine learning for drug-target interaction prediction.Molecules, 2018, 23(9): 2208
[2]Huang Y, Zhu L, Tan H, et al.Predicting drug-target on heterogeneous network with co-rank.Cham: Springer International Publishing, 2020
[3]Keiser M J, Roth B L, Armbruster B N, et al.Relating protein pharmacology by ligand chemistry.Nature Biotechnology, 2007, 25(2): 197-206
[4]Pujadas G, Vaque M, Ardevol A, et al.Protein-ligand docking: a review of recent advances and future perspectives.Current Pharmaceutical Analysis, 2008, 4(1): 1-19
[5]Li H, Gao Z, Kang L, et al.TarFisDock: a web server for identifying drug targets with docking approach.Nucleic Acids Research, 2006, 34(suppl 2): W219-W224
[6]Cheng A C, Coleman R G, Smyth K T, et al.Structure-based maximal affinity model predicts small-molecule druggability.Nature Biotechnology, 2007, 25(1): 71-75
[7]Bleakley K, Yamanishi Y.Supervised prediction of drug-target interactions using bipartite local models.Oxford: Oxford University Press, 2009
[8]Zheng X, Ding H, Mamitsuka H, et al.Collaborative matrix factorization with multiple similarities for predicting drug-target interactions // Proceedings of the 19th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining.San Fran-cisco, 2013: 1025-1033
[9]Luo Y, Zhao X, Zhou J, et al.A network integration approach for drug-target interaction prediction and computational drug repositioning from heterogeneous information.Nature Communications, 2017, 8(1): 1-13
[10]Wan F, Hong L, Xiao A, et al.NeoDTI: neural integration of neighbor information from a hetero-geneous network for discovering new drug-target interactions.Bioinformatics, 2019, 35(1): 104-111
[11]Zhou J, Cui G, Hu S, et al.Graph neural networks: a review of methods and applications.AI Open, 2020, 1: 57-81
[12]Liu Z, Chen Q, Lan W, et al.GADTI: graph auto-encoder approach for DTI prediction from hetero-geneous network.Frontiers in Genetics, 2021, 12: 650821
[13]Kip F T N, Welling M.Semi-supervised classifica-tion with graph convolutional networks [EB/OL].(2016‒09‒09) [2021‒03‒19].https://arxiv.org/abs/16 09.02907
[14]Sun Y, Han J, Yan X, et al.PathSim: meta path-based Top-K similarity search in heterogeneous information networks.Proceedings of the Vldb Endowment, 2011, 4(11): 992-1003
[15]Wang X, Bo D, Shi C, et al.A survey on hetero-geneous graph embedding: methods, techniques, app-lications and sources [EB/OL].(2020‒11‒30) [2021‒ 03‒17].https://arxiv.org/abs/2011.14867
[16]Knox C, Law V, Jewison T, et al.DrugBank 3.0: a comprehensive resource for ‘omics’ research on drugs.Nucleic Acids Research, 2010, 39(suppl 1): D1035-D1041
[17]Mei J P, Kwoh C K, Yang P, et al.Drug-target interac-tion prediction by learning from local information and neighbors.Bioinformatics, 2013, 29(2): 238-245
[18]Wang W, Yang S, Zhang X, et al.Drug repositioning by integrating target information through a hetero-geneous network model.Bioinformatics, 2014, 30 (20): 2923-2930
Drug-Target Interactions Prediction Based on Meta-path of Heterogeneous Information Network
Abstract The paper proposes a graph neural network model based on meta-path to predict drug target interactions(GMDTI).Firstly, based on drugs, targets, diseases and side effects in eight datasets, and the eight different types of action relationships between them, the authors construct a drug-target heterogeneous information network (HIN).Then, two different meta-paths are defined to capture the different sub-topology information of HIN and the latent semantic information between different nodes.Especially, the graph neural network method is applied to represent the node by aggregating the information of the first-order neighbor nodes and the nodes of the meta-path.Finally, DTIs prediction is completed effectively by end-to-end learning method.This method takes the first-order topology and the semantic information of meta-path of the drug-target HIN into account, which is helpful to learn more potential drug target relationships.The experiment results show that the proposed method achieves 98.6% in AUC and 94.5% in AUPR, which are higher than all baseline models.At the same time, GMDTI has better robustness than all baseline models by sparsity experiments of datas and reduction experiments of noise.
Key words drug-target interaction prediction; graph neural network; heterogeneous information network; meta-path; feature representation
doi: 10.13209/j.0479-8023.2021.105
收稿日期: 2021‒05‒08;
修回日期: 2021‒08‒09
国家自然科学基金(61402220)、湖南省自然科学基金(2020JJ4525)、湖南省教育厅重点科研项目(19A439)和南华大学研究生科研创新项目(213YXC007)资助