河口生态系统属于海洋生态系统, 是陆地和海洋的过渡带, 地球四大圈层的交汇处[1]。由于其特殊的位置, 河口生态系统具有环境复杂多变、较高的生物多样性和生产力以及受人类扰动程度大等特征, 因此河口生态系统是最特殊的海洋生态系统[2]。浮游细菌是水体中营浮游生活的原核微生物类群,它们在水生生态系统中具有极其重要的作用。浮游细菌是河口生态系统中多样性最高、生物量最大的生命形态[3], 也是生物地球化学循环、物质合成和分解以及能量流动的最重要的基础[4–5]。浮游细菌提供了最重要的河口生态系统服务功能。
目前, 珠江口区域的主要浮游细菌有放线菌门(Actinobacteria)、蓝细菌门(Cyanobacteria)、浮霉菌门(Planctomycete)、厚壁菌门(Firmicute)、绿菌门(Chlorobi)、拟杆菌门(Bacteroidetes)、脱铁菌门(Deferribacteres)、疣微菌门(Verrucomicrobia)、绿弯菌门(Chloroflexi)、变形菌门(Proteobacteria)和酸杆菌门(Acidobacteria)[6]。Wu 等[7]发现, 珠江河口表层水的优势类群为变形菌门的 γ-Proteobacteria 和厚壁菌门的芽孢菌属(Bacillus Clostridium), 而不动杆菌(Acinetobacter)作为优势物种分布相当广泛[8]。Zhang 等[9]比较研究珠江河口总群落(基于 DNA 分析)和活性群落(基于 RNA 分析), 发现变形菌门和蓝细菌门是优势类群。由微生物驱动的各种生态过程并非个体所能完成, 而是密切依赖于群落内部复杂交织的相互作用, 并受到环境条件的强烈影响,会造成浮游细菌种群结构和代谢功能的变化, 最终影响其生态系统服务功能。
生态位(ecological niche)又称生态龛, 表示生态系统中每种生物生存所必需的生境最小阈值。生物对食物和空间的竞争, 适应环境的物理因子限度,大体上决定了其地理的或生境的规模范围。生态位的概念于 1924 年由 Grinnell[10]首创, 主要强调时间和空间上的意义。1927 年, Elton[11]将其意义进一步拓展, 强调生态位概念中功能与营养的关系。在自然环境里, 每一个特定位置都有不同种类的生物,其活动以及与其他生物的关系取决于它所处的环境、特殊结构、生理和习性行为, 故形成自己独特的生态位[12]。生态中的重合生态位会导致趋同演化, 即两个物种虽然不是同一物种, 却各自独立地进化出相似的结果和功能[13], 因此同一生态位内部具有亲缘关系复杂的特征。河口中浮游细菌群落特征不仅具有显著的时空差异, 而且易受环境参数变化的影响, 形成特殊的共生网络。因此, 了解浮游细菌的群落特征对正确理解生态系统的状态有着十分重要的意义[14]。
16S rRNA 基因扩增子测序技术广泛应用于海洋[15]、湖泊[16]、水库[17]及河流[18]浮游细菌群落结构的研究中, 如丰度统计、组成变化、区域环境物种数目(Alpha 多样性)及沿地理、环境梯度基于物种有无和丰度的群落组成相异性(Beta 多样性), 能够表明时间、空间与环境参数对浮游细菌群落结构均有影响[19]。已有研究关注河口微生物多样性在空间和时间[20]尺度上的变化特征。在环境参数方面, 研究者阐述了盐度[21]、溶解氧[22]和营养盐[23–24]等环境因子的影响。其中, 在河口地区, 有研究表明, 盐度的变化对细菌的分布有直接影响[21]。盐度变化也是海湾和沿海等地区细菌群落结构变化较大的原因之一[25]。范艳君等[26]在研究珠江河口颗粒附着微生物群落沿环境梯度的空间分布特征时, 通过典范分析发现, 盐度和氮素相关营养盐水平是影响水体颗粒附着微生物群落分布格局的两个主要因素。
调查河口浮游细菌群落因环境参数而分化的生态位特征, 能够反映浮游细菌对环境变化的适应性, 对河口生态系统优化管理、生态环境可持续发展具有重要的科学意义。以往对珠江口环境因子和微生物群落的调查研究没有结合生态位理论和群落共发生网络情况, 对一定的环境限度内珠江口浮游细菌群落内部是否形成和形成怎样的生态位缺乏深入的探讨。因此, 本研究基于高通量测序技术,针对珠江口浮游细菌的 16S rRNA 基因序列进行多样性分析、共发生网络分析以及代谢功能预测分析。我们首先调查确定了珠江口区域对浮游细菌影响程度最大的单一环境参数为盐度, 并探究在不同盐度下, 各组间是否具有明显的多样性差异, 是否因此形成具有特殊的结构和生态功能的生态位群落, 从物种组成以及共发生网络方面, 对浮游细菌受环境干扰而在珠江口水体中形成的生态特征和模式提出新的见解。
1 研究区域与方法
1.1 研究区域基本情况
珠江口是典型的河口生态系统, 位于我国华南地区, 为我国华南人口最为集中的河口区域, 在我国的社会经济发展中具有重要的战略地位。该地区与香港和澳门共同构筑环珠江三角洲城市带, 组成粤港澳大湾区[27], 是一个典型的复合型的具有区域特性的生态与经济共繁荣系统[28]。在地理位置上,珠江口位于中国广东省海岸线的中部, 经纬度坐标范围在 21.50—22.45°N, 112.5—114.5°E 之间。珠江的河口区东起香港, 西达上川岛, 北起珠江各分流河口, 南至大壕岛和上川岛一线, 总面积约为 2600 km2。珠江三角洲地貌以发育河口放射状汉道为基本特征, 整个珠江三角洲由多个河口三角洲组合而成, 这种复合三角洲的地貌发育模式在我国各大河口中独具特色。珠江三角洲河网水系在出海处形成八大口门, 分别汇入伶仃洋、磨刀门施和崖门海区。
1.2 样品和环境参数的采集
本研究组于 2016 年 9 月在珠江口水域自上而下至海洋采取水样, 每个点取 3~4 个平行样, 分布于该区域各个方位的 9 个位置(图 1 中 P1~P9), 共 34个水样。每个样品就地取 15 L 水样, 取样时设置两个分组: 1) 水下 0.5 m 代表表层水(Surface), 水与沉积物交界处之上 1 m 代表底层水(Bottom); 2) 使用 PVDF 滤膜(Osmonics, 美国)过滤, 0.22 μm 滤膜过滤的样品代表自由生活态(free-living, FL)的细菌,3 μm 滤膜过滤的样品代表颗粒附着态(particleattached, PA)的细菌。水样用滤膜过滤后, 立即用锡箔纸包裹, 4°C 保存, 运至实验室, 放入−80°C 冰箱避光储存, 用于 DNA 提取。
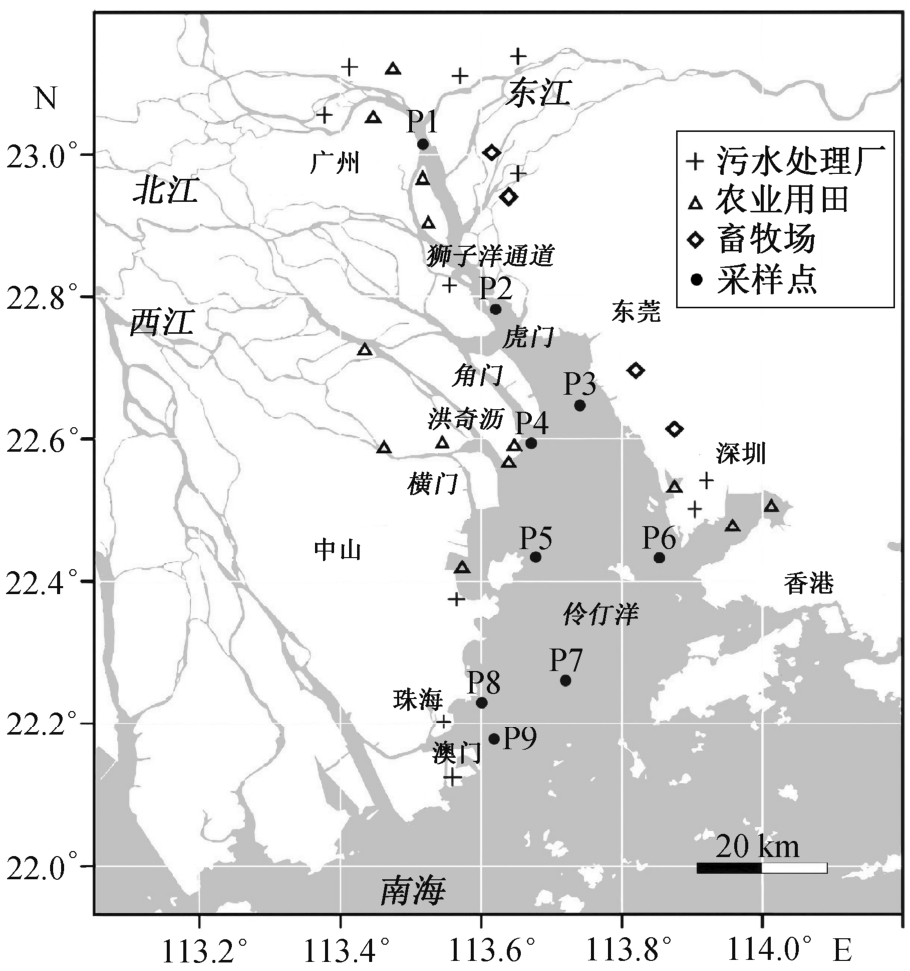
图1 采样点位置分布
Fig. 1 Locations and distribution of sampling sites
使用便携式盐度计 Thermo Eutech A301729,就地对样品盐度进行测量。使用 Vario EL cube 元素分析仪, 获取样品中总 C 和总 N 的含量(µg/g)。使用 pH 计, 实地获取样品的 pH 值。图 2 展示 C 和N 含量、pH 值和盐度等环境参数在各采样点的变化情况。
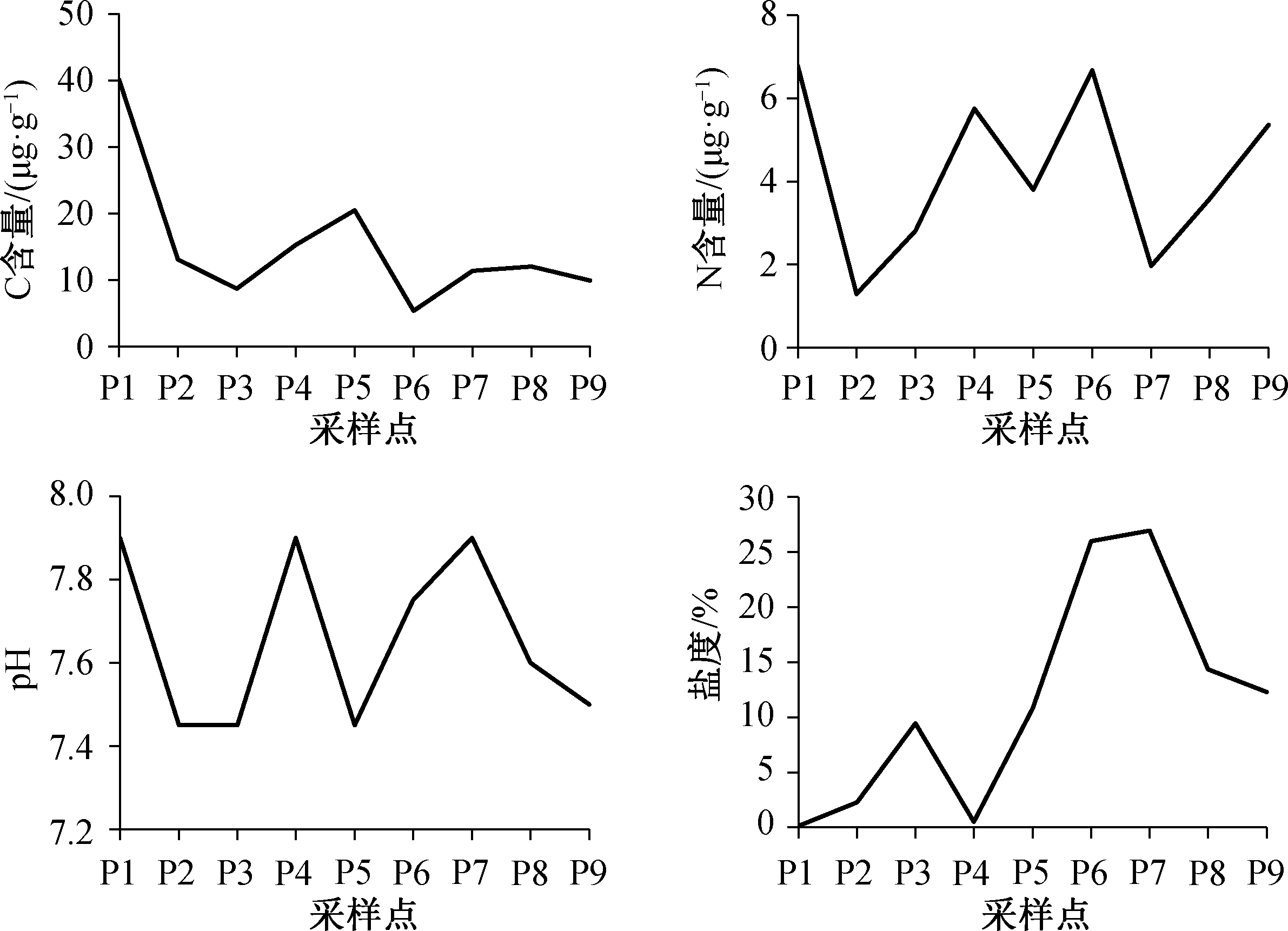
图2 采样点环境参数
Fig.2 Numerical environmental factors of sampling sites
1.3 DNA 的提取
将滤膜剪碎, 置于 1.5 mL 离心管中, 加入研磨珠及 Buffer, 在高速研磨仪上进行研磨。反复操作过滤后, 收集上清液, 作为待提取溶液。使用OMEGA Bacterial DNA Kit (OMEGA, 美国)和 Fast DNA SPIN Kit for Soil (MP Biomedicals, 法国)两种试剂盒进行 DNA 提取, 每个样品提取 4 个离心管的DNA, 分别溶于 90 µL DES 溶液中。在提取 DNA时, 通常伴随多种腐植酸类、多酚等 PCR 抑制物的残留, 可能对后续测序产生偏向性影响, 并夹杂扰乱 PCR 扩增的抑制因子, 因此需对提取的 DNA 样品进行纯化。由于纯化步骤会使所提取的 DNA 产生部分损失, 所以将所有 DNA 提取液收集起来, 分装于 200 µL 离心管中, 每个离心管再用于纯化。使用 PowerClean DNA Clean-Up Kit (MoBio, 美国)试剂盒, 对上述步骤得到的 DNA 进行纯化后, 溶解于90 µL DES 溶液中。保证样品的 DNA 浓度达到 100 ng, 在振荡器上微微震荡后放置, 使 DNA 沉淀在离心管底部, 再用新的干净离心管取 50 µL 保存, 用封口胶密封。
1.4 高通量测序及数据处理
取用 10 µg DNA 模板量, 对测序区域为 V3~V4区的目的区域进行扩增: 选择 V3~V4 区域对应的特异扩增引物(引物为 341F-805R), 继而对扩增出的目的片段进行富集, 同时加入特异 index 序列, 构建基因文库。使用 Qubit2.0 进行初步定量, 稀释文库至 1 ng/µL。使用 Agilent 2100 对文库的 insert size进行检测。insert size 符合预期后, 使用 Bio-RAD CFX 96 荧光定量 PCR 仪和 Bio-RAD KIT iQ SYBR GRN 进行 qPCR[29], 对文库的有效浓度进行准确的定量, 以保证文库的质量。选择测序策略 PE250,使用 Hiseq 对上述步骤的文库进行测序, 得到 16s 序列测序结果, 平均每个样品得到 47317 条高质量序列(范围为 36631 ~ 55226), 总计 1608797 条。
原始 16S rDNA 测序得到的序列数据长度为150 bp, 质量分数为 30 的序列占每组数据的 90%以上, 清洁率大于 90%。使用 FastQC 软件[30]进行质量查看后, 使用脚本对序列进行过滤。在 Linux 系统中使用 Qiime 2 软件[31], 对测序得到的 16S rRNA基因序列进行 OTUs (operational taxonomic units,操作分类单元)的过滤, 用收集得到的较高质量的OTUs 计算得到 OTU 丰度表以及物种分类信息表(Taxonomy)。
1.5 16s rRNA 基因分析
使用 Qiime2 获取 Alpha 多样性指数表, 在对数据进行 Shapiro-Wilk 正态性和 Bartlett 方差齐性检验后, 使用 R 软件中的 Phyloseq 包[32]对 Alpha 多样性指数进行可视化, 包括 Shannon 指数(代表丰富度)、Simpson 指数(代表均匀度, 值越接近 1 代表丰度分布越不均匀)和 PD_whole_tree 指数(代表遗传多样性, 值越大说明该分组下遗传的多样性越高)。基于有参分析的 Beta 多样性排序分析的变量解释度[33], 在 R 软件中查找引起微生物群落组成差异的关键环境因素, 再使用不同的 Beta 多样性分析聚类方法在环境参数上进行聚类。保留丰度大于0.01%的 OTUs 和对应的分类信息, 进行物种组成展示。
共发生网络构建过程分为 5 个步骤: 基础参数设置、相关系数标准化处理、网络获取、可视化及模块化分析。基础参数设置时, 不同 OTUs 之间的相关性选用 Pearson 和 Spearson 检验, 相似性选用Mutual information 方法, 距离矩阵选择 Bray Curtis和 Kullback-Leibler dissimlarity 度量法[34]。为了减少低丰度 OTUs 的影响, 使用 R 语言的 igraph 包对OTU 进行共发生网络参数计算, 过滤一些低丰度或低频的类群, 过滤条件如下: 1) 选择至少同时出现在 50%样品中的 OTUs[35]; 2) 将 Spearman 相关系数低于 0.7 的类群剔除, 即 r ≥0.7; 3) 保留相对丰度总和高于 0.005 的属。在进行相关系数标准化处理的时候, 对每一个交互和连线进行 100 次重新标准化和引导。以上步骤在 R 软件中使用 R 包 Hmisc[36]和igraph[37]进行操作和数据文件导出。可视化部分使用软件 Gephi 0.9.2[38]进行物种信息、交点、边和度等值的注释并导出。
2 结果与讨论
2.1 环境参数与微生物多样性关联分析
图 3(a)展示基于典范对应分析得到的珠江口浮游细菌 Beta 多样性的环境参数解释度比例, 为保证环境参数的丰富性, 将经纬度也纳入计算。非限制性排序只使用物种组成数据的排序, 代表生物因素的影响; 限制性排序同时使用物种和环境因子组成数据进行排序, 代表生物因素和非生物因素的共同影响。使用生物因素能够解释 78.2%的细菌多样性,生物因素和非生物因素共同作用能够解释的比例为21.8%。在非生物的单一环境参数中, 盐度能够解释 5.1%生物多样性, 其次是 pH (3.4%)、纬度(2.9%)、碳氮比(2.7%)、碳含量(2.7%)和氮含量(2.4%)。“非生物联合”表示本研究使用的非生物因素变量联合解释度。图 3(a)表明, 珠江口样品中, 单一变量对细菌的组成不构成主要影响, 但盐度是主要的单一环境因素。非生物因素变量联合的解释度反而最低, 说明本实验环境参数对样品的影响不在同一方向上。这一结果表明在水体的复杂环境下, 微生物群落的多样性是受多因素环境因子影响的, 但仍有个别单一因素具有重要影响。
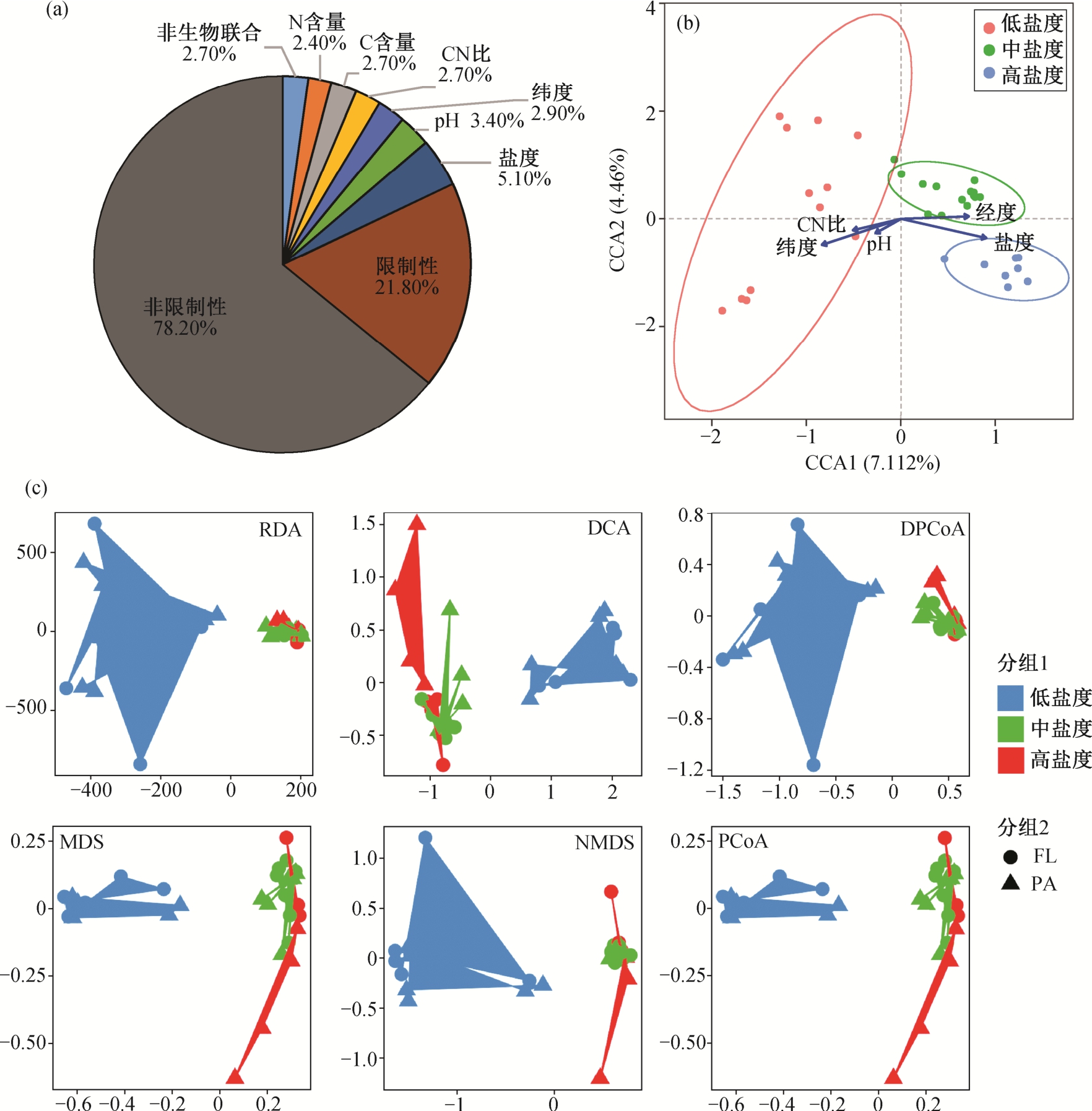
图3 Beta 多样性变量解释度
Fig. 3 Explanable degree of the variables on the Beta diversity
(a) CCA 单一环境变量解释度; (b) CCA 排序分析聚类; (c) Beta 多样性多种距离算法排序聚类
为进一步探究盐度的影响, 我们对盐度梯度进行分组。对河口盐度梯度的分组方法有以下两种:第一种, 按照淡水区(0.2‰~0.3‰)、混合区(0.7‰~17.7‰)及海水中的高盐度区(25.7‰~31.6‰)进行划分[39], 第二种, 根据淡水(0~1‰)、咸水(1~10‰)和盐水(10~100‰)进行划分[40]。本文结合已有的分组依据与本研究的样品数量和分布情况, 将盐度分为低盐度(0~10‰)、中盐度(10‰~20‰)以及高盐度(>20‰) 3 组(低、中、高是对于本文数据而言)。
典范对应分析聚类(CCA)结果表明, CCA 算法选取的最大影响程度的排序轴 CCA1 和 CCA2 分别能解释 7.11%和 4.46%的多样性变化。根据本文的盐度分组, 34 个样品在排序轴上具有明显的聚类效果, 低、中、高盐度样品在排序轴上能够完全地区分开。由于解释度过低, 图 3(b)中 R 语言可视化算法将 C 和 N 含量剔除, 由图可知, 盐度是排序轴上解释度最大的环境参数, 纬度也与盐度息息相关,且与排序轴的夹角为锐角, 与排序轴正相关。由于数据均符合有参和无参分析的要求(具有正态性和方差齐性), 因此选用非限制排序算法(只使用物种组成数据排序, 包括去趋势对应分析(DCA)、主坐标分析(PCoA)、非度量多维尺度分析(NMDS)、去趋势主成分分析(DPCoA)和多维尺度分析(MDS))以及限制性排序算法(同时使用物种和环境因子组成数据的排序, 包括主成分分析(RDA)、典范对应分析(CCA) 和等多重排序算法)进行联合比较(图 3(c))。可以发现, 珠江口数据在盐度分组上有很好的聚类效果和区分度, 且所有算法的聚类结果均为低盐度或中高盐度的数据具有明显的区分度, 说明无论是无参算法, 还是有参算法, 低盐度与中高盐度的微生物群落结构均具有明显的差异。
选取 Shannon, Simpson 和 PD_whole_tree 指数进行 Alpha 多样性分析, 统计显著性检验结果 p 值(p < 0.05 表示显著)。将盐度分组作为数据分组, 与采样时的浮游细菌状态(自由生活菌 FL 和颗粒附着菌 PA)和采样水层(底层和表层)两种设置进行 Alpha多样性及其组间显著性对比。图 4 显示, 浮游细菌状态和采样水层在 3 个 Alpha 多样性指数上的组间差异均不显著(p>0.05), 而盐度分组 3 个指数的 p 值均小于 0.05, 差异最明显(p 值最小)的为遗传多样性, 且低盐度浮游细菌的遗传多样性显著高于中高盐度。
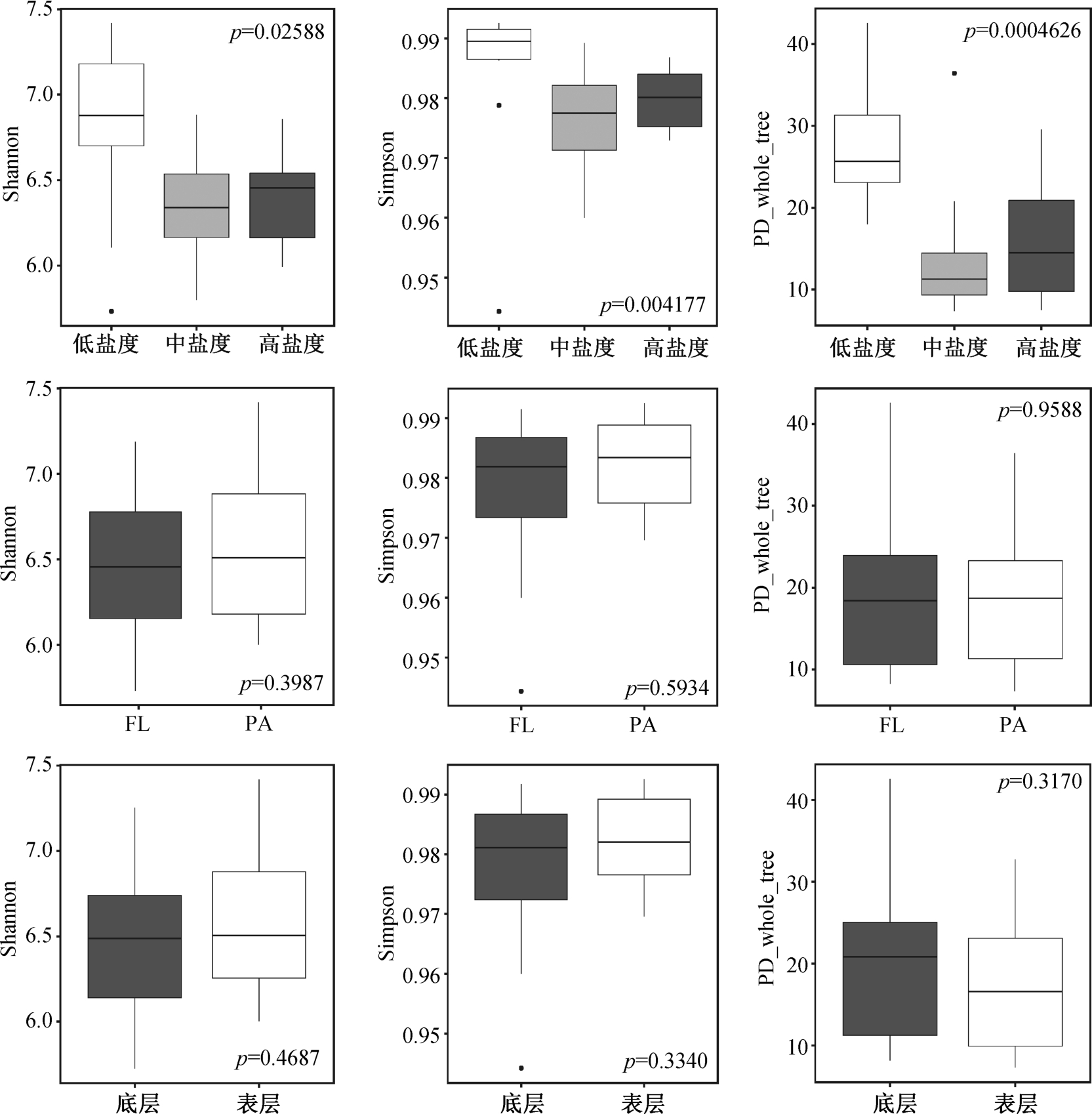
图4 组间Alpha 多样性差异比较
Fig. 4 Comparison of Alpha diversity between groups
2.2 珠江口浮游细菌物种组成
本实验中 16s OTU 原始数据的细菌分类学组成绝大多数是细菌(Bacteria), 包括 23 个门(Phylum),54 个纲(Class), 94 个目(Order), 160 个科(Family),250 个属(Genus), 269 个种(Species)。
在门水平上, 检出 9 个门类(图 5(a)), 包括变形杆菌门(Proteobacteria)(相对丰度为 16.2%~62.9%)、酸性杆菌门(Actinobacteria)(3.1%~46.7%)、拟杆菌门(Bacteroidetes)(5.2%~50.6%)、蓝菌门(Cyanobacteria) (0%~28.8%)、扁平菌门(Planctomycetes)(0%~11.2%)、疣微菌门(Verrucomicrobia)(0%~13.42%)、SAR406 菌门(0%~0.9%)、Qiime2 未识别菌门(0%~22.8%)以及其他相对丰度过低的门类(0%~10.8%)。
变形杆菌门(Proteobacteria)是本珠江口样品中丰度最大的菌门, 与 Zhang 等[41]的研究结果相符。变形杆菌门(Proteobacteria)包括 5 个纲(Class), 1 个未知菌纲, 占变形杆菌门(Proteobacteria)的相对丰度从大到小排序为 γ-变形菌纲(Gammaproteobacteria) (38.3%)、α-变形菌纲(α-Proteobacteria)(36.1%)、β-变形菌纲(β-Proteobacteria)(22.4%)、ε-变形菌纲(ε-Proteobacteria)(1.3%)、δ-变形菌纲(δ-Proteobacteria)(0.97%), 其他未知菌纲占 0.98% (图5(b))。
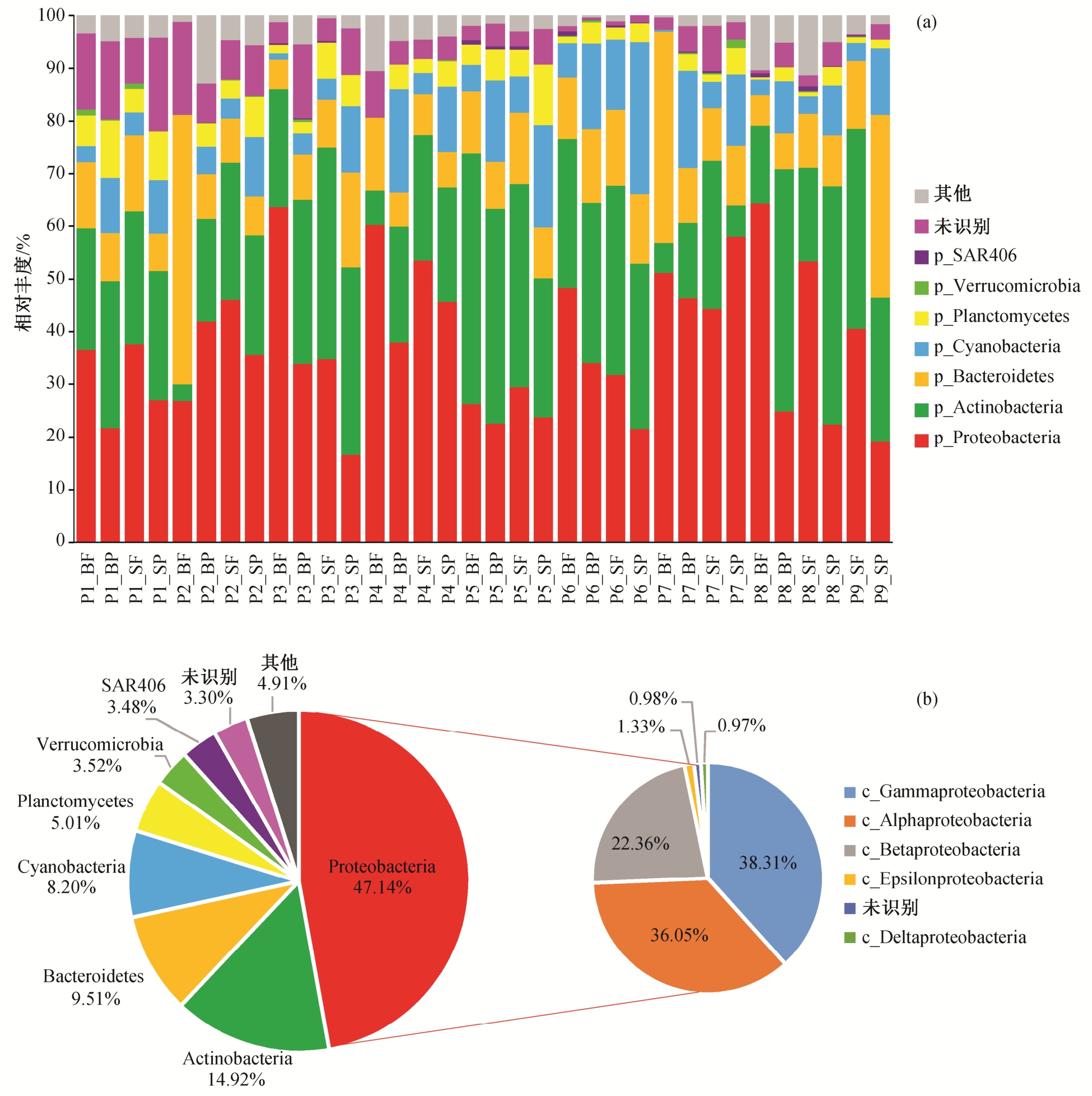
图5 珠江口浮游细菌的细菌组成
Fig. 5 Bacteria composition of plankonbacteria of PRE
(a) 总样本在门水平上的相对丰度; (b) 总样品的平均丰度比例及优势菌门Proteobacteria 的纲水平平均丰度比例
2.3 核心微生物物种组成
已有报道中对核心 OTU 的定义较为宽泛。Huse 等[42]将人体核心微生物群定义为出现在 95%样本中的 OTU, Hernandez-Agreda 等[43]; 有研究将珊瑚核心微生物定义为出现在 30%以上样本中的OTU; Shade 等[44]将在地下水输送管道中平均丰度大于 1%的微生物属定义为核心 OTU。结合已有研究和河口的环境微生物样本实际状况, 并严格保证筛选标准, 我们制定如下筛选原则: 总样品中, 核心 OTU 筛选条件为平均丰度 ≥0.02%, 且样品出现率≥70%(在 70%的样品中有丰度值); 分组样品中,核心 OTU 筛选条件为绝对丰度≥0.02%, 且样品出现率≥80%。
总样品中只有 otu1, otu2, otu3, otu13 和 otu14符合标准, 属于总样本核心 OTU。核心 OTU 在总样本中的丰度累计为 9.86%, 且主要分布在中盐度(19.67%)和高盐度(11.54%)的样品中, 低盐度样品(0.82%)中几乎没有(图 6(a))。5 种 OTU 均来自细菌中的酸性杆菌门(Actinobacteria)酸微菌纲(Acidimicrobiia)酸微菌目(Acidimicrobiales)的 OCS155 菌科类, 无法继续分类(图 6(b))。其中, otu1 为总样品核心菌科, 属于未培养的放线菌门 OCS155 科克隆的IMS3D85 16S 核糖体 RNA 基因的部分序列, 根据NCBI 数据库的序列信息, 该菌可能具有与颗粒附着细菌和自由生活菌的组成相关的酶功能, 且大多出现在沿海或近海[45]。由图 6(c)可知, 5 种总样本核心 OTU 在 3 个盐度分组中的分布情况较为类似。
在低盐度 OTU 中, 筛选出 8 个核心 OTU, 均为细菌, 分属于 3 个门, 5 个纲, OTU 的名字(ID)分别是 otu7, otu12, otu24, otu25, otu32, otu38, otu45 和otu55, 核心 OTU 在低盐度总样本中的丰度为11.67%。其中, otu7 (22.41%), otu12 (16.48%), otu25(9.93%), otu45 (9.18%)和 otu55 (8.72%)属于放线菌门, otu24 属于变形杆菌门, otu32 和 otu32 属于拟杆菌门。丰度最大的 otu7 属于放线菌门酸性菌纲酸微菌目 C111 科发光杆菌属 fluminis 种, 出现在所有低盐度样本中。
在中盐度 OTU 中, 筛选出的核心 OTU 分属 4个门, 比低盐度多一个蓝细菌门, 7 个纲, 5 个已知科。其中, 相对丰度最大的菌科来自酸性杆菌门(Actinobacteria)酸微菌纲(Acidimicrobia)的 OSC115 菌科(77.41%)。
在高盐度 OTU 中, 筛选出的核心 OTU 分属 4个门, 7 个纲, 5 个已知科, 相对丰度最大的同样是OSC115 菌科(52.7%), 丰度比中盐度组略低。属于蓝细菌的绿藻门(Cholorophyta)相对丰度排第二, 是中盐度的 10 倍。这两个科在低盐度核心 OTU 中均未出现。并且, 在总体样本上属于核心 OTU 的菌属(OSC115)在低盐度样本中均未出现(图 6(d))。在 Beta 多样性上, 聚类距离相对较近的中盐度和高盐度组(图 3(b)和(c))的核心 OTU 也只有 3 个菌科重复, 其余核心 OTU 均不重复。
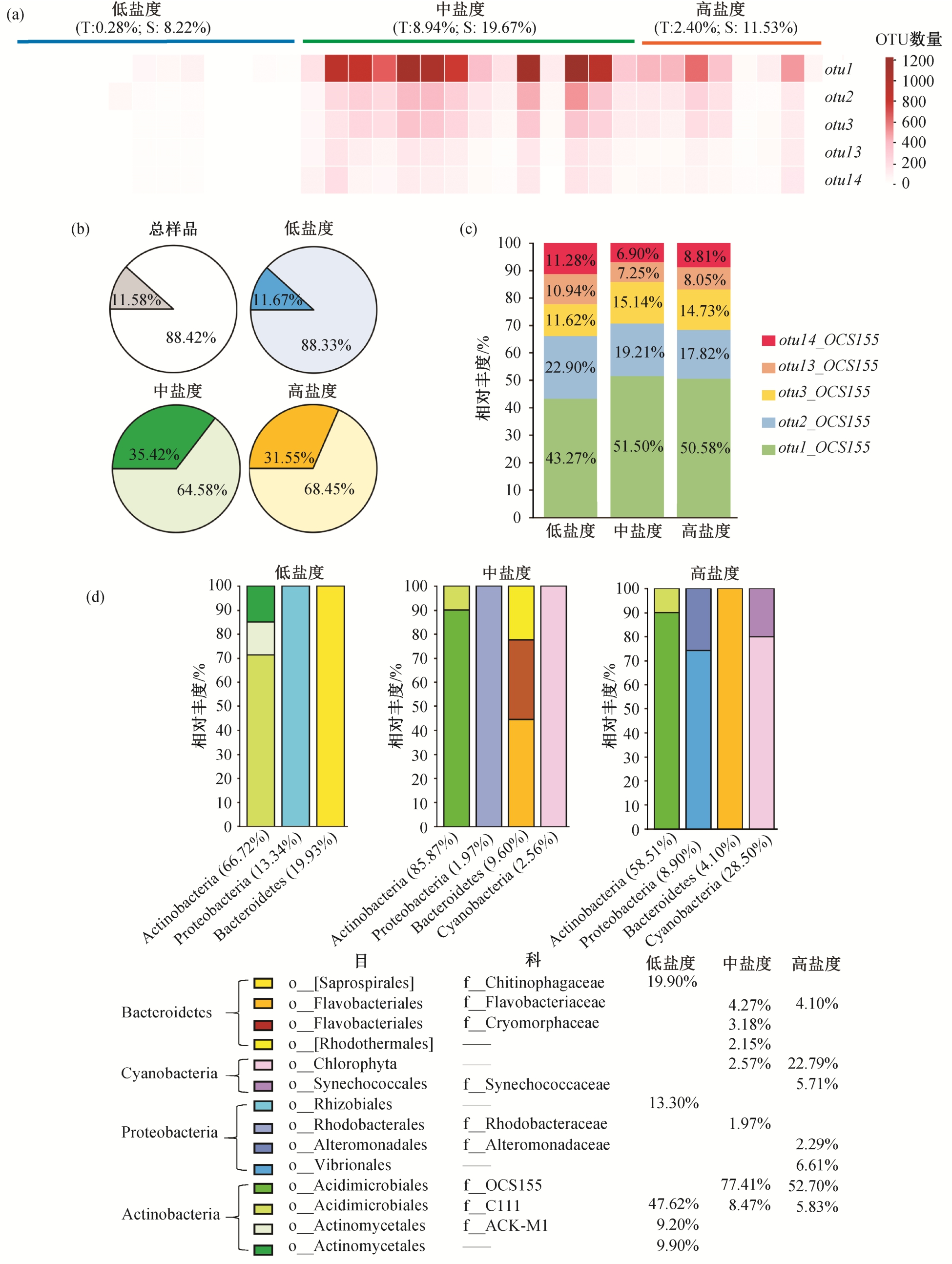
图6 盐度分组下各分组核心OTU 的物种组成
Fig. 6 Taxonomic composition of core OTUs on salinity groups
(a) 总样品核心 OTU 在所有样品中的丰度分布情况; (b) 总样品核心 OTU 和盐度样品核心 OTU 的相对丰度占比; (c) 总样品核心 OTU 在各盐度分组中的分布; (d) 各盐度样品核心 OTU 在物种组成
2.4 细分盐度分组下的共发生网络关系
根据核心 OTU 的细菌组成可以判断, 珠江口微生物群落在盐度分组上具有明显的差异, 根据生态位理念, 我们猜测珠江口浮游细菌在盐度这一环境因素的影响下形成特殊的生态位, 即微生物的“社区”。因此, 本节根据 OTU 丰度表和分类学信息, 结合低中高盐度分组, 通过取交、并、补集进一步细分盐度组, 以便从微生物共存与否的角度观察生态位独立与否。
用隶属于不同科的 OTUs 构建浮游细菌共发生网络, 所有数据分为 3 个盐度生态位组, 分别代表低盐度、中盐度和高盐度样品。使用 Venn 图可视化结果获得 7 个细分盐度分组, 分别是 Shared (在低中高盐度组均存在的 OTU, 75 个)、L/M/H-Only(仅在低、中、高盐度组中单独存在的 OTU, 分别有 415, 269 和 189 个)、L-M/H、M-H (仅存在于两种盐度中的 OTU, 低–中/高、中–高盐度组分别有125, 27 和 109 个)(图 7(a))。结果表明, Shared 中OTU 较少, 但相对丰度最高(25.59%), 高于其他OTU 数更大的组; L-H 的 OTU 只有 27 个, 且丰度最少(1.8%)(表 1), 说明低盐度与高盐度物种差异较大, 本实验划分的盐度具有较好的区分性。共存的分组 L-M 和 M-H 的丰度分别为 16.36%和 16.10%,仅次于 Shared, 而 L-Only 不仅具有最多的 OTU 数(415 个), 还具有较高的相对丰度(12.44%), 表明珠江口水体中大多数细菌生活在盐度环境相对宽泛的生态位中, 但有相当丰富的浮游细菌只生活在低盐度环境下。
表1 细分盐度分组及其OTU 信息
Table 1 Sub-divided salinity groups and its OTU information

说明: Low, Middle 和High 分别为低、中、高盐度组。∩表示交集,∪表示并集, CAB∪C 表示 A∪B∪C 作为全集, 除去 B∪C 后剩下的来自 A 的补集, 如图7(a)所示。
盐度区域 存在盐度区域 OTU 数量 丰度/%Shared Low∩Middle∩High 75 25.59 L-M CLow∩Middle High 125 16.36 L-H CLow∩High Middle 27 1.80 M-H CMiddle∩High Low 109 16.10 L-Only CLow Middle∪High 415 12.44 M-Only CMiddle Low∪High 269 8.25 H-Only CHigh Low∪Middle 189 4.67
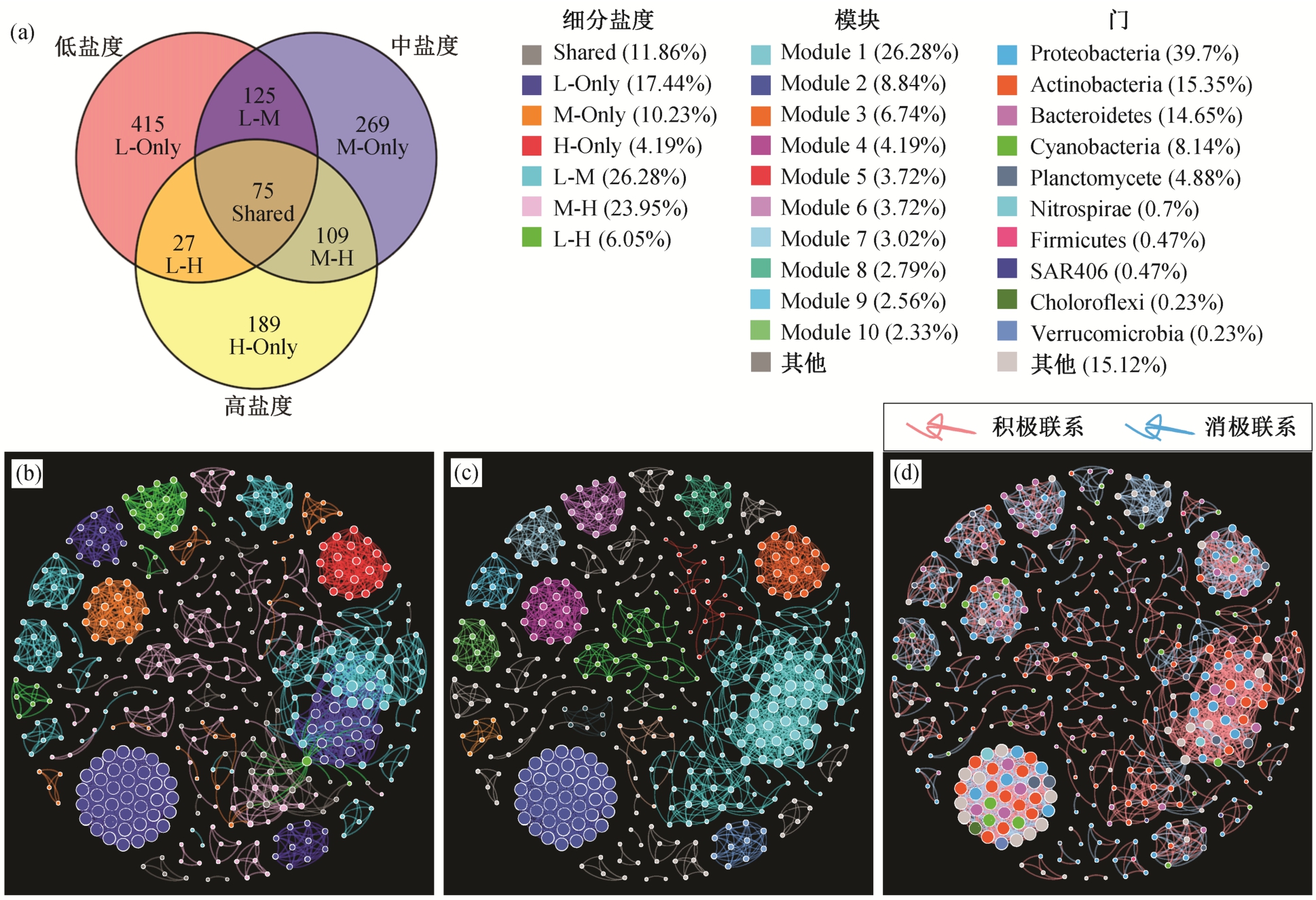
图7 细分盐度分组共发生网络分析
Fig. 7 Co-occurrence network of Sub-divided salinity groups
(a) 交叉细分盐度分组 Venn 图; (b) 根据细分盐度分组注释的共发生网络图; (c) 根据紧密度模拟的群落 Modules 注释的共发生网络图; (d) 根据OTU 的菌门注释的共发生网络图
共发生网络模块可以反映栖息地的异质性、系统发育上亲缘关系较近物种的聚集、生态位的重叠和物种的共进化, 被认为是系统发育、进化或功能的独立单元[34]。珠江口浮游细菌共发生网络被划分成多个模块, 模块是网络中高度连接的区域。图7(b)是基于细分生态位进行着色; 图 7(c)是对紧密度模拟计算出的微生物模块进行着色; 图 7(d)根据浮游细菌菌门进行着色, 平均丰度小于 0.22%的菌门归类到其他组。网络图中每个节点代表一个 OTU,节点越大代表“度”越大, “度”是反映该 OTU 与其他 OTU 的连接数的指标; 网络图中的连线(边)代表 OTU 之间的相互关系。图 7(d)中对“边”–OTU 的相互关系进行区分着色, 红色表示积极联系, 蓝色表示消极联系。积极联系包括互惠互利的共处、互生和共生等促进关系, 消极联系包括竞争、拮抗和捕食等抑制关系。通过 R 语言 igraph 包[37]和 Gephi软件[38]构建的珠江口浮游细菌网络图的基本参数如表 2 所示。
表2 细菌共发生网络拓扑学参数
Table 2 Topological parameters of co-occurrence network
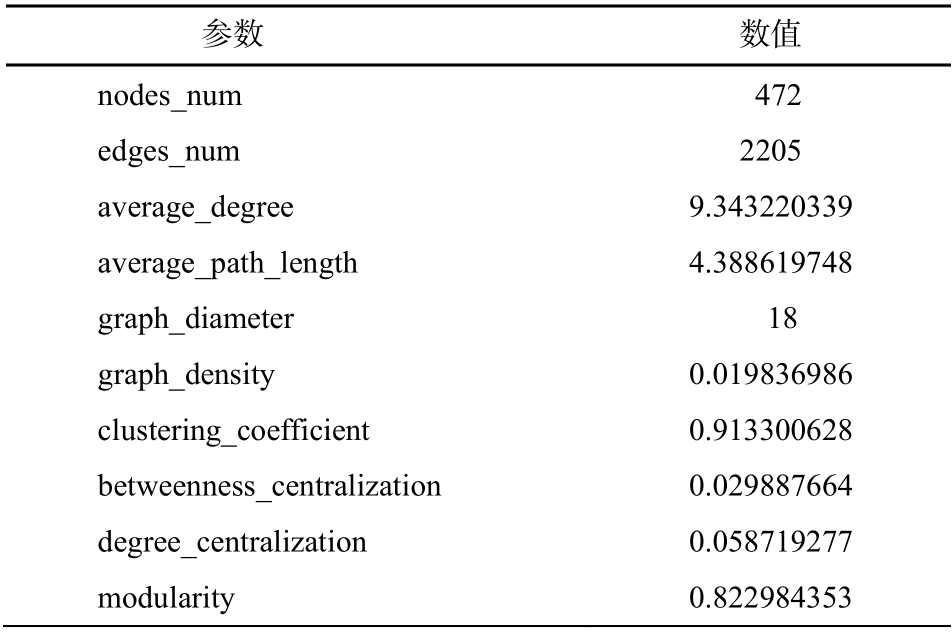
参数 数值nodes_num 472 edges_num 2205 average_degree 9.343220339 average_path_length 4.388619748 graph_diameter 18 graph_density 0.019836986 clustering_coefficient 0.913300628 betweenness_centralization 0.029887664 degree_centralization 0.058719277 modularity 0.822984353
Shared 是丰度最大的微生物簇, 经过低频筛选,其比例减少至 11.86%, 且几乎在网络关系中看不到紧密联系 Shared 组的 OTU, 其在样品中分布广, 联系松散。而 L-Only 与 Module2、M-Only 与 Module 4、H-Only 与 Module3、L-H 与 Module6 以及部分L-Only 与 Module7 等模块完全重合, 说明这些物种形成独立且紧密联系的微生物簇。从图 7(d)可知,每个模块中, 物种组成复杂, 紧密的模块不由单一分类的浮游细菌组成(如 L-Only 模块中由 Actinobacteria, Cyanobacteria, Procteobacteria, Planctomytcete 及大量其他菌门组成), 这些浮游细菌因为低盐度这一环境因素, 在珠江口河口生态系统中自发地形成一个多元的微生物“社区”, 这些物种并非亲缘相似, 而是因为盐度生态位形成独立的功能单元。Module1 是丰度最大的模块, 但由部分 LOnly、L-M 及少量其他细分盐度组的 OTU 组成的紧密程度比 Module2 松散, 与其他模块的边界不清晰。一个共生网络中, 积极和消极联系应该处于平衡状态。从图 7(d)可知, Module1 内部的积极联系占据主要地位, 因此 Module1 的度较小, 各个节点之间联系松散(边较长且边数少), 该网络不够稳定。由图 7(b) 可知, Module1 内部来自两个细分盐度生态位, 形成重合生态位, 推测该模块会因为共生关系的单一而逐渐分化, 其他紧密联系的模块中积极和消极联系的数量大致处于平衡状态, 形成具有相对稳定生态位共生群落。珠江口浮游细菌的共发生网络表明, 本文对盐度的细分具有科学意义,珠江口的浮游细菌群落在盐度上形成内部紧密联系、亲缘关系复杂的独立生态位。其中, 仅存在低盐度中的微生物群落形成的网络最为紧密, 内部共生关系最为平衡。
3 结论
本文利用 16s rRNA 基因序列分析, 研究珠江口水域(从珠江下游至入海口)水体中浮游细菌的微生物多样性以及主要影响的单一环境参数, 并据此进行优势菌和核心菌的物种组成分析和共发生网络分析, 得到如下结论。
1) 盐度是影响珠江口浮游细菌多样性的主要单一环境参数。根据 Beta 多样性聚类, 珠江口水体中浮游细菌在低(0~10‰)、中(10‰~20‰)、高(>20‰)盐度聚类成 3 个群落, 在这 3 个群落中具有显著的遗传多样性, 其中低盐度浮游细菌的遗传多样性显著高于中高盐度。
2) 珠江口浮游细菌的优势菌门依次为变形杆菌 门(Proteobacteria) 、 酸 性 杆 菌 门(Actinobacteria)、拟杆菌门(Bacteroidetes)和蓝菌门(Cyanobacteria)。总样品中核心菌为酸杆菌门的 OCS155, 是一种未培养的细菌。优势菌侧重于数量多, 核心菌兼具数量多和分布广。二者均与研究区域的主要生态功能息息相关, 核心菌具有更加全面的意义。
3) 低盐度中主要的核心菌是 C111, 中高盐度的核心菌是 OCS11。3 种盐度中的核心菌具有较少冗余的分类学特征, 尤其是低盐度细菌与中高盐度的核心菌完全不重合。据此, 本文假设珠江口浮游细菌因盐度形成特殊的生态位, 并进行细分, 通过共发生网络图得知, 珠江口浮游细菌在细分盐度组上形成联系紧密的共发生网络, 其中, 仅存在于低盐度的 OTU 紧密度最高。
4) 各个共发生网络模块联系紧密且亲缘关系复杂, 不同物种分布于不同的模块, 形成多元的特殊微生物簇, 与生态位理论中重合生态位的趋同演化行为相符, 验证了珠江口浮游细菌在盐度上形成特殊生态位的假设。
[1] Tait E, Carman M, Sievert S M. Phylogenetic diversity of bacteria associated with ascidians in Eel Pond(Woods Hole, Massachusetts, USA). Journal of Experimental Marine Biology and Ecology, 2007, 342(1):138–146
[2] 陆健健. 河口生态学. 北京: 海洋出版社, 2003
[3] 马丽丽, 淡水生态环境中的浮游微生物检测及群落结构变化研究[D]. 天津: 南开大学, 2015
[4] Falkowski P G, Fenchel T, Delong E F. The microbial engines that drive Earth’s biogeochemical cycles. Science, 2008, 320: 1034–1039
[5] Fuhrman J A, Steele J A, Hewson I, et al. A latitudinal diversity gradient in planktonic marine bacteria.PNAS, 2008, 105(22): 7774–7778
[6] 刘诚刚, 宁修仁, 蔡昱明, 等. 南海北部及珠江口细菌生产力研究. 海洋学报, 2007, 29(2): 112–122
[7] Wu M, Song L S, Ren J P, et al. Assessment of microbial dynamics in the Pearl River Estuary by 16S rRNA terminal restriction fragment analysis. Continental Shelf Research, 2004, 24(16): 1925–1934
[8] 王松鸽, 珠江河口浮游细菌群落结构及影响因素[D]. 上海: 上海海洋大学, 2019
[9] Zhang W P, Bougouffa S, Wang Yong, et al. Toward understanding the dynamics of microbial communities in an estuarine system. Plos One, 2014, 9(4): e94449
[10] Grinnell J. Geography and evolution. Ecology, 1924,5: 225–229
[11] Elton C. Animal ecology. London: Sidgewick & Jackson, 1927: 204
[12] 李㛃, 朱金兆, 朱清科. 生态位理论及其测度研究进展. 北京林业大学学报, 2003, 25(1): 100–107
[13] 张峰, 上官铁梁. 翅果油树群落优势种群生态位分析. 西北植物学报, 2004, 24(1): 70–74
[14] Carpenter S R, Stanley E H, vander Zanden M J. State of the world’s freshwater ecosystems: physical, chemical, and biological changes. Annual Review of Environment and Resources, 2011, 36: 75–99
[15] Mo Yuanyuan, Zhang Wenjing, Yang Jun, et al. Biogeographic patterns of abundant and rare bacterioplankton in three subtropical bays resulting from selective and neutral processes. ISME Journal, 2018, 12(9): 2198–2210
[16] Song J X, Xu Z X, Liu C M, et al. Ecological and environmental instream flow requirements for the Wei River — the largest tributary of the Yellow River.Hydrological Processes, 2007, 21(8): 1066–1073
[17] Liu Lemian, Yang Jun, Yu Zheng, et al. The biogeography of abundant and rare bacterioplankton in the lakes and reservoirs of China. ISME Journal, 2015,9: 2068–2077
[18] Kolmakova O V, Gladyshev M I, Rozanov A S, et al.Spatial biodiversity of bacteria along the largest Arctic river determined by next-generation sequencing.FEMS Microbiol Ecol, 2014, 89(2): 442–450
[19] Ruiz-Gonzalez C, Proia L, Ferrera I, et al. Effects of large river dam regulation on bacterioplankton community structure. FEMS Microbiology Ecology, 2013,84(2): 316–331
[20] Canion A, Kostka J E, Gihring T M, et al. Temperature response of denitrification and anammox reveals the adaptation of microbial communities to in situ temperatures in permeable marine sediments that span 50°in latitude. Biogeosciences, 2014, 11: 309–320
[21] Santos L, Vaz L, Gomes N C M, et al. Impact of freshwater inflow on bacterial abundance and activity in the estuarine system Ria de Aveiro. Estuarine Coastal and Shelf Science, 2014, 138: 107–120
[22] Zehr J P, Kudela R M. Nitrogen cycle of the open ocean: from genes to ecosystems. Annual Review of Marine Science, 2011, 3(1): 197–225
[23] 李佳霖. 典型河口区沉积物的硝化和反硝化过程[D]. 青岛: 中国海洋大学, 2009
[24] Kearns P J, Weston N B, Bowen J L, et al. Tidal freshwater marshes harbor phylogenetically unique clades of sulfate reducers that are resistant to climatechange-induced salinity intrusion. Estuaries and Coasts, 2016, 39(4): 981–991
[25] Sharp J H, Yoshiyama K, Parker A E, et al. A biogeochemical view of estuarine eutrophication: seasonal and spatial trends and correlations in the Delaware Estuary. Estuaries and Coasts, 2009. 32(6): 1023–1043
[26] 范艳君, 朱玲. 珠江口颗粒附着微生物群落结构的研究//2010 年中国水产学会学术年会. 西安, 2010:63
[27] 李锐仪. 珠江广州河段沉积物中氮营养物质的时空分布特征. 环境, 2014(增刊1): 50–52
[28] 姜丽晶. 珠江口沉积物微生物多样性及其与环境相互关系的研究[D]. 厦门: 国家海洋局第三海洋研究所, 2007
[29] Bustin S A, Vladimir B, Garson J A, et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clinical Chemistry, 2009, 55(4): 611–622
[30] Brown J, Pirrung M, McCue L A. FQC dashboard:integrates FastQC results into a web-based, interactive, and extensible FASTQ quality control tool.Bioinformatics, 2017, 33(19): 3137–3139
[31] Bolyen E, Rideout J R, Dillon M R, et al. Reproducible, interactive, scalable and extensible microbiome data science using QIIME 2. Nature Biotechnology, 2019. 37: 852–857
[32] Mcmurdie P J, Holmes S. PHYLOSEQ: a bioconductor package for handling and analysis of highthroughput phylogenetic sequence data // Altman R B,Dunker A K, Hunter L, et al. Pacific Symposium on Biocomputing. Kohala Coast, Hawaii, 2012: 235–246
[33] Hitchcock J N, Mitrovic S M, Kobayashi T, et al.Responses of estuarine bacterioplankton, phytoplankton and zooplankton to dissolved organic carbon(DOC) and inorganic nutrient additions. Estuaries and Coasts, 2010, 33(1): 78–91
[34] 张宝刚. 中国大豆根系微生物的生物地理分布和共发生模式研究[D]. 杨凌: 西北农林科技大学, 2018
[35] Zhou Jizhong, Deng Ye, Luo Feng, et al. Phylogenetic molecular ecological network of soil microbial communities in response to elevated CO2. MBio, 2011,2(4): e00122-11
[36] Harrell Jr F E, Harrell Jr M F E. Package ‘hmisc’.CRAN2018, 2019: 235-6
[37] Csárdi G, Nepusz T. The igraph software package for complex network research. Interjournal Complex Systems, 2006, 1695(5): 1–9
[38] Bastian M, Heymann S, Jacomy M. Gephi: an open source software for exploring and manipulating networks [C/OL] // Third International AAAI Conference on Weblogs and Social Media. San Jose, California,2009 [2020–06–01]. https://www.aaai.org/ocs/index.php/ICWSM/09/paper/viewPaper/154
[39] Chen, S., Wang P, Liu H, et al. Population dynamics of methanogens and methanotrophs along the salinity gradient in Pearl River Estuary: implications for methane metabolism. Applied Microbiology and Biotechnology, 2020, 104(3): 1331–1346
[40] Sang S, Zhang X, Dai H, et al. Diversity and predictive metabolic pathways of the prokaryotic microbial community along a groundwater salinity gradient of the Pearl River Delta, China. Scientific Reports,2018, 8: 17317
[41] Zhang Y, Zhao Z, Dai M, et al. Drivers shaping the diversity and biogeography of total and active bacterial communities in the South China Sea. Molecular Ecology, 2014, 23(9): 2260–2274
[42] Huse S M, Ye Y. A core human microbiome as viewed through 16S rRNA sequence clusters. PLoS One,2012, 7(6): e34242
[43] Hernandez-Agreda A, Gates R D, Ainsworth T D.Defining the core microbiome in corals’ microbial soup. Trends in Microbiology, 2017, 25(2): 125–140
[44] Shade A, Handelsman J. Beyond the Venn diagram:the hunt for a core microbiome. Environmental Microbiology, 2012, 14(1): 4–12
[45] D’Ambrosio L, Ziervogel K, MacGregor B, et al.Composition and enzymatic function of particleassociated and free-living bacteria: a coastal/offshore comparison. ISME Journal, 2014, 8(11): 2167–2179