
图1 用于培养生物膜的FlowCell系统装置
Fig. 1 Flow Cell system used for culturing biofilm
摘要 采用流动室(Flow Cell)生物膜发生装置和激光共聚焦显微镜观察技术, 揭示不同环境条件下含氮杂环化合物(nitrogenous heterocyclic compounds, NHCs)降解菌的生物膜形成特征。结果表明, 增加初始接种菌液浓度并延长初始黏附时间, 有利于吡啶降解菌在基质表面的黏附和生物膜形成; 降低培养基流速, 细菌生物膜更加均质化且形成水通道结构; 不同条件下的生物膜都存在内层活细胞比例较低, 外层活细胞比例较高的规律。此外, 还发现相对于单一菌株, 混合菌株在生物膜厚度、基质覆盖率与活细胞比例上都更具优势。目标 NHC 浓度对喹啉降解菌生物膜的形态和活性也有明显的影响: 低浓度下降解菌团聚结构分布较零散, 但具有较大的体积; 高浓度下降解菌团聚结构明显变小, 在基质表面的分布则更均匀; 低浓度下生物膜的活细胞比例显著高于高浓度下的生物膜。
关键词 生物膜; 降解菌; 含氮杂环化合物; 流动室系统
含氮杂环化合物(nitrogenous heterocyclic com-pounds, NHCs)是煤化工行业(如焦化和煤气化等企业)生产排放的废水中一类典型的有机污染物[1]; 同时, 吡啶、喹啉和吲哚等 NHC 也是重要的化工原料, 广泛用于染料、农药、消毒剂和防冻剂以及其他日用化学品的生产[2]。研究表明, NHC 类物质一般具有较高的毒性、致突变性和致癌性, 对生态系统中各类生命体有潜在的危害作用[3]; NHC 类物质的结构比较稳定, 难以被微生物分解利用, 且杂环的结构使其极性高, 水溶性好, 更容易在水体和土壤等环境介质中迁移扩散[4], 造成更大的环境风险。传统的废水生物处理系统难以对 NHC 类物质进行有效的处理, 使其成为制约相关行业绿色可持续发展的瓶颈问题。
采用基于高效降解菌的生物强化技术来处理难降解有机物, 是一种有效且经济的改进策略。目前, 已有大量的研究从各类环境介质中分离筛选出针对NHC 类物质的高效降解菌, 如从受污染土壤中分离的 Micrococcus luteus[5]、Rhizobium 属[6]吡啶降解菌和 Dietzia cinnamea[7]、Bacillus 属[8]喹啉降解菌, 从焦化废水中分离的 Shinella zoogloeoides[9]、Paracoc-cus属[10]吡啶降解菌和 Pseudomonas 属[11]、Rhodoco-ccus 属[12]喹啉降解菌等。然而, 高效降解菌是以菌剂的形式直接投加到生物强化处理体系中, 往往在长期运行过程中会逐渐流失, 不会成为系统中主导的细菌物种[13-14], 可能会影响其对相应污染物的稳定处理。有研究发现, 在序批式生物膜反应器中, 通过促进高效降解菌形成生物膜(biofilm),可以显著地提升该系统的耐冲击负荷能力和对目标有机污染物的处理效率[15]。
生物膜是细菌等微生物中广泛存在的一种群体性生长形式, 指微生物细胞通过黏附在基质材料表面并分泌胞外聚合物(extracellular polymeric subs-tance, EPS)将自身包裹, 从而形成具有一定结构的聚集体[16]。全球 40%~80%的细胞存在于生物膜中, 这种活跃的生命形态驱动着几乎所有的地球生物化学循环[17]。研究证明, 与游离的微生物相比, 生物膜可以提供诸多好处, 例如增强细胞之间的交流与合作, 提高对有毒有害物质的抵御能力, EPS 中丰富的胞外降解酶可强化对难降解有机物的利用能力等[18]。因此, 生物膜在废水处理工程中也得到广泛的应用, 但污染环境中高效降解菌的生物膜形成过程和机制尚未充分探明。
目前, 研究细菌生物膜形成的实验室装置和方法有多种, 如在微孔板体系中培养并定量生物膜[19], 在摇瓶中加入载体材料(玻璃珠、玻璃片等)培养生物膜[20-21], 但这些方法都属于封闭体系的批式培养, 与实际的环境条件相差较远。在流动室(Flow Cell)系统[22]和 CDC 生物膜系统[23]等连续培养体系中, 可以通过实时调节营养成分、水力条件等培养参数, 更好地模拟实际的动态环境。特别地, Flow Cell 系统培养出的生物膜还可以在保持结构不被破坏的前提下, 在原位即时地进行荧光染色和显微镜观察[24], 从而提供了更加方便和精准的研究手段。但是, 针对高效降解菌的生物膜形成过程和机制, 采用 Flow Cell 系统的研究案例还比较少。
本研究针对 4 株实验室保藏的吡啶或喹啉高效降解菌, 采用 Flow Cell系统培养生物膜, 结合荧光染色和激光共聚焦显微镜(confocal laser scanning microscopy, CLSM)观察成像技术, 探究不同运行参数下高效降解菌的生物膜形成特征, 并识别 NHC类污染物浓度对相应菌株生物膜形成的影响。本研究将为废水处理系统中高效降解菌生物膜的实际应用提供理论依据, 同时建立的以 Flow Cell 系统为基础的生物膜研究方法可为相关研究提供借鉴。
吡啶和喹啉购自北京化工厂(中国), 胰蛋白胨和酵母提取物购自 Oxoid (英国), 琼脂购自 Sigma-Aldrich (日本), 硫酸卡那霉素购自天根生化(中国), 荧光染料 LIVE/DEAD BacLightTM Bacterial Viability Kit 购自 Invitrogen (美国)。在菌株富集培养和生物膜培养的实验中, 主要采用 Luria-Bertani(LB)液体培养基[25]; 同时, 为避免杂菌污染, 在 LB 培养基中加入 20 mg/L的硫酸卡那霉素和 200 mg/L 的吡啶或喹啉。培养基及盛装的器皿在使用前用 121oC, 1.05 kg/cm2的高温高压蒸汽灭菌20分钟以上。
本研究选用的菌株是从焦化废水处理系统活性污泥中经驯化分离而来的吡啶和喹啉高效降解菌, 分别是吡啶降解菌 Shinella zoogloeoides BC026 和Paracoccus sp. BW001, 喹啉降解菌 Pseudomonas sp. BW003 和 Pseudomonas sp. BW004, 这些菌株在中国普通微生物菌种保藏管理中心的保藏号分别为CGMCC2224, CGMCC2225, CGMCC3008 和CGMC C4221。这 4 株降解菌均为好氧生长, 对硫酸卡那霉素具有抗性。
1.3.1装置组成与搭建
本研究采用的 Flow Cell 系统(型号 FLCAS0001,Stovall Life Science, 美国)主要由培养基补给瓶、蠕动泵(ISMATEC, 瑞士)、除泡器、流动培养室及废液瓶组成(图 1(a))。全新的系统用伽马射线灭菌, 组装需在无菌室进行, 且组装前补给瓶、连接硅胶管和连通盖等全部经高温高压灭菌, 组装时各连接口处应浸泡于 75%乙醇中灭菌。流动培养室(图1(b))是生物膜的形成位置, 尺寸如图 1(c)所示, 包含 3 个 40mm´4mm´1mm 的流动通道。
1.3.2接种与培养
Flow Cell 系统组装完成后, 先通入液体培养基并除去气泡。接种前关闭系统各阀门及蠕动泵, 用1mL 注射器插入硅胶管, 向每个流动通道注射入300μL 菌液, 拔出注射器后迅速用硅胶将针孔密封。接种后, 在无流速情况下培养一定的时间(即初始黏附时间), 使降解菌黏附于流动培养室的基质表面, 从而在此基础上继续形成生物膜。最后, 将流动培养室正置, 设定目标流速, 在室温条件下的无菌室培养。
1.4.1荧光染色
使用复合荧光染色剂 LIVE/DEAD BacLightTM Bacterial Viability Kit 对生物膜中的细胞进行染色, 该染料由两种成分构成: 一种是红色荧光核酸染料PI (propidium iodide, 碘化丙啶), 可以标记细胞膜结构损坏的死细胞; 另一种是绿色荧光核酸染料SYTO9, 可以标记细胞膜结构完整的活细胞。染色步骤具体如下: 关闭 Flow Cell 系统各阀门及蠕动泵, 用 1mL 注射器插入培养室前的硅胶管道, 向每个通道注射 300μL 染料, 拔出注射器后迅速用硅胶将针孔密封; 系统避光染色 15 分钟后, 打开各阀门, 用培养基灌流 15 分钟, 除去多余染料; 最后关闭所有阀门, 取下流动培养室, 待用。
1.4.2 CLSM 观察和图像信息处理
将长有生物膜的流动培养室置于 CLSM (Zeiss LSM 710 NLO & DuoScan System, 德国)下观察。对于 SYTO9 荧光, 设置激发波长为 488nm, 发射波长 545nm; 对于 PI 荧光, 设置激发波长 543nm, 发射波长为 654nm; 使用 10 倍物镜。对不同培养条件的生物膜样品, 每个样本随机选取 3 个以上区域, 由生物膜与培养室透明基底相贴的一面, 向外(生物膜游离的一面)逐层 Z 轴扫描(扫描间距约 1.5 μm), 得到系列 2 维图像, 并使用 IMARIS 软件(Bitplane, 瑞士)进行 3 维图像重建。

图1 用于培养生物膜的FlowCell系统装置
Fig. 1 Flow Cell system used for culturing biofilm
对于所获得的生物膜CLSM图像, 采用显微镜配套软件 ZEN Lite 2012 (Blue edition, Carl Zeiss)和Image Pro Plus 图像统计软件, 对不同培养条件的生物膜样品(每个样品选取 3 个随机区域图像作为平行样), 计算如下 3 个指标: 1)用生物膜的平均最大厚度, 间接地代表生物膜的生物量; 2)用单位面积红色和绿色荧光面积之和占基质总面积的比例, 计算细胞的基质覆盖率, 代表降解菌群落在材料表面定殖的效率; 3)用绿色与红色荧光强度之比, 计算活细胞/死细胞比例, 并分别计算最内层、中间层和最外层(即每个样品沿 Z 轴拍摄的系列 2 维图像的第 1 张、正中间的 1 张和最后 1 张)绿色与红色荧光强度之比, 确定生物膜结构中活细胞/死细胞的分布。
在 Flow Cell 系统中, 从初始细菌接种量、初始黏附时间以及培养过程的培养基流速三方面考虑,设置两种生物膜培养条件: “低负荷”条件和“高负荷”条件。低负荷条件下, 初始细菌接种的浓度低(OD600=0.1), 初始黏附时间短(1 小时), 培养基流速快(2mL/(h·channel)); 高负荷条件下, 初始细菌接种浓度高(OD600=0.5),、初始黏附时间长(3 小时), 培养基流速慢(1.2mL/(h·channel))。同时, 探究高负荷条件下不同培养时间段(60 和 84 小时)的生物膜形成特征。
对两株不同属的吡啶高效降解菌, 研究单一菌株和混合菌株生物膜形成的差异, 即在流动培养室的 3 个通道中, 分别接种 BC026 单菌、BW001 单菌及 BC026+BW001 混合菌株(等比例混合), 采用低负荷和高负荷两种培养条件。前期的研究发现, 在12 孔板培养体系中, 喹啉高效降解菌的生物膜形成对喹啉浓度具有一定的响应规律[19], 因此本研究继续在动态的 Flow Cell 系统中探究高浓度喹啉对降解菌生物膜形成的影响, 设置喹啉浓度为 200 和800mg/L 的两组实验, 接种菌株为喹啉降解菌的混合菌株 BW003+BW004 (等比例混合)。
对于 NHC 高效降解菌, 尚无使用 Flow Cell 系统进行生物膜培养的先例。我们经过尝试, 设置低负荷和高负荷两种运行参数及不同的培养时间, 经过培养得到的降解菌生物膜如图 2~4所示。
由图 2 可以看出, 在低负荷条件下, 降解菌在材料表面的定殖效率较低, 成膜量较少。虽然有堆状(mount-like)团聚结构(图 2(a))和水波纹状(ripple-like)生物膜(图 2(b))或条带状(stream-like)生物膜(图2(c))形成, 但没有形成覆盖完整的生物膜结构, 成膜位置具有一定随机性。图 3 显示, 高负荷条件下的成膜量明显更多, 降解菌能够形成具有一定厚度、致密均一、具有明显分布与结构特征的生物膜, 且几乎覆盖整个流动通道。此外, 不同随机视野之间的差异也较小, 说明结果更可靠。比较这两种运行参数条件, 说明高负荷条件更有利于 Flow Cell 系统对降解菌生物膜的培养与观察。
为确定合适的培养时间, 在高负荷条件下, 适当延长培养时间, 结果如图 4 所示。经 84 小时培养的降解菌生物膜中, 红色荧光标记的细胞大幅增加, 说明降解菌细胞大量死亡; 生物膜量也明显低于 60小时, 说明原本黏附并成膜的降解菌发生脱落。一般认为, 生物膜的发生和发展过程是周期性的, 生物膜的最后阶段就是细胞死亡、结构老化和脱落裂解[16]。因此, 84 小时可能正处于降解菌生物膜裂解死亡的发展阶段, 不适合观察降解菌成膜。
初始黏附是生物膜形成的启动阶段, 若细菌细胞无法黏附到材料表面, 则生物膜无法形成[26]。我们发现, 初始细菌接种浓度的提高与初始黏附时间的延长有利于降解菌在流动培养室基底材料的表面发生黏附, 从而进一步形成生物膜。Wang 等[27]发现, 对于 P. aeruginosa 和 Escherichia coli, 初始黏附时间从 1 小时提高到 3 小时, 细胞黏附量可分别提高 84%和 116%。培养基流速的降低, 减少对降解菌的水力剪切力, 有助于细菌更均匀地分布在材料表面, 因而形成的生物膜更加均质化, 这也符合Rochex 等[28]提出的高水力剪切力可减缓生物膜形成的结论。Stoodley 等[29]发现, 在较高水力剪切力条件下, 细菌偏好形成丝带状生物膜, 而在低剪切力条件下, 易形成较厚的堆状结构。综上所述, 在初始接种浓度为 OD600=0.5、初始黏附时间为 3 小时、培养基流速为 1.2 mL/(h·channel)和培养时间为60 小时的条件下, 更适合降解菌生物膜在 Flow Cell系统中的培养与观察。
在实际工程环境中, 生物膜往往不是由单一种类的微生物形成, 而是由多种不同的微生物聚集构成。因此, 本研究对比吡啶降解菌的单一菌株和混合菌株形成生物膜特征的差异。在低负荷和高负荷两种条件下, 单一菌株和混合菌株经 60 小时培养而形成的生物膜特征指标如图 5 所示。
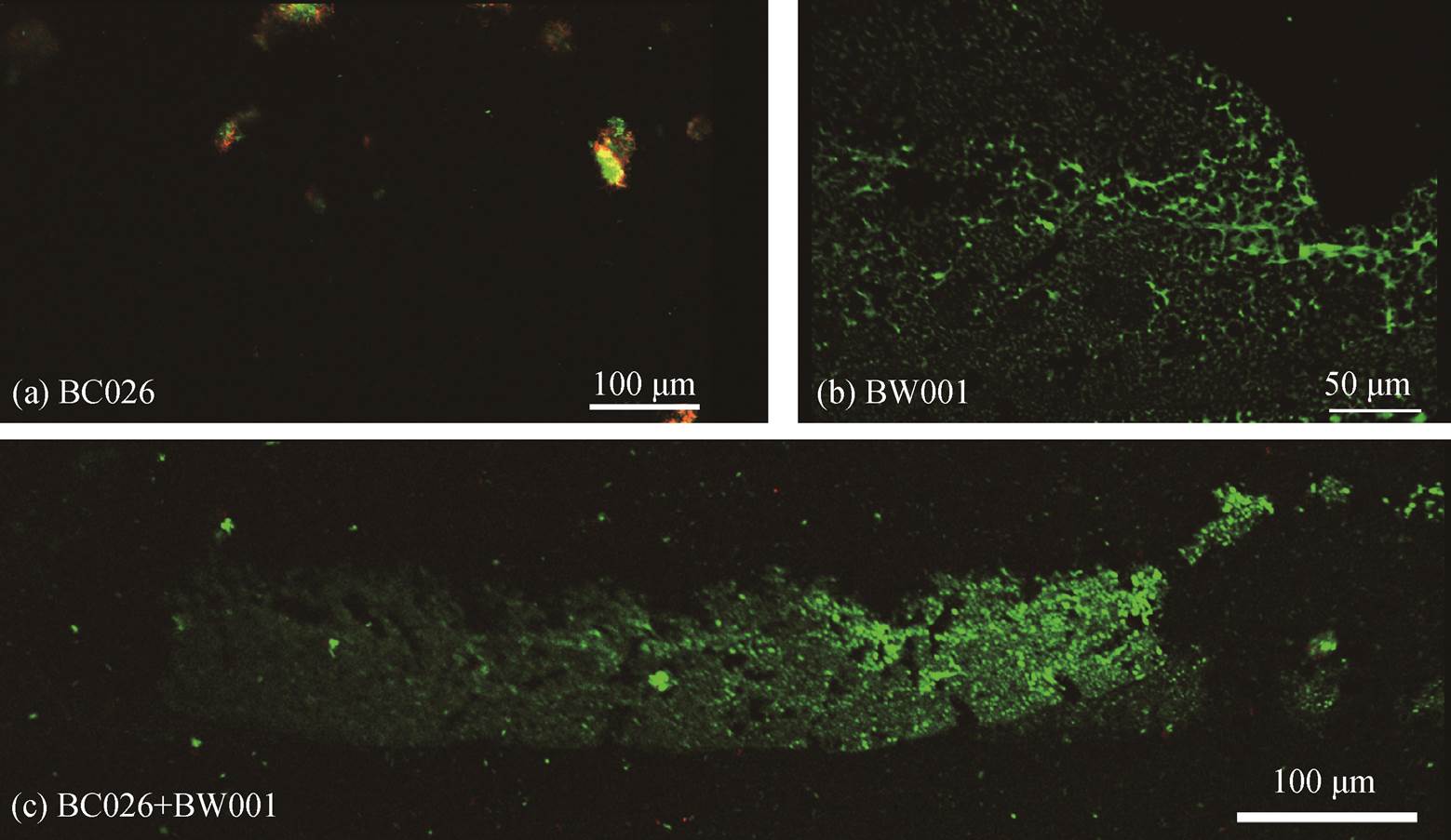
图2 低负荷条件下吡啶降解菌的生物膜2维CLSM照片(60小时)
Fig. 2 2D CLSM images of biofilm formed by the pyridine-degrading bacteria under the low load condition (60 hrs)

图3 高负荷条件下吡啶降解菌的生物膜3维CLSM照片(60小时)
Fig. 3 3D CLSM images of biofilm formed by the pyridine-degrading bacteria under the high load condition (60 hrs)

图4 高负荷条件下吡啶降解菌的生物膜2维CLSM照片(84小时)
Fig. 4 2D CLSM images of biofilm formed by the pyridine-degrading bacteria under the high load condition (84 hrs)
低负荷条件(图 5(a))下, 无论是单一菌株还是混合菌株, 平均基质覆盖率都很低(7%以下), 说明均未形成覆盖完整的生物膜。BC026 单菌的平均最大厚度最大, 达到 44mm 左右, 主要是因为形成明显的堆状团聚结构。但是, 其活细胞比例较低, 可能是已进入老化阶段, 团聚体结构内的细胞死亡率升高。混合菌株生物膜的活细胞比例为 58.22%, 介于 BC026 单菌(35.02%)与 BW001 单菌(88.17%)之间。但是, 从生物膜形态来看, 混合菌株生物膜的条带状与 BW001 单菌的水波纹状更相似, 没有出现BC026 单菌的堆状结构, 且平均最大厚度接近, 可能是因为 BW001 在该条件下有更强的成膜能力, 或是 BW001 比 BC026 对营养条件更具竞争优势, 从而导致混合菌株中的 BW001 更具有优势, 使混合菌株生物膜表现出与 BW001 单菌更相似的特征。
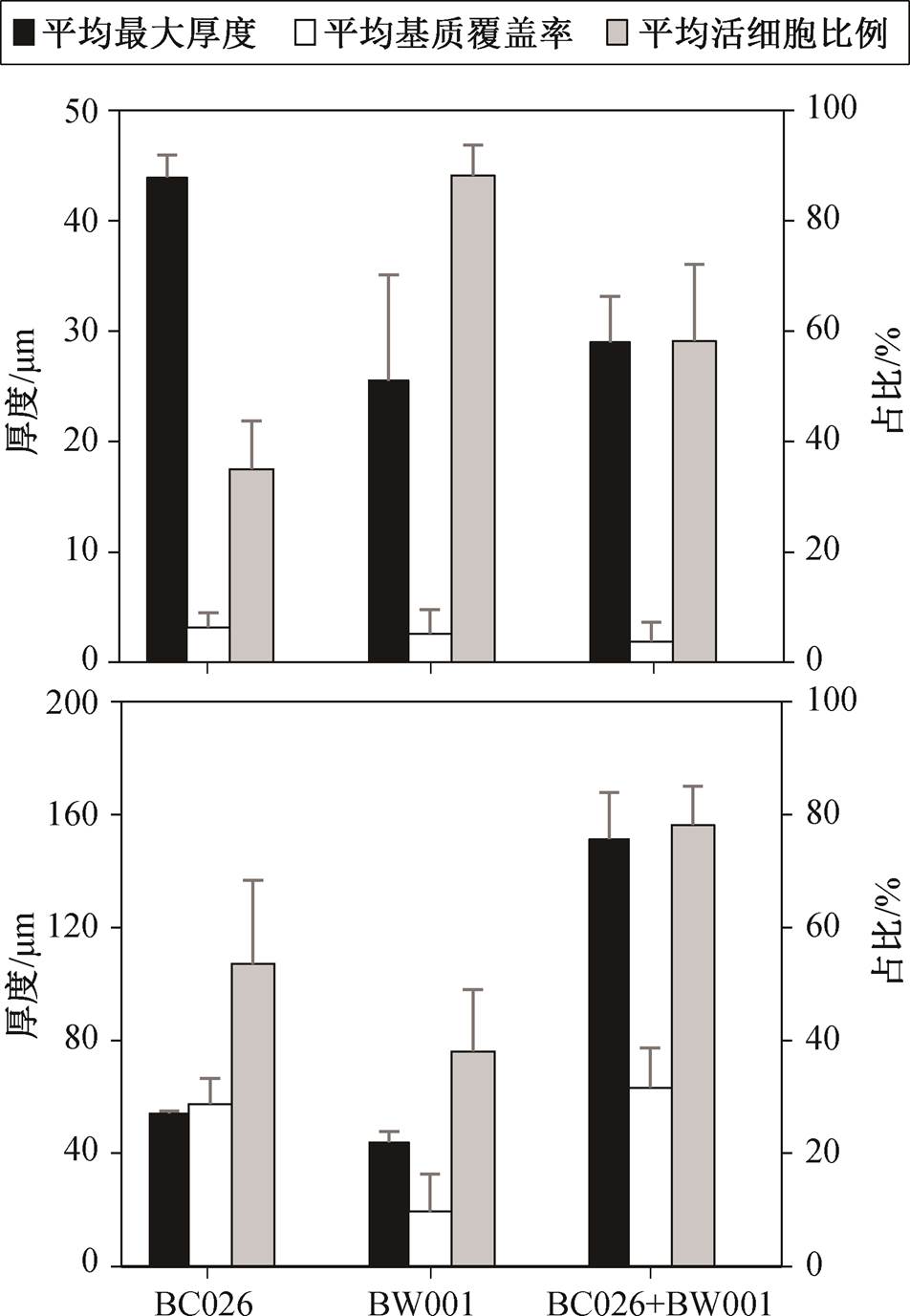
图5 低负荷(a)和高负荷(b)条件下单一菌株和混合菌株生物膜特征比较
Fig. 5 Characteristics of single-strain or dual-strain biofilm formed by the pyridine-degrading bacteria under the low (a) and high (b) load condition
高负荷条件(图 5(b))下, 单一菌株和混合菌株的平均最大厚度和平均基质覆盖率都显著升高, 即形成较为完整的生物膜。对比来看, 混合菌株在生物膜形成上具有明显优势, 表现在平均最大厚度、基质覆盖率和活细胞比例 3 个指标上, 混合菌株的生物膜平均最大厚度分别约为 BC026 和 BW001 单菌的 2.8 倍和 3.4 倍, 基质覆盖率分别约为 BC026和 BW001 单菌的 1.1 倍和 3.3 倍, 活细胞比例分别约为 BC026 和 BW001 单菌的 1.5 倍和 2.1 倍。这表明在高负荷条件下, 两种降解菌之间产生较好的合作互补关系, 从而形成比单菌更多的生物膜。从基质覆盖率、活细胞比例和生物膜形态来看, 混合菌株生物膜与 BC026 单菌生物膜的特征更相似, 说明混合菌株中的 BC026 更具有竞争优势。此外, 经84 小时培养的混合菌株生物膜的活细胞比例是单一菌株的 2~10 倍, 也间接地说明混合菌株生物膜的存活能力更强, 更能保证自身的活性。
根据前期研究, BC026 的运动能力、初始黏附能力和 EPS 分泌能力较强, BW001 的运动能力、初始黏附能力和 EPS 分泌能力显著低于其他降解菌株[19]。一般认为, 细菌鞭毛和纤毛介导的运动能力反映其与材料基质表面发生接触和黏附的能力, 运动能力越强, 就越容易发生初始黏附, 从而有助于后续团聚和成膜[30-31]; 此外, 运动能力还直接影响细菌生物膜的形态结构[32]。EPS 是细菌生物膜的框架, 其中的多糖和蛋白等物质更是直接参与生物膜结构的构建和维持[33]。因此, BC026 在低负荷条件下能够形成堆状团聚结构, 在高负荷条件下可以形成更厚的生物膜, 很可能与其较强的运动能力和EPS 分泌能力有关。还有研究发现, 在混合菌株的生物膜体系中, 如果两种细菌在某一方面具有不同的特性, 往往可以发挥协同作用。例如, 一种具有快速黏附能力的细菌可以形成一层由细胞构成的表面, 帮助另一种黏附能力较弱的细菌发生黏附[34]。本研究中, 可以推测在高负荷条件下, 黏附能力和EPS 分泌能力较强的 BC026 帮助 BW001 在已形成的生物膜结构上进行黏附和生长, 从而产生较为明显的协同效应。
通过荧光染色对生物膜内的活细胞与死细胞进行原位观察和空间定位, 既能直观地反映生物膜特征, 又可以间接地了解生物膜的形成机制, 推测成膜过程。
从图 6 可以看出, 对于低负荷条件下混合菌株形成的生物膜, 从最靠近基质表面的生物膜层(内层)到远离基质表面的生物膜层(外层), 活细胞百分比逐渐升高, 0~5mm 的生物膜层活细胞只占 40%~ 45%, 而 30~40mm 的生物膜层活细胞占 70%左右。图7展示高负荷条件下混合菌株生物膜最内层、中间层和最外层的 CLSM 照片, 可以清楚地看到各层中活细胞与死细胞的分布情况, 最内层生物膜的死细胞明显较多, 最外层的活细胞明显较多, 各层的活细胞占比分别为 67.52%, 85.25%和 87.82%。这间接地反映降解菌生物膜的形成过程, 即完整的生物膜结构是在内层细胞发生初始黏附的基础上发展而来, 符合前人研究的结论[35]。同时还发现, 在吡啶降解菌生物膜中, 存在未被荧光染色的黑色空隙或管状结构, 很可能是水通道(water channel)结构。环境中的很多营养物质经过水通道输入生物膜内部,这些通道被认为是降解菌在成膜过程中, 由于原本黏附的细胞死亡脱落, 在生物膜结构中留下空隙而形成[16]。
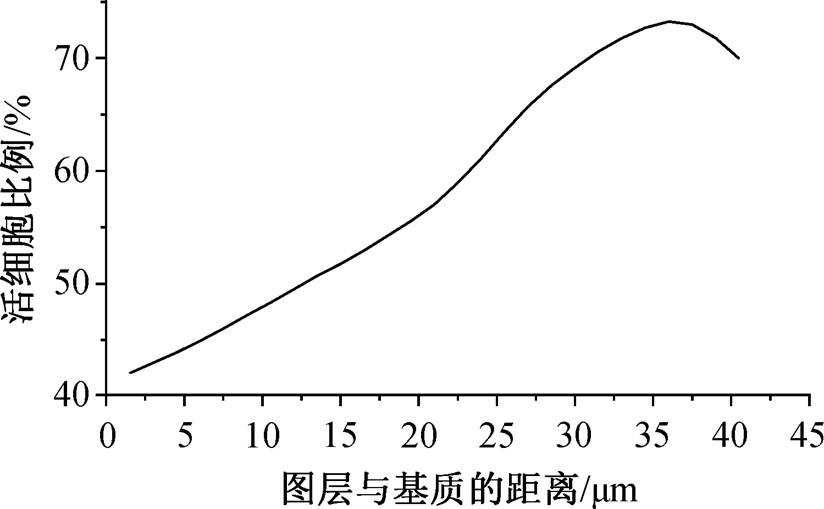
图6 低负荷条件下混合菌株生物膜中各图层的活细胞比例
Fig. 6 The ratio of living cells in different layers of the dual-strain biofilm under the low load condition
有研究者认为, 细菌生物膜的复杂结构形成和分化是伴随着细胞凋亡而实现的, 对 P. aeruginosa生物膜来说, 细胞凋亡首先发生在内部[36]。从本研究的结果来看, 这种规律也适用于吡啶降解菌混合菌株所形成的生物膜, 即从内部先开始的细胞凋亡导致内层生物膜的活细胞比例较低。同时, 这种细胞凋亡很可能导致水通道结构的产生, 从而塑造降解菌生物膜的形态结构。此外, 由于生物膜是一个由细胞、EPS 及溶液构成的非均匀体系[37], 营养物质在该体系中的纵向传输存在浓度梯度, 即外层的细胞能够接触到较多的营养, 内层的细胞所获得的营养较少[18], 因此外层生物膜的细胞活性比内层生物膜的细胞活性高。
在最适宜的 Flow Cell 运行参数下, 喹啉降解菌 BW003 和 BW004 的混合菌株在喹啉浓度分别为200 和 800mg/L 时的生物膜 3 维 CLSM 照片见图 8, 相应的生物膜特征指标见表 1。可以看出, 两种喹啉浓度下的降解菌生物膜具有明显的差异。
一方面, 团聚结构的特征差异较大。200mg/L浓度下, 喹啉降解菌的团聚结构分布较为零散, 但每个团聚结构的体积都比较大, 平均最大厚度约为800mg/L 浓度下的 1.7 倍, 说明降解菌在初始黏附的基础上逐渐发展, 形成具有一定空间结构的生物膜; 800mg/L 浓度下, 喹啉降解菌的团聚结构明显地变小, 在基质表面的分布更均匀, 但与 200mg/L浓度下生物膜的平均基质覆盖率差异不大, 可见喹啉的浓度对降解菌的定殖效率影响不大。这一结果说明, 在不同的污染物浓度下, 降解菌细胞都可以在基质表面发生不可逆的初始黏附, 但较高浓度的污染物不利于降解菌形成空间结构较大的团聚体。Cheng 等[38]在利用生物强化体系处理 3,5-二硝基苯甲酸时也发现, 难降解污染物与一般营养物质的比例对降解菌生物膜的形成具有重要影响, 比例较高时难以形成明显的生物膜, 比例降低时则会形成很大的细胞聚集体, 并最终形成较厚的生物膜。
另一方面, 生物膜内活细胞比例与分布的差异明显。200mg/L 浓度下的生物膜中活细胞比例是800mg/L 浓度下的 2.1 倍左右, 说明低浓度下降解菌生物膜更容易保持生物活性, 高浓度的污染物对生物膜细胞活性有一定的抑制作用。两种浓度条件下, 距离基质不同高度的生物膜层内活细胞比例的变化趋势也稍有不同(图 9)。200mg/L 浓度下, 最外层、中间层和最内层的活细胞比例呈逐渐下降的趋势(分别为 84.02%, 83.72%和 79.73%), 但差异不大; 800mg/L 浓度下, 最外层、中间层和最内层的活细胞比例先上升后下降(分别为 41.22%, 45.54%和29.08%), 即最先发生黏附的内层细胞大量凋亡, 中间层的细胞可能因为受到生物膜结构的保护[39], 得以保持较高的细胞活性, 而与高浓度污染物直接接触的外层细胞活性也较低。此外, 生物膜结构中包含大量胞外酶, 类似一种“外部消化系统”[40], 可以帮助细胞更好地利用一些难降解物质, 这也可能促使处于这种结构内部的细胞活性更高。
上述结果表明, 高浓度的喹啉会影响降解菌生物膜的形态和分布特征, 并抑制生物膜的发生发展以及生物膜内的细胞活性。这也启发我们, 在难降解有机污染物浓度高的废水处理系统中, 要促使降解菌更多地以生物膜形式存在, 从而保持高降解效率, 需要采取合适的人为调控策略。

图7 高负荷条件下混合菌株生物膜不同图层中活细胞与死细胞的分布
Fig. 7 Distribution of living and dead cells in different layers of the dual-strain biofilm under the high load condition
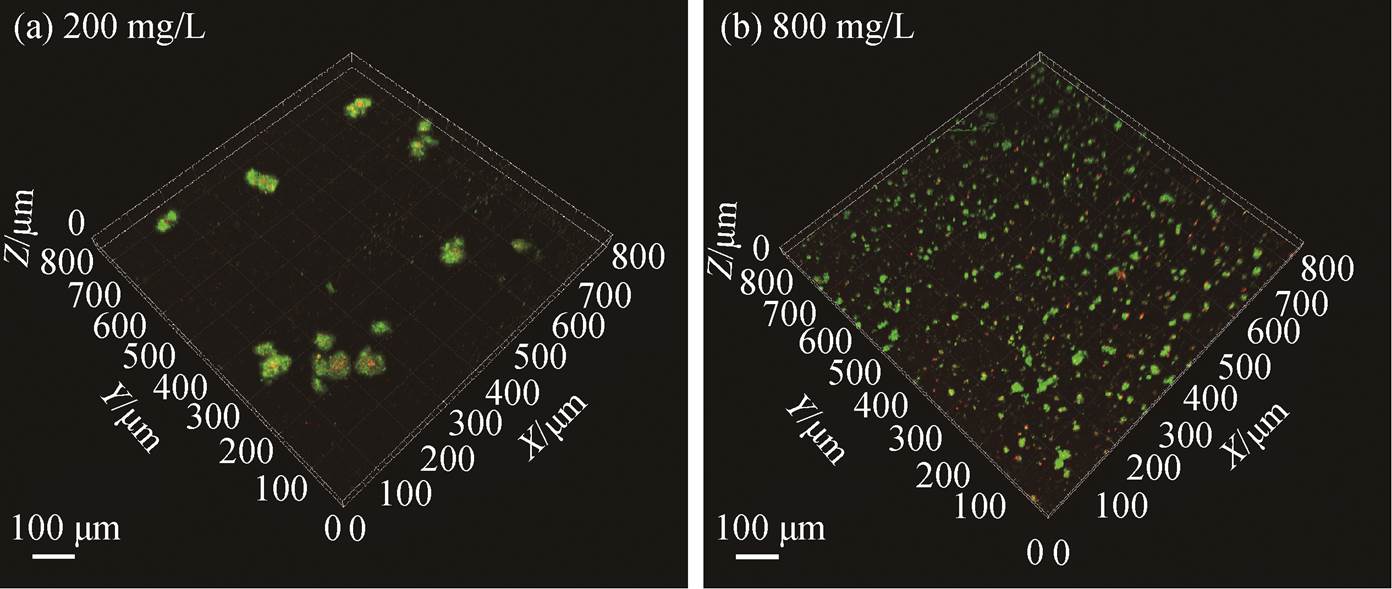
图8 不同喹啉浓度下降解菌生物膜的3维CLSM照片
Fig. 8 3D CLSM images of biofilm formed by quinoline-degrading bacteria under different quinoline concentration
表1 不同喹啉浓度下降解菌生物膜特征指标
Table 1 Characteristics of biofilm formed by quinoline-degra-ding bacteria under different quinoline concentration

实验条件平均最大厚度/μm平均基质覆盖率/%平均活细胞比例/% 200 mg/L, 60小时51.0560±3.19103.54±0.8982.49±6.78 800 mg/L, 60小时30.4107±6.69083.61±0.6138.69±8.03
本研究利用 Flow Cell 系统, 探究不同条件下 4株 NHC 高效降解菌的生物膜形成特征, 得到如下主要结论。
1)初始接种细菌量更高、初始黏附时间更长、培养基流速更低时, 降解菌更容易形成结构完整、致密厚实的生物膜。适合降解菌生物膜培养与观察的 Flow Cell 运行参数如下: 初始接种浓度 OD600= 0.5, 初始黏附时间为 3 小时, 培养基流速为 1.2mL/ (h·channel), 培养时间为 60 小时。
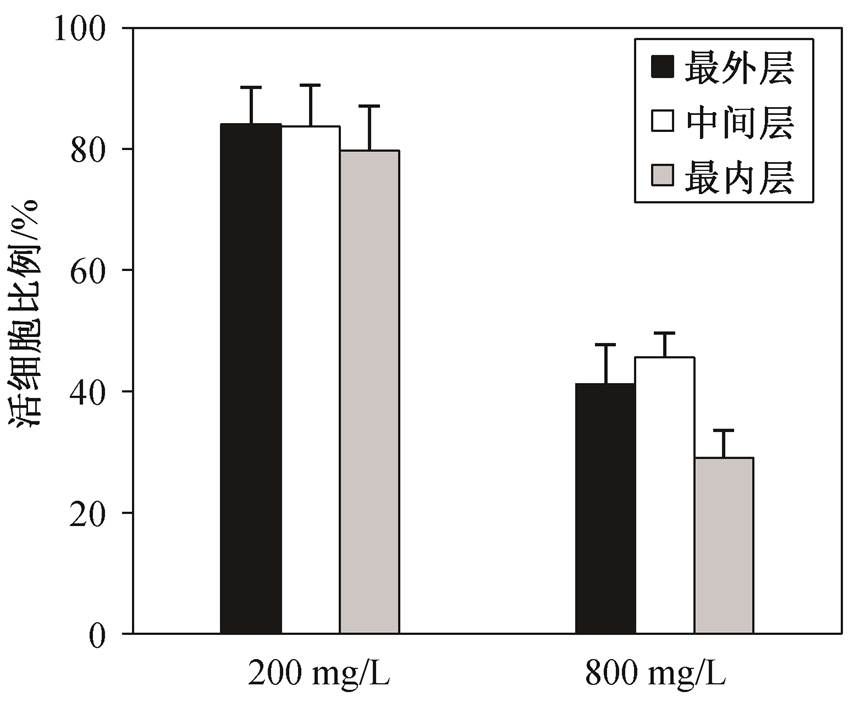
图9 喹啉降解菌生物膜不同图层活细胞比例的变化趋势
Fig. 9 The ratio of living cells in different layers of biofilm formed by quinoline-degrading bacteria
2)最适培养条件下, 与单菌生物膜相比, 吡啶降解菌 BC026 和 BW001 混合菌株形成的生物膜在生物膜厚度、基质覆盖率和活细胞比例上都更具优势, 表明这两个菌株产生合作互补关系, 形成协同效应。
3)降解菌生物膜在纵向上表现出细胞活性的差异, 即距离基质表面近的内层活细胞比例低, 而距离基质表面远的外层活细胞比例高。
4)喹啉浓度对降解菌 BW003 和 BW004 的生物膜形成有显著影响, 高浓度的喹啉会影响降解菌生物膜的形态结构, 抑制生物膜的发生发展以及生物膜内的细胞活性。200mg/L 的浓度下, 降解菌可以形成很大的细胞团聚结构, 且活细胞比例较高; 而800 mg/L浓度下形成的团聚结构明显变小, 活细胞比例也显著下降。
参考文献
[1] Xu Peng, Han Hongjun, Zhuang Haifeng, et al. Anoxic degradation of nitrogenous heterocyclic com-pounds by activated sludge and their active sites. Journal of Environmental Sciences, 2015, 31(5): 221-225
[2] Xing Xuan, Zhu Xiuping, Li Hongna, et al. Elec-trochemical oxidation of nitrogen-heterocyclic com-pounds at boron-doped diamond electrode. Chemos-phere, 2012, 86(4): 368-375
[3] Kaiser J P, Feng Y C, Bollag J M. Microbial meta-bolism of pyridine, quinoline, acridine, and their derivatives under aerobic and anaerobic conditions. Microbiological Reviews, 1996, 60(3): 483-498
[4] Bi E, Schmidt T C, Haderlein S B. Sorption of hetero-cyclic organic compounds to reference soils: column studies for process identification. Environmental Sc-ience & Technology, 2006, 40(19): 5962-5970
[5] Sims G K, Sommers L E, Konopka A. Degradation of pyridine by Micrococcus luteus isolated from soil. Applied and Environmental Microbiology, 1986, 51 (5): 963-968
[6] Shen Jinyou, Zhang Xin, Chen Dan, et al. Characte-ristics of pyridine biodegradation by a novel bacterial strain, Rhizobium sp. NJUST18. Desalination and Water Treatment, 2015, 53(7): 2005-2013
[7] Von Der Weid I, Marques J M, Cunha C D, et al. Identification and biodegradation potential of a novel strain of Dietzia cinnamea isolated from a petroleum-contaminated tropical soil. Systematic and Applied Microbiology, 2007, 30(4): 331-339
[8] Tuo Baohua, Yan Jiabao, Fan Baoan, et al. Biodegra-dation characteristics and bioaugmentation potential of a novel quinoline-degrading strain of Bacillus sp. isolated from petroleum-contaminated soil. Biore-source Technology, 2012, 107(3): 55-60
[9] Bai Yaohui, Sun Qinghua, Zhao Cui, et al. Aerobic degradation of pyridine by a new bacterial strain, Shinella zoogloeoides BC026. Journal of Industrial Microbiology & Biotechnology, 2009, 36(11): 1391-1400
[10] Bai Yaohui, Sun Qinghua, Zhao Cui, et al. Microbial degradation and metabolic pathway of pyridine by a Paracoccus sp. strain BW001. Biodegradation, 2008, 19(6): 915-926
[11] Sun Qinghua, Bai Yaohui, Zhao Cui, et al. Aerobic biodegradation characteristics and metabolic products of quinoline by a Pseudomonas strain. Bioresource Technology, 2009, 100(21): 5030-5036
[12] Zhu Shunni, Liu Dongqi, Fan Li, et al. Degradation of quinoline by Rhodococcus sp. QL2 isolated from ac-tivated sludge. Journal of Hazardous Materials, 2008, 160(2/3): 289-294
[13] Zhang Jing, Wen Donghui, Zhao Cui, et al. Bio-augmentation accelerates the shift of bacterial com-munity structure against shock load: a case study of coking wastewater treatment by zeolite-sequencing batch reactor. Applied Microbiology and Biotechno-logy, 2014, 98(2): 863-873
[14] Wen Donghui, Zhang Jing, Xiong Ruilin, et al. Bio-augmentation with a pyridine-degrading bacterium in a membrane bioreactor treating pharmaceutical waste-water. Journal of Environmental Sciences, 2013, 25 (11): 2265-2271
[15] Yue Wenlong, Chen Mei, Cheng Zhongqin, et al. Bioaugmentation of strain Methylobacterium sp. C1 towards p-nitrophenol removal with broad spectrum coaggregating bacteria in sequencing batch biofilm reactors. Journal of Hazardous Materials, 2018, 344 (2): 431-440
[16] Stoodley P, Sauer K, Davies D G, et al. Biofilms as complex differentiated communities. Annual Review of Microbiology, 2002, 56(1): 187-209
[17] Flemming H C, Wuertz S. Bacteria and archaea on Earth and their abundance in biofilms. Nature Reviews Microbiology, 2019, 17(4): 247-260
[18] Flemming H C, Wingender J, Szewzyk U, et al. Biofilms: an emergent form of bacterial life. Nature Reviews Microbiology, 2016, 14(9): 563-575
[19] 张楠, 熊富忠, 温东辉, 等. 环境因素对降解型 生物膜形成的影响. 北京大学学报(自然科学版), 2016, 52(2): 345-353
[20] Mangwani N, Shukla S K, Rao T S, et al. Calcium-mediated modulation of Pseudomonasmendocina NR802 biofilm influences the phenanthrene degrada-tion. Colloids and Surfaces B: Biointerfaces, 2014, 114(2): 301-309
[21] Zhou Gang, Li Longjie, Shi Qingshan, et al. Effects of nutritional and environmental conditions on plank-tonic growth and biofilm formation of Citrobacter werkmanii BF-6. Journal of Microbiology and Biote-chnology, 2013, 23(12): 1673-1682
[22] Lim C P, Mai P N Q, Sade D R, et al. Biofilm development of an opportunistic model bacterium analysed at high spatiotemporal resolution in the framework of a precise flow cell. NPJ Biofilms and Microbiomes, 2016, 2: 16023
[23] Williams D L, Bloebaum R D. Observing the biofilm matrix of Staphylococcus epidermidis ATCC 35984 grown using the CDC biofilm reactor. Microscopy and Microanalysis, 2010, 16(2): 143-152
[24] Crusz S A, Popat R, Rybtke M T, et al. Bursting the bubble on bacterial biofilms: a flow cell methodology. Biofouling, 2012, 28(8): 835-842
[25] Sezonov G, Joseleau-Petit D, D'ari R. Escherichia coli physiology in Luria-Bertani broth. Journal of Bacteriology, 2007, 189(23): 8746-8749
[26] Palmer J, Flint S, Brooks J. Bacterial cell attachment, the beginning of a biofilm. Journal of Industrial Mic-robiology & Biotechnology, 2007, 34(9): 577-588
[27] Wang H, Sodagari M, Chen Y J, et al. Initial bacterial attachment in slow flowing systems: effects of cell and substrate surface properties. Colloids and Sur-faces B-Biointerfaces, 2011, 87(2): 415-422
[28] Rochex A, Godon J J, Bernet N, et al. Role of shear stress on composition, diversity and dynamics of biofilm bacterial communities. Water Research, 2008, 42(20): 4915-4922
[29] Stoodley P, Cargo R, Rupp C J, et al. Biofilm material properties as related to shear-induced deformation and detachment phenomena. Journal of Industrial Micro-biology & Biotechnology, 2002, 29(6): 361-367
[30] O’toole G A, Kolter R. Flagellar and twitching motility are necessary for Pseudomonas aeruginosa biofilm development. Molecular Microbiology, 1998, 30(2): 295-304
[31] Shrout J D, Chopp D L, Just C L, et al. The impact of quorum sensing and swarming motility on Pseudo-monas aeruginosa biofilm formation is nutritionally conditional. Molecular Microbiology, 2006, 62(5): 1264-1277
[32] Guttenplan S B, Kearns D B. Regulation of flagellar motility during biofilm formation. FEMS Microbiology Reviews, 2013, 37(6): 849-871
[33] Flemming H C, Neu T R, Wozniak D J. The EPS matrix: The “House of Biofilm cells”. Journal of Bacteriology, 2007, 189(22): 7945-7947
[34] Andersson S, Rajarao G K, Land C J, et al. Biofilm formation and interactions of bacterial strains found in wastewater treatment systems. FEMS Microbiology Letters, 2008, 283(1): 83-90
[35] O’toole G, Kaplan H B, Kolter R. Biofilm formation as microbial development. Annual Review of Micro-biology, 2000, 54: 49-79
[36] Webb J S, Thompson L S, James S, et al. Cell death in Pseudomonas aeruginosa biofilm development. Journal of Bacteriology, 2003, 185(15): 4585-4592
[37] Flemming H C, Wingender J. The biofilm matrix. Nature Reviews Microbiology, 2010, 8(9): 623-633
[38] Cheng Zhongqin, Meng Xiangxun, Xie Liqun, et al. Nutrition effects on the biofilm immobilization and 3,5-DNBA degradation of Comamonas testosteroni A3 during bioaugmentation treatment. Biotechnology and Applied Biochemistry, 2015, 62(5): 719-725
[39] Jefferson K K. What drives bacteria to produce a biofilm?. FEMS Microbiology Letters, 2004, 236(2): 163-173
[40] Tielen P, Kuhn H, Rosenau F, et al. Interaction between extracellular lipase LipA and the polysac-charide alginate of Pseudomonas aeruginosa. BMC Microbiology, 2013, 13(1): 159
Investigation on Biofilm-Forming Characteristics of the Nitrogenous Heterocyclic Compound-Degrading Bacteria in a Flow Cell System
Abstract The flow cell system and confocal laser scanning microscopy were applied to investigate the characteristics of biofilm formed by the nitrogenous heterocyclic compound (NHC)-degrading bacteria under different environmental conditions. The results showed that increasing the initial inoculum density and prolonging the initial attachment time were beneficial to the adhesion and biofilm formation of the NHC-degrading bacteria on the surface of the substrate. When the flow rate of the medium was reduced, the biofilm became more homo-geneous and formed more water channel structures. Moreover, the biofilm under different conditions presented a same phenomenon that the inner layer (near the surface) had a low ratio of living cells and the outer layer (distant from the surface) had a high ratio of living cells. Compared with the single-strain biofilm, the dual-strain biofilm had an advantage in thickness, surface coverage ratio and living cell ratio. The target NHC concentration also had a significant effect on the morphology and cell viability of the quinoline-degrading bacteria biofilm: at lower concentration of quinoline, the bacteria formed large and developed aggregates; while at higher concentration, the bacterial aggregates became much smaller and evenly dispersive. Besides, the ratio of living cells of the biofilm formed at lower concentration was remarkably higher than that at higher concentration.
Key words biofilm; degrading bacteria; nitrogenous heterocyclic compounds; flow cell system
doi: 10.13209/j.0479-8023.2020.060
收稿日期: 2019-08-29;
修回日期: 2019-10-10
国家自然科学基金(51378019, 51529801)资助