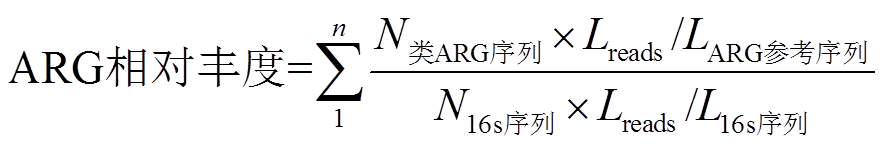 ,
,摘要 为了研究丹江口水库和汉江抗生素抗性基因(Antibiotic Resistance Genes, ARGs)的污染现状与分布规律, 采用高通量测序的方法, 于 2014 年春季和秋季分别对丹江口水库及汉江沿程的 ARGs 多样性进行检测。分别在春秋两季样本中检测出 21 和 19 类 ARGs, 其中 9 类 ARGs 是水体 ARGs 的主要组成部分。杆菌肽类 ARGs 是水体中最主要的 ARGs, 而 β-内酰胺类 ARGs 在春季丹江口水库陶岔采样点中占比最高。秋季水体各采样点的ARGs 组成结构差异比春季小, 通过 NMDS 和 ANOSIM 分析发现 ARGs 的组成存在显著的季节差异, 甲氧苄氨嘧啶类、多粘菌素类和多重耐药类 ARGs 是具有显著季节差异的 ARGs 种类。由相关性分析发现 13 类具有互相显著强相关关系的 ARGs, 其中相关性最强的 ARGs 很可能共存于一种微生物中。此外, 四环素类和氨基糖苷类 ARGs可能作为预测水源区中与其共存 ARGs 相对丰度的指示种类。研究结果可为饮用水源区的水质保护和 ARGs 污染防治与管理提供科学依据。
关键词 饮用水源; 抗生素抗性基因; 季节差异; ARGs相关关系
饮用水安全对人类的健康至关重要, 是环境保护的重要组成部分[1]。饮用水源(如河流、水库等)的水质安全是饮用水安全中的重要环节[2]。饮用水源水质检测指标包括微生物群落[3]、重金属[4]和药物残留物[5]等, 其中抗生素检测受到越来越多的关注。抗生素是一种用来治疗和防止细菌感染的抗菌药物[6], 目前主要用于治疗和预防人类或动物疾病, 也作为饲料添加剂来促进动物生长发育[7]。但是, 生物体并不能完全代谢抗生素[8], 其随污水进入污水处理厂后, 也不能被完全去除[9]。例如, 在香港两个污水处理厂发现, 抗生素的平均去除效率只有26%~68%[10]。残留抗生素会进入自然水体, 威胁饮用水源的安全[11]。
抗生素的滥用和误用造成的选择压力会导致环境中具有抗生素抗性和携带抗生素抗性基因的细菌数量大量增加[12–13]。抗生素抗性基因(antibiotic resistance genes, ARGs)是一种新兴的环境污染物[14],广泛存在于湖泊[15]、污水处理厂[16]、饮用水厂[17]和农业土壤[18]等各种环境中。江月等[19]在长江下游某水源型水库水体中检测出 118 种 ARGs, 沉积物中检测出 124 种 ARGs, 其中 β-内酰胺类、多重抗药类等 54 种 ARGs 为水库中占主导的 ARGs。Be-rgeron 等[20]在美国路易斯安纳州东南部水源中检测出四环素类和磺胺类 ARGs。抗生素抗性细菌可以通过水平基因转移或增殖传播 ARGs, 释放的 ARGs会转移至包括致病菌在内的其他细菌中。环境中抗生素抗性细菌和 ARGs 对人类健康的影响, 在一定程度上比微量残留的抗生素危害更大[21–22], 因此检测饮用水源中的 ARGs 至关重要。
南水北调工程是解决我国水资源分布不均及区域缺水问题的重大战略工程[23], 其中线工程从丹江口水库陶岔闸引水, 承担为京、津、冀、豫四省市调水的任务[24]。汉江发源于陕西宁强县, 于白河县流入湖北境内, 在十堰市丹江口与其支流丹江汇合, 最终于武汉市汇入长江[25]。丹江口水库年来水量的 90%都源于汉江[26], 而水库也会影响汉江中下游的水量和水质。汉江和丹江口水库是南水北调中线工程以及汉江中下游地区重要的水源区, 但高人口密度和人类活动对其水质有一定程度的影响[27–28], 因此有必要对其 ARGs 的污染现状展开研究, 为饮用水源区的保护提供支持。
沿汉江自陕西省安康市至湖北省武汉市布置 6 个采样点(图1), 分别为白河(BH)、丹江口坝下(DJK)、陶岔(TC)、襄阳(XY)、仙桃(XT)和集家嘴(JJZ), 其中陶岔位于丹江口水库内。
本研究于 2014 年春季(3 月)和秋季(10 月)采集水体样本。按照采样季节与样本性质, 将样本分为春季水体组(Water_Spring samples)和秋季水体组(Water_Autumn samples)。对每个采样点, 分别使用 5L 无菌PE瓶, 采集 10L 水样并立即送往临近实验室, 于 0~4°C 保存, 在 24 小时之内, 使用 0.22 μm 聚碳酸酯滤膜(Millipore, 美国)过滤全部水样, 并将滤膜置于−80°C冰箱中保存。
用无菌镊子夹取 1~2 张滤膜, 置于 2 mL 无菌离心管中, 用液氮速冻后破碎, 参照标准流程, 使用FastDNA® SPIN Kit for Soil 试剂盒(MP Biomedicals, 美国)提取 DNA。每个样本进行多次提取后混合。使用 1%琼脂糖凝胶电泳和NanoDrop 2000 (Thermo Scientific, 美国), 分别检测 DNA 的完整性及其浓度和纯度[29]。将所有 DNA 样品置于干冰中保存, 送至上海美吉生物医药科技有限公司, 用 Illumina Hiseq 4000 平台(Illumina, 美国)进行双端测序, 每个样本的下机数据不小于 20 GB。
采用 Sickle[30]和 NGSQC Tool kit[31](v2.3.3)对下机数据 raw reads 进行质量控制, 去除低质量数据, 得到 clean reads。使用 DIAMOND[32], 将 clean reads与 ARGs 数据库进行比对(https://bitbucket.org/gusphdproj/deeparg-ss/downloads/), 比对参数 E-value 为10−10。按照相似度≥90%, 匹配上的氨基酸数量≥25个的标准筛选 ARGs[33]。
由于数据库中不同 ARGs 的参考序列长度不同, 采用下式[34]对检测结果进行标准化:
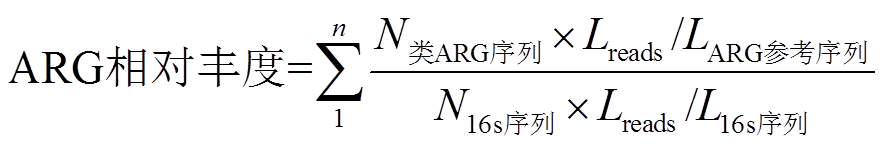 ,
,其中, N 类 ARG序列是检测为某一种特定 ARG 的 clean reads 数量; Lreads 为高通量测序中 clean reads 的长度, 本研究中春季样本均为 100 个碱基, 秋季样本均为150 个碱基; LARG参考序列为这一种特定 ARG 参考序列的长度; N16S序列是在高通量测序中识别的 16S 序列数量[29]; L16S序列是 Greengene 数据库中 16S 序列的平均长度 1432bp[34–35]。计算出的 ARGs 相对丰度单位为 ARG 拷贝数/16S rRNA 拷贝数(简称ratio)[34]。
使用 R 语言(v3.4.4) pheatmap 包绘制 ARGs 相对丰度热图, 使用 R 语言 vegan 包对 ARGs 相对丰度进行非度量多维标度(non-metric multidimensional scaling, NMDS)和相似性(analysis of similarities, ANOSIM)分析与作图。使用 STAMP[36](v2.1.3)进行组间差异分析, 使用 SPSS 18.0 进行 spearman 相关性分析。
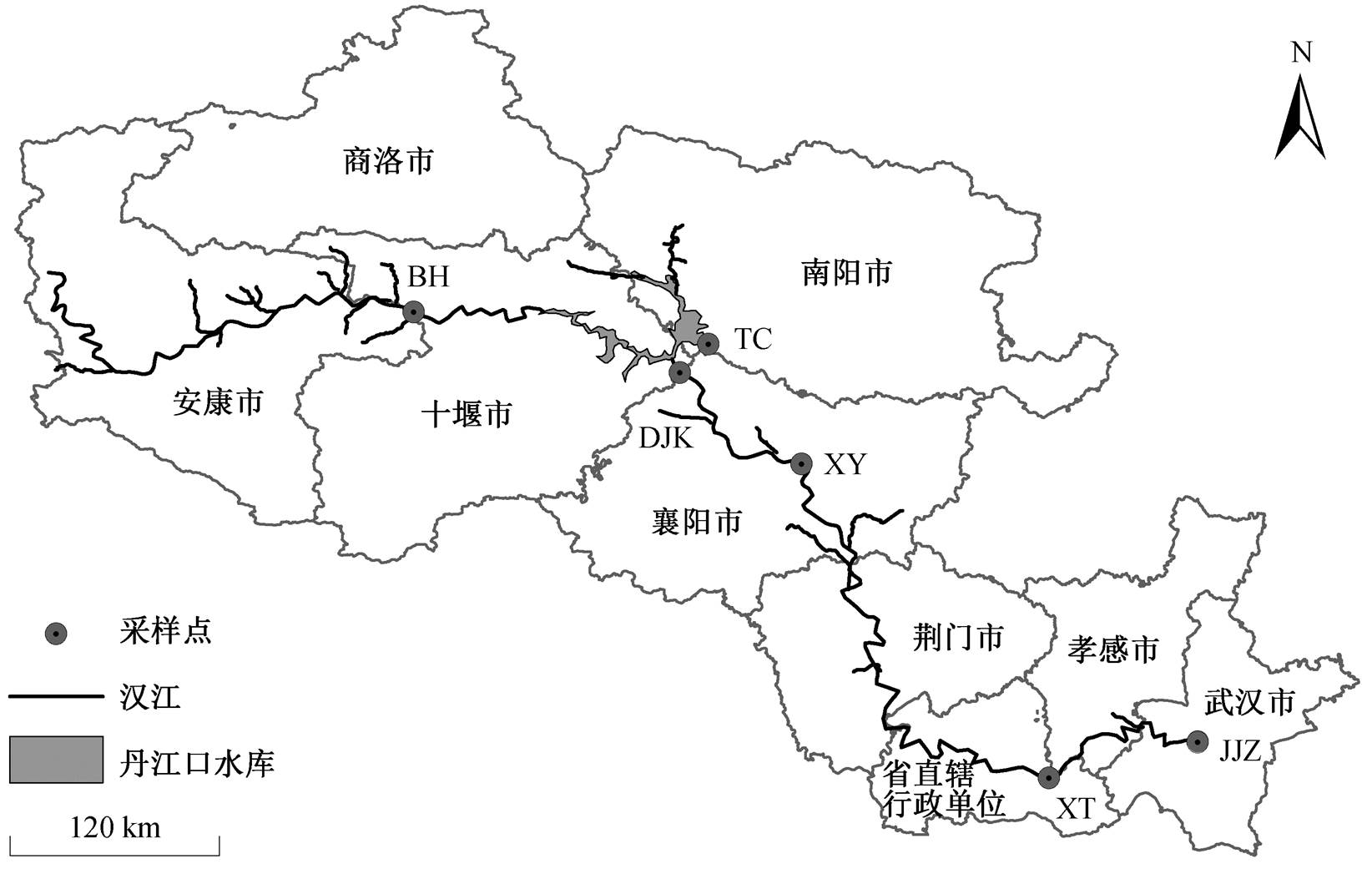
图1 采样点位置分布
Fig. 1 The distribution of sampling sites
在春季和秋季水体样本组分别检测到 21 类和 19 类 ARGs, 相对丰度如图 2 所示。春季和秋季样本组的 ARGs 总相对丰度范围分别为 8.29×10−2~ 3.38×10−1 和 6.35×10−2~1.43×10−1ratio。在春季和秋季样本组都检测出 18 类相同的 ARGs, 平均相对丰度范围分别为 2.86×10−5~7.91×10−2 和 3.58×10−6~6.51 ×10−2ratio, 占各自样本组中所有 ARGs 平均相对丰度总和的 99.983%和 99.997%。春季样本组中有 3 类独有的 ARGs, 分别是氨基香豆素类(amino-coumarin, 相对丰度为 9.34×10−6~6.69×10−5ratio)、肽类(peptide, 仅在 XY 采样点检测到, 相对丰度为1.14×10−5ratio)和截短侧耳素类(pleuromutilin, 仅在DJK 采样点检测到, 相对丰度为 1.39×10−5ratio), 特曲霉素类(tetracenomycin) ARGs 则只在秋季样本组的 DJK 采样点检测到, 相对丰度为 1.33×10−5ratio, 可以看出, 春季和秋季样本组独有的 ARGs 相对丰度很低, 而两组共有的 18 类 ARGs 是抗生素抗性组的主体。
优势 ARGs 的判断标准为其在一个样本组中的平均相对丰度在该样本组所有 ARGs 平均相对丰度总和中的占比大于等于 1%。春季水体样本组有 10类优势 ARGs, 占比从大到小排序为杆菌肽类(bacit-racin, 相对丰度为 5.44×10−2~1.01×10−1ratio, 平均相对丰度占比为 47.52%, 下同)、β-内酰胺类(beta-lactam, 2.36×10−3~2.26×10−1ratio, 28.85%)、甲氧苄氨嘧啶类(trimethoprim, 1.31×10−2~1.96×10−2ratio, 9.37%)、MLS 类(大环内酯–林可胺–链霉杀阳菌素类, macrolide-lincosamide-streptogramin, 1.19×10−3~ 1.95×10−2 ratio, 3.13%)、氨基糖苷类(aminoglycoside, 3.79×10−4~1.32×10−2ratio, 2.36%)、四环素类(tetra-cycline, 2.41×10−4~1.39×10−2 ratio, 1.74%)、磺胺类(sulfonamide, 1.04×10−3~6.42×10−3 ratio, 1.68%)、多粘菌素类(polymyxin, 1.81×10−3~3.94×10−3 ratio, 1.54%)、多重耐药类(multidrug, 3.8×10−4~4.9×10−3 ratio, 1.28%)和莫匹罗星类(mupirocin, 1.35×10−3~ 3.78×10−3 ratio, 1.19%) ARGs。
秋季水体样本组有 9 类优势 ARGs, 占比从大到小排序为: 杆菌肽类(5.21×10−2~9.25×10−2ratio, 74.16%)、β-内酰胺类(2.98×10−4~2.06×10−2ratio, 6.66%)、多重耐药性(1.16×10−3~6.98×10−3ratio, 4.05%)、氨基糖苷类(4.63×10−4~8.09×10−3ratio, 3.3%)、MLS类(1.53×10−4~7.14×10−3ratio, 3.06%)、磺胺类(9.72×10−4~4.91×10−3ratio, 2.93%)、甲氧苄氨嘧啶类(3.68×10−4~2.94×10−3ratio, 1.44%)、莫匹罗星类(5.61×10−4~1.73×10−3ratio, 1.34%)和四环素类(6.43×10−5~2.36×10−3ratio, 1.07%) ARGs。排除多粘菌素类 ARGs, 春、秋季两个水体样本组的共有优势 ARGs 有 9 类, 分别占各自 ARGs 平均相对丰度总和的 97.12%和98.01%, 说明这 9 类优势 ARGs是水体 ARGs的主要组成部分。
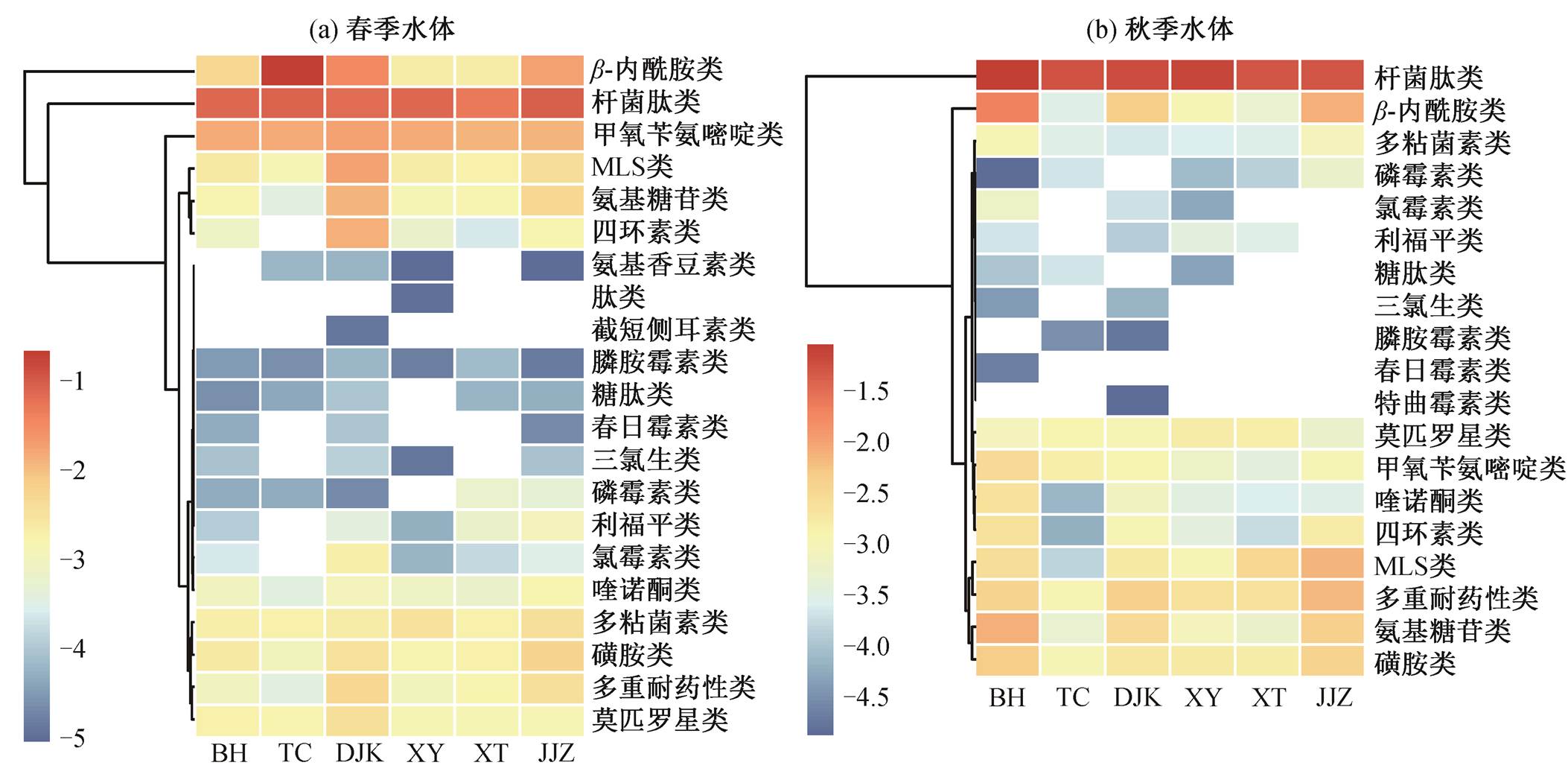
图例中的数字表示相对丰度的常用对数(以10为底)值, 图中白色方块表示该处的 ARGs 在对应采样点未检出
图2 ARGs相对丰度热图
Fig. 2 Heatmap of ARGs relative abundance
每个采样点的 ARGs 相对丰度占比如图 3 所示。春、秋季两个水体样本组占主导地位的 3 类ARGs 分别是杆菌肽类、甲氧苄氨嘧啶类和 β-内酰胺类 ARGs (春季水体)以及杆菌肽类、β-内酰胺类和多重耐药类 ARGs (秋季水体)。两个样本组之间具有季节差异, 秋季水体中杆菌肽类 ARGs 占绝对主导地位(60.18%~89.43%), 高于其在春季水体中的占比(26.35%~72.31%); 秋季水体中多重耐药类ARGs 占比(1.83%~7.83%)高于其在春季水体中的占比(0.11%~2.62%); 甲氧苄氨嘧啶类 ARGs 在秋季水体中的占比较低(0.58%~2.32%), 在春季水体中则具有一定的占比(4.81%~15.83%)。此外, 春季水体样本组 TC 和 DJK 采样点中 β-内酰胺类 ARGs 的占比分别为 66.74%和 17.99%, 明显高于其在该样本组的其他采样点(2.1%~11.53%)和秋季水体样本组中(0.47%~14.36%)的占比。
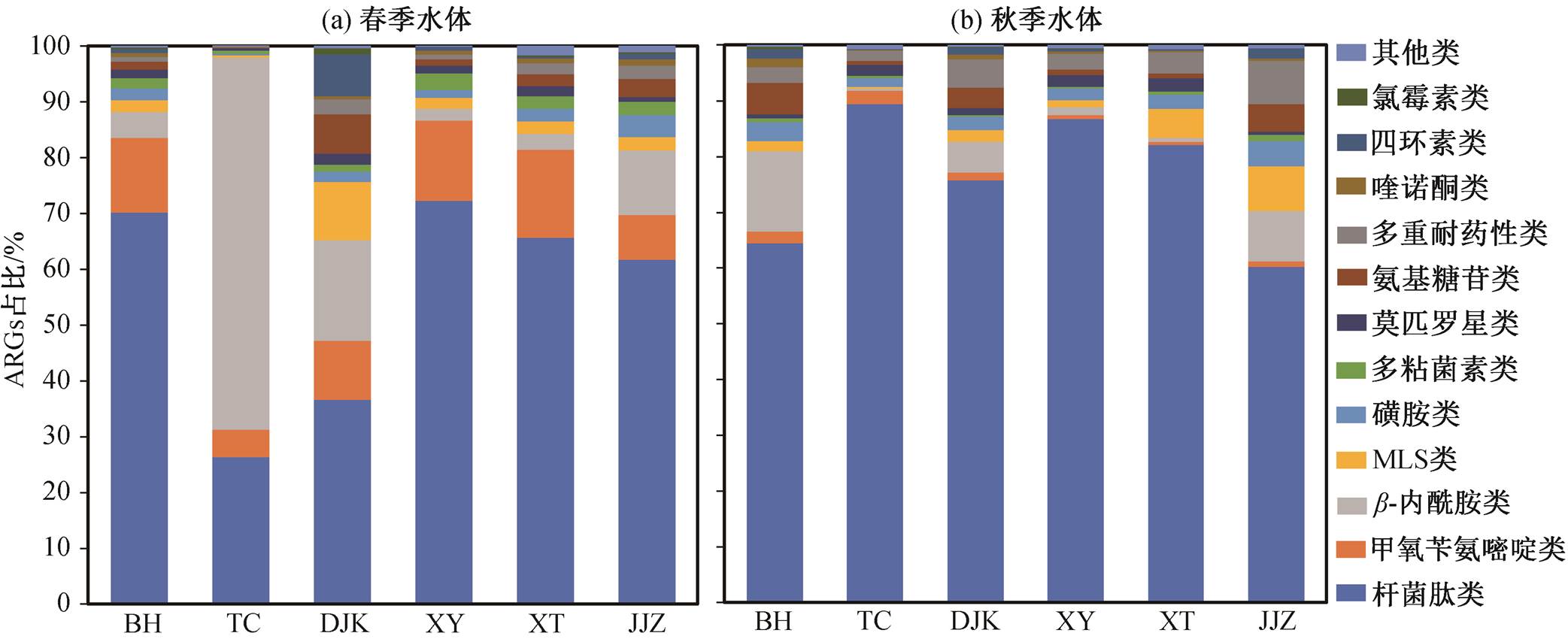
“其他类”代表在所有采样点中的占比均未达到 1%的 ARGs
图3 各采样点ARGs相对丰度的占比
Fig. 3 Proportion of ARGs relative abundance in each sampling sites
影响环境中 ARGs 相对丰度与组成的因素较多, 大量使用的抗生素对环境微生物的选择作用导致具有抗生素抗性的微生物产生和快速传播[37–38]。Forsberg 等[39]发现, 细菌群落组成是土壤 ARGs 组成的最主要决定因素, 说明环境微生物群落对ARGs 的组成结构有重要影响。温度和营养盐等各种环境因子会影响微生物生长, 从而改变微生物的群落结构[40–41], 间接地影响 ARGs 的组成。此外, 污水处理厂是重要的 ARGs 汇集与传播场所[42], 其出水排放会影响受纳水体中 ARGs 与细菌群落的组成和结构[43]。
在春季和秋季水体中, 杆菌肽类 ARGs 占比基本上是最高的。杆菌肽是一种在临床上用于对抗皮肤感染的广谱抗生素[44]。Li 等[34]发现, 杆菌肽类ARGs 是珠三角河水和人类粪便中最主要 ARGs 之一, 与杆菌肽作为人类和动物药物的大量使用有关。Christgen 等[45]通过对英格兰北部市政污水处理厂出水的检测, 发现多重耐药类、杆菌肽类和四环素类 ARGs 是出水中 ARGs 的主要部分, 分别占29.4%, 13%和 16.8%。此外, Jia等[46]通过高通量测序, 发现多重耐药类、杆菌肽类和磺胺类 ARGs 是南京地区饮用水中最主要的 3 类 ARGs。因此, 杆菌肽类 ARGs 广泛分布于各种环境中, 是抗生素抗性组的重要组成部分之一。
由图 3 还发现, 与春季相比, 秋季不同采样点ARGs 的组成更加稳定。可能的原因是, 秋季雨水多, 河流水位高于春季, 水体的混合作用比较明显, 导致沿程 ARGs 的组成变化不大; 春季雨水较少, 不同采样点附近人类活动和污水排放对 ARGs 的组成结构和相对丰度影响较大。Knapp 等[47]对古巴哈瓦那 Almendares 河流的研究也发现, 旱季水体中ARGs 的空间变化比雨季更明显, 河道附近的废水排放显著地影响不同河段的 ARGs 丰度水平。
在春季样本组中, 位于丹江口水库上游的 BH以及下游的 XY, XT 和 JJZ 采样点的 ARGs 组成结构较为相似, 而位于水库内的 TC 及丹江口大坝下的DJK 采样点中 β-内酰胺类占比明显高于其他采样点。BH, XY, XT 和 JJZ 采样点均位于人口稠密的地区, 人类对 ARGs 组成的影响较大, 而 TC 采样点所处的库区周围人类活动影响很小, 可能导致 ARGs的组成结构与其他采样点不同。Zhang 等[48]通过统计发现, 2013 年 β-内酰胺类是中国第二大使用量的抗生素。2016—2017 年对丹江口水库的抗生素检测也发现 β-内酰胺类是主要抗生素种类之一, 抗生素总浓度在春季最高, 可能的原因是其在低流量和较冷的温度条件下, 生物降解和光解速率均较慢[49]。这些因素都可能导致春季水体样本组中 TC 采样点的 β-内酰胺类 ARGs 占比远高于同组其他采样点和秋季样本组。又因为 DJK 采样点位于丹江口大坝下, 受水库放水影响, 故春季水体样本组中该采样点的 β-内酰胺类 ARGs 占比也较大。
为了验证 ARGs 的季节差异性, 对春、秋季水体样本组的 ARGs 相对丰度做 NMDS 和 ANOSIM分析。如图 4(a)所示, 春季和秋季水体的样本点聚为两簇, 说明分组效果较好。秋季水体的 6 个样本点均相对紧密地聚集在一起, 春季水体的 BH, XY, XT 和 JJZ 样本点聚集在一起, 而 TC 和 DJK 样本点与同组其他 4 个样本点的距离较远, 可能的原因是这两个样本点的 ARGs 组成结构与其他样本点差异较大。ANOSIM 分析进一步验证了 NMDS 分析的结果。如图 4(b)所示, 春季与秋季水体样本组之间具有较为显著的差异性(R=0.267, P=0.042), 组间相异指数(dissimilarity ranks)为 3~66, 相异指数中位数为 38; 春季水体组内相异指数为 1~62, 相异指数中位数为40; 秋季水体组内相异指数为 2~50, 相异指数中位数为 16。可以发现, 秋季水体组内的差异较小, 而因库区内 TC 采样点和丹江口大坝下DJK 采样点的 ARGs 组成与组内其他采样点不同, 春季水体组内具有一定的差异, 不过从整体上看, 春季与秋季水体样本组还是具有显著差异。
使用 STAMP 软件进一步分析两个水体样本组ARGs 种类的组间差异, 结果如图 5 所示, 发现甲氧苄氨嘧啶类、多粘菌素类和多重耐药类 ARGs 在组间具有显著差异(P<0.05)。甲氧苄氨嘧啶类和多粘菌素类 ARGs 在春季水体组的平均相对丰度占比分别为9.37%和 1.54%, 大于其在秋季水体组的 1.44%和 0.57%; 多重耐药类 ARGs 在秋季水体组中的平均相对丰度占比则大于春季水体组, 分别为 4.05%和1.28%。这一结果与图 3 相符。图 3 中, 甲氧苄氨嘧啶类和多粘菌素类 ARGs 在春季水体组中每个采样点的相对丰度占比都高于秋季水体组中的对应采样点, 而多重耐药类 ARGs 在春季水体组中所有采样点的相对丰度占比低于秋季水体组中对应的采样点。因此, 两个样本组间的 ARGs 组成存在显著的季节差异。
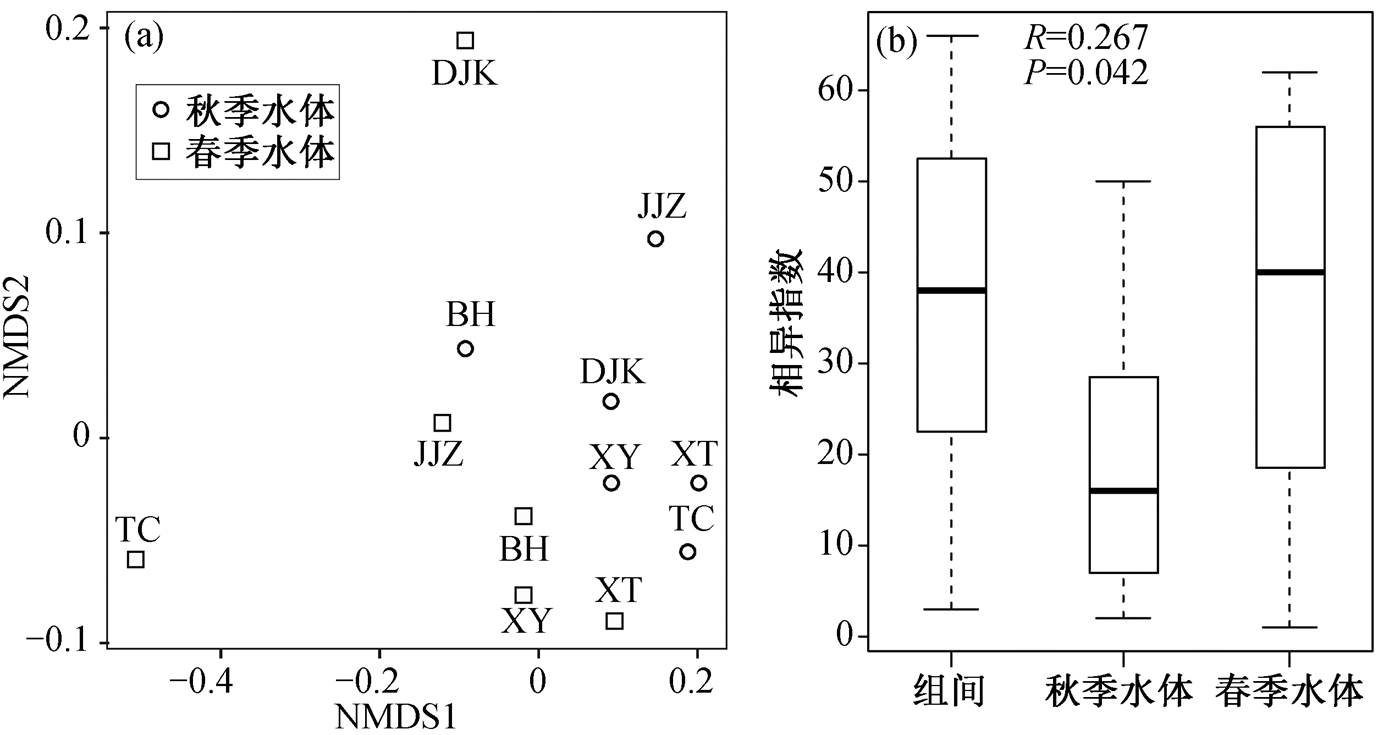
图4 ARGs的NMDS和ANOSIM分析
Fig. 4 NMDS and ANOSIM analysis of ARGs
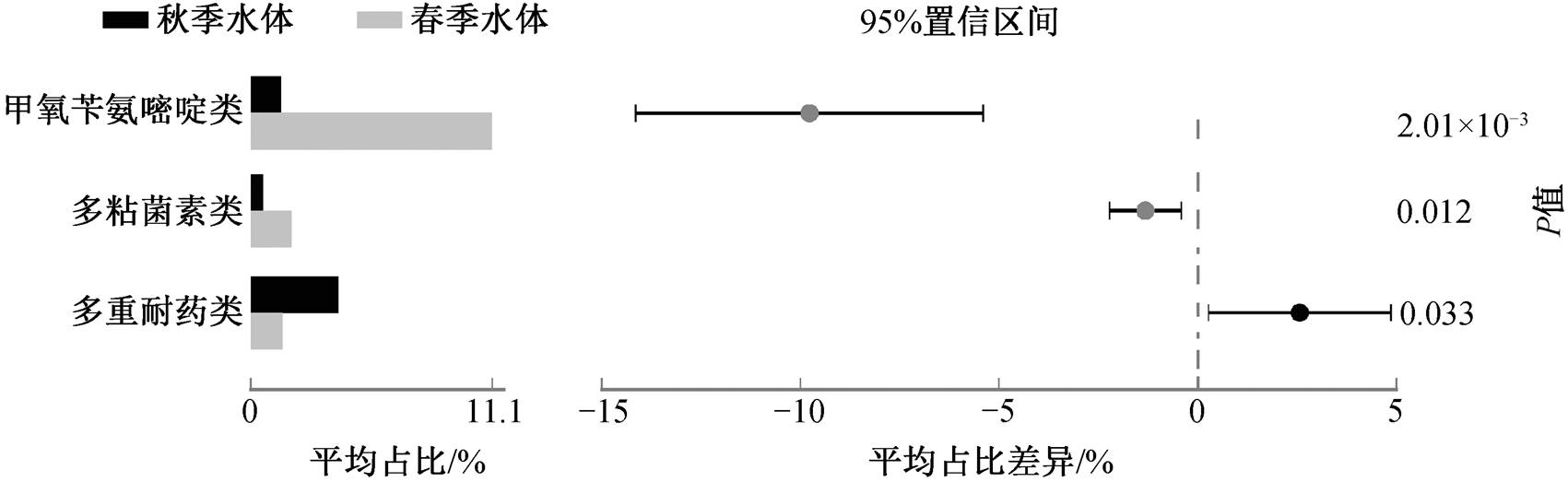
图5 显著差异的ARGs种类
Fig. 5 Significant different types of ARGs
不同种类的 ARGs 会被相同的微生物携带, 从而具有共存关系[50]。有研究表明多种 ARGs 广泛共存于大量病原体中, 并可以在土壤细菌和病原体间进行交换[51]。这些共存的 ARGs 被环境微生物, 尤其是致病菌携带后, 对人体健康的威胁远高于单种ARGs 的威胁, 因此有必要对水源区共存的 ARGs种类进行研究。
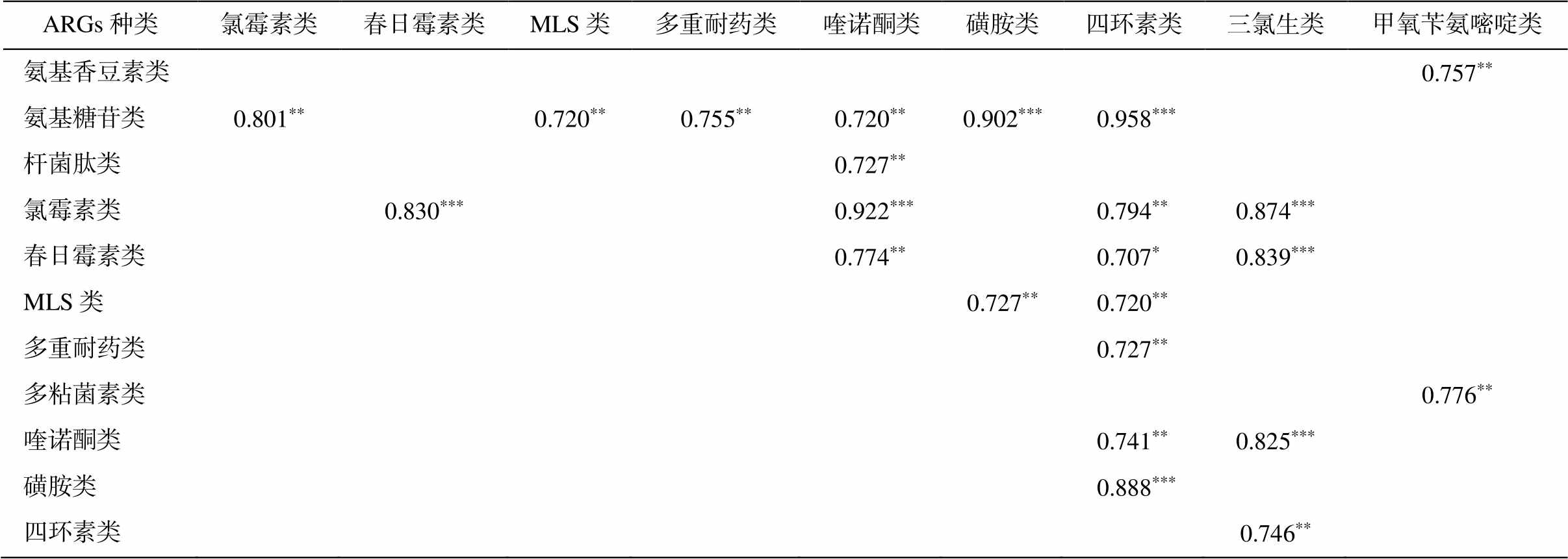
使用 SPSS 18.0 软件对所有 ARGs 相对丰度进行 Spearman 相关性分析, 去除重复值、无显著性以及有显著性但相关系数小于 0.7的记录, 结果如表 1所示。可以发现, 相关系数大于 0.7 的 ARGs 组合共有 23 对, 其中 P 值在 0.01~0.05之间的有 1 对, 在0.001~0.01 之间的有 14 对, 小于等于 0.001 的有 8对。这 23 对 ARGs 组合均具有显著的相关关系, 涵盖 13 种 ARGs。相关系数大于等于 0.9 的有 3 对, 分别是喹诺酮类与氯霉素类 ARGs、磺胺类与氨基糖苷类 ARGs、四环素类与氨基糖苷类 ARGs, 相关系数分别为 0.922, 0.902和 0.958, P 值均小于等于0.001。这种显著的强相关关系说明 3 对组合中的ARGs 极有可能分别共存于一种微生物中。
在表 1 中还可以发现, 与最多种 ARGs 具有显著较强相关关系的是四环素类和氨基糖苷类 ARGs, 分别形成 7 种和 6 种组合。Li 等[34]对水体、土壤、沉积物和生物膜样本中的 ARGs 进行 network 共存网络分析, 发现属于四环素类 ARGs 的 tetM 以及氨基糖苷类抗性蛋白在共存网络图中与最多的 ARGs相连, 是图中的两个关键节点, 可能的原因是它们都被环境中广泛存在的特定微生物所携带; 此外, 这两种 ARGs 及与其共存的 ARGs 相对丰度之间具有变化的一致性, 用幂函数对两种相对丰度的拟合性较好(R2=0.86~0.92), 因此可用来预测共存的ARGs 相对丰度。本研究结果与 Li 等[34]的结果相似, 因此四环素类和氨基糖苷类这两种 ARGs 可以作为预测环境中与其共存 ARGs 相对丰度的指示种类, 在反映饮用水源区的 ARGs 污染程度时可以发挥一定的作用。
表1 ARGs相对丰度Spearman相关性分析
Table 1 Spearman correlation analysis of the relative abundance of different ARGs
ARGs种类氯霉素类春日霉素类MLS类多重耐药类喹诺酮类磺胺类四环素类三氯生类甲氧苄氨嘧啶类 氨基香豆素类0.757** 氨基糖苷类0.801**0.720**0.755**0.720**0.902***0.958*** 杆菌肽类0.727** 氯霉素类0.830***0.922***0.794**0.874*** 春日霉素类0.774**0.707*0.839*** MLS类0.727**0.720** 多重耐药类0.727** 多粘菌素类0.776** 喹诺酮类0.741**0.825*** 磺胺类0.888*** 四环素类0.746**
说明: 重复值、无显著性关系值以及显著相关系数小于0.7的值未列出; * P=0.01~0.05, ** P=0.001~0.01, *** P≤0.001。
本文通过对南水北调中线工程水源区 ARGs 的多样性、相对丰度以及 ARGs 之间的相关性等方面进行分析, 得到如下结论。
1)在丹江口水库和汉江水源区春季和秋季水体中分别检测到 21 类和 19 类 ARGs, 其中共有的 9类优势 ARGs 相对丰度占 97%以上, 是水体 ARGs的主要组成部分。
2)杆菌肽类 ARGs 是水源区最主要的 ARGs, 而春季丹江口水库陶岔采样点的β-内酰胺类 ARGs占比最高。沿程 ARGs 组成结构的差异秋季比春季小, 可能与春季雨水量少以及沿岸人类活动影响有关。
3)水体中 ARGs 存在显著的春、秋季节差异, 甲氧苄氨嘧啶类、多粘菌素类和多重耐药类是具有显著季节差异的 ARGs 种类。
4)具有显著较强相关关系的有 13 类 ARGs, 可分为 23 对组合, 其中 3 对组合的相关性最强, 构成组合的 ARGs 很可能分别共存于一种微生物中。四环素类和氨基糖苷类 ARGs 可能作为预测水源区环境中与其共存 ARGs 相对丰度的指示种类。
本研究得到的水源区内主导 ARGs 种类和ARGs 组成的显著春秋季节差异以及可能作为指示种类的 ARGs 等结果, 有助于了解水源区 ARGs 的污染现状, 也可为饮用水源区的水质保护和 ARGs污染的针对性防治与管理提供借鉴意义。
参考文献
[1] Wang Henian, YU Xinxiao. A review of the protection of sources of drinking water in China. Natural Re-sources Forum, 2014, 38(2): 99–108
[2] 郭蓉, 孙雨婷, 曹军. 饮用水源健康风险评价现状. 中国资源综合利用, 2018, 36(9): 126–128
[3] Farenhorst A, Li R, Jahan M, et al. Bacteria in drinking water sources of a First Nation reserve in Canada. Science of the Total Environment, 2017, 575: 813–819
[4] Gupta A, Singh R, Singh P, et al. Heavy metals in drinking water sources of Dehradun, using water quality indices. Analytical Chemistry Letters, 2017, 7(4): 509–519
[5] Caban M, Lis E, Kumirska J, et al. Determination of pharmaceutical residues in drinking water in Poland using a new SPE-GC-MS(SIM) method based on Speedisk extraction disks and DIMETRIS derivatiza-tion. Science of the Total Environment, 2015, 538: 402–411
[6] Mark A M. What is antibiotic prophylaxis?. Journal of the American Dental Association, 2016, 147(6): 526
[7] 徐晨. 三峡库区水体、土壤和沉积物中抗生素与抗生素抗性基因的污染特征[D]. 武汉: 中国科学院武汉植物园, 2017
[8] Zhang Tong, Zhang Ming, Zhang Xuxuang, et al. Tetracycline resistance genes and tetracycline resisant lactose-fermenting Enterobacteriaceae in activated sludge of sewage treatment plants. Environmental Science and Technology, 2009, 43(10): 3455–3460
[9] Li Bing, Zhang Tong. Different removal behaviours of multiple trace antibiotics in municipal wastewater chlorination. Water Research, 2013, 47(9): 2970–2982
[10] Li Bing, Zhang Tong. Mass flows and removal of antibiotics in two municipal wastewater treatment plants. Chemosphere, 2011, 83(9): 1284–1289
[11] Padhye L P, Yao H, Kung'u F T, et al. Year-long evaluation on the occurrence and fate of pharmaceu-ticals, personal care products, and endocrine disrup-ting chemicals in an urban drinking water treatment plant. Water Research, 2014, 51: 266–276
[12] Fernando D M, Tun H M, Poole J, et al. Detection of antibiotic resistance genes in source and drinking water samples from a First Nations community in Canada. Applied and Environmental Microbiology, 2016, 82(15): 4767–4775
[13] Davies J, Davies D. Origins and evolution of anti-biotic resistance. Microbiology and Molecular Bio-logy Reviews, 2010, 74(3): 417–433
[14] Pruden A, Pei R, Storteboom H, et al. Antibiotic resistance genes as emerging contaminants: studies in northern Colorado. Environmental Science and Tech-nology, 2006, 40(23): 7445–7450
[15] Yang Yuyi, Liu Wenzhi, Xu Chen, et al. Antibiotic resistance genes in lakes from middle and lower reaches of the Yangtze River, China: effect of land use and sediment characteristics. Chemosphere, 2017, 178: 19–25
[16] Aydin S, Ince B, Ince O. Development of antibiotic resistance genes in microbial communities during long-term operation of anaerobic reactors in the treatment of pharmaceutical wastewater. Water Re-search, 2015, 83: 337–344
[17] Guo Xueping, Li Jing, Yang Fan, et al. Prevalence of sulfonamide and tetracycline resistance genes in drinking water treatment plants in the Yangtze River Delta, China. Science of the Total Environment, 2014, 493: 626–631
[18] Su Jianqiang, Wei Bei, Xu Chunyan, et al. Functional metagenomic characterization of antibiotic resistance genes in agricultural soils from China. Environment International, 2014, 65: 9–15
[19] 江月, 陈奕涵, 何义亮. 长江下游某水源型水库抗生素抗性基因污染研究. 环境科学学报, 2018, 38 (3): 884–891
[20] Bergeron S, Boopathy R, Nathaniel R, et al. Presence of antibiotic resistant bacteria and antibiotic resis-tance genes in raw source water and treated drinking water. International Biodeterioration and Biodegrada-tion, 2015, 102: 370–374
[21] Martínez J L, Coque T M, Baquero F. What is a resistance gene? Ranking risk in resistomes. Nature Reviews Microbiology, 2015, 13(2): 116–123
[22] 胡亚茹, 姜蕾, 张天阳, 等. 华东地区某饮用水源地中磺胺类抗性基因的分布特征. 环境科学, 2018, 39(9): 4222–4228
[23] 朱烨, 李杰, 潘红忠. 南水北调中线调水对汉江中下游水文情势的影响. 人民长江, 2019, 50(1): 79–83
[24] 田勇. 南水北调中线总干渠叶绿素 a 与藻密度相关性研究. 人民长江, 2019, 50(2): 65–69
[25] 袁洁. 汉江流域上游氮污染的时空变化格局及其来源解析[D]. 武汉: 中国科学院武汉植物园, 2017
[26] 刘海, 武靖, 殷杰, 等. 丹江口水库动库容估算及其变化. 应用生态学报, 2018, 29(8): 2658–2666
[27] Hu Ying, Yan Xue, Shen Yun, et al. Antibiotics in surface water and sediments from Hanjiang River, Central China: occurrence, behavior and risk assess-ment. Ecotoxicology and Environmental Safety, 2018, 157: 150–158
[28] Wang Beibei, Cao Menghua, Zhu Hudi, et al. Distri-bution of perfluorinated compounds in surface water from Hanjiang River in Wuhan, China. Chemosphere, 2013, 93(3): 468–473
[29] Liu Tang, Zhang Anni, Wang Jiawen, et al. Integrated biogeography of planktonic and sedimentary bacterial communities in the Yangtze River. Microbiome, 2018, 6: 16
[30] Joshi N A, Fass J N. Sickle: A sliding-window, adaptive, quality-based trimming tool for FastQ files (Version 1.33) [EB/OL]. (2011–02–06) [2019–04–06]. https://github.com/najoshi/sickle
[31] Patel R K, Jain M. NGS QC toolkit: a toolkit for quality control of next generation sequencing data. PLoS ONE, 2012, 7(2): e30619
[32] Buchfink B, Xie C, Huson D H. Fast and sensitive protein alignment using DIAMOND. Nature Methods, 2014, 12(1): 59–60
[33] Yang Ying, Li Bing, Zou Shichun, et al. Fate of antibiotic resistance genes in sewage treatment plant revealed by metagenomic approach. Water Research, 2014, 62: 97–106
[34] Li Bing, Yang Ying, Ma Liping, et al. Metagenomic and network analysis reveal wide distribution and co-occurrence of environmental antibiotic resistance genes. ISME Journal, 2015, 9(11): 2490–2502
[35] Albertsen M, Hugenholtz P, Skarshewski A, et al. Genome sequences of rare, uncultured bacteria ob-tained by differential coverage binning of multiple metagenomes. Nature Biotechnology, 2013, 31(6): 533–538
[36] Parks D H, Tyson G W, Hugenholtz P, et al. STAMP: Statistical analysis of taxonomic and functional pro-files. Bioinformatics, 2014, 30(21): 3123–3124
[37] Guo Xinyan, Yan Zheng, Zhang Yi, et al. Behavior of antibiotic resistance genes under extremely high-level antibiotic selection pressures in pharmaceutical was-tewater treatment plants. Science of the Total Envi-ronment, 2018, 612: 119–128
[38] Qiu Wenhui, Sun Jing, Fang Meijuan, et al. Occurrence of antibiotics in the main rivers of Shenzhen, China: Association with antibiotic resistance genes and mic-robial community. Science of The Total Environment, 2018, 653: 334–341
[39] Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats. Nature, 2014, 509: 612–616
[40] Zhang Yanying, Ling Juan, Yang Qingsong, et al. The diversity of coral associated bacteria and the environ-mental factors affect their community variation. Eco-toxicology, 2015, 24: 1467–1477
[41] Tago K, Okubo T, Shimomura Y, et al. Environmental factors shaping the community structure of ammonia-oxidizing bacteria and archaea in sugarcane field soil. Microbes and Environments, 2015, 30(1): 21–28
[42] Guo Jianhua, Li Jie, Chen Hui, et al. Metagenomic analysis reveals wastewater treatment plants as hot-spots of antibiotic resistance genes and mobile gene-tic elements. Water Research, 2017, 123(1): 468–478
[43] Marti E, Jofre J, Balcazar J L. Prevalence of anti-biotic resistance genes and bacterial community composition in a river influenced by a wastewater treatment plant. PLoS ONE, 2013, 8(10): e78906
[44] Radeck J, Gebhard S, Orchard P S, et al. Anatomy of the bacitracin resistance network in Bacillus subtilis. Molecular Microbiology, 2016, 100(4): 607–620
[45] Christgen B, Yang Y, Ahammad S Z, et al. Metageno-mics shows that low-energy anaerobic-aerobic treat-ment reactors reduce antibiotic resistance gene levels from domestic wastewater. Environmental Science and Technology, 2015, 49(4): 2577–2584
[46] Jia Shuyu, Shi Peng, Hu Qing, et al. Bacterial com-munity shift drives antibiotic resistance promotion during drinking water chlorination. Environmental Science and Technology, 2015, 49(20): 12271–12279
[47] Knapp C W, Lima L, Olivares-Rieumont S, et al. Seasonal variations in antibiotic resistance gene transport in the Almendares River, Havana, Cuba. Frontiers in Microbiology, 2012, 3: 1–11
[48] Zhang Qianqian, Ying Guangguo, Pan Changgui, et al. Comprehensive evaluation of antibiotics emission and fate in the river basins of China: Source analysis, multimedia modeling, and linkage to bacterial resis-tance. Environmental Science and Technology, 2015, 49(11): 6772–6782
[49] Li Si, Shi Wanzi, You Mingtao, et al. Antibiotics in water and sediments of Danjiangkou Reservoir, China: Spatiotemporal distribution and indicator scree-ning. Environmental Pollution, 2019, 246: 435–442
[50] Feng Jie, Li Bing, Jiang Xiaotao, et al. Antibiotic resistome in a large-scale healthy human gut micro-biota deciphered by metagenomic and network ana-lyses. Environmental Microbiology, 2018, 20(1): 355–368
[51] Forsberg K J, Reyes A, Wang Bin, et al. The shared antibiotic resistome of soil bacteria and human patho-gens. Science, 2012, 337: 1107–1111
Diversity of Antibiotic Resistance Genes in Water Source Areas of the Middle Route of the South-to-North Water Transfer Project
Abstract In order to study the pollution status and regularity of antibiotic resistance genes (ARGs), high-throughput sequencing methods were used to analyze the diversity of ARGs along Danjiangkou Reservoir and Hanjiang River in spring and autumn of 2014. 21 and 19 types of ARGs were detected in spring and autumn samples respectively. Among them, 9 types of ARGs were the main types of ARGs content. Bacitracin was the most important ARGs in water, while β-lactam accounted for the highest proportion in the Taocha sampling sites of Danjiangkou Reservoir. The composition difference of ARGs among sampling sites in Water_Autumn samples was smaller than that in spring. NMDS and ANOSIM analysis showed significant seasonal differences in the composition of ARGs. Trimethoprim, polymyxin and multidrug were ARGs which had significant seasonal differences. Correlation analysis found 13 types of ARGs with significantly strong correlations with each other, and the most relevant ARGs were likely to coexist in the same microorganism. In addition, tetracycline and aminoglycoside may be useful as indicators for predicting the relative abundance of ARGs coexisting in the water source region. This study could provide reference for water quality protection and ARGs pollution prevention and management in drinking water source areas.
Key words drinking water source; antibiotic resistance genes; seasonal differences; correlation of ARGs
doi: 10.13209/j.0479-8023.2020.024
国家自然科学基金(51539001)资助
收稿日期: 2019–04–15;
修回日期: 2019–06–28