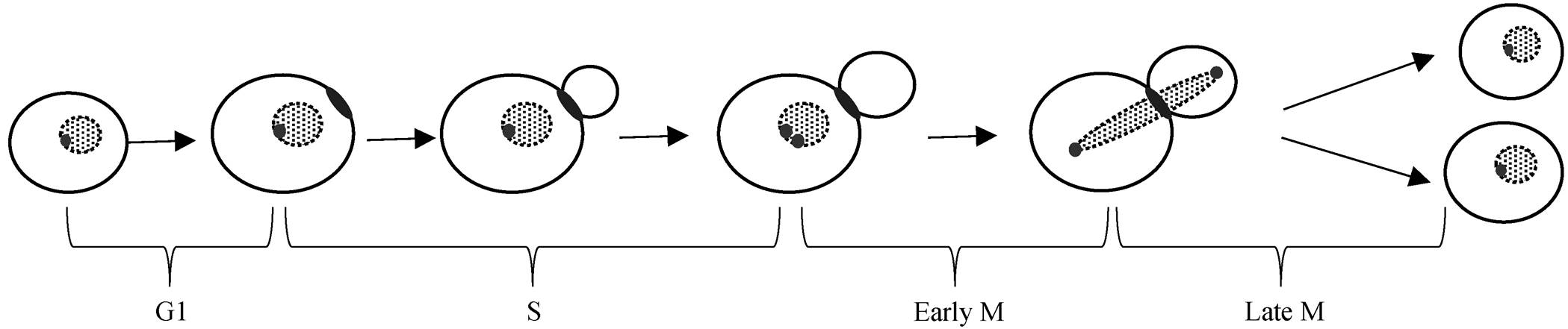
图1 芽殖酵母细胞周期示意图
Fig. 1 Schematic diagram of the budding yeast cell cycle
摘要 在芽殖酵母中构建 CDC10-mCherry 和 SPC42-mCherry 荧光蛋白, 作为酵母细胞周期不同时期的报告系统。根据酵母细胞中芽环(CDC10-mCherry)的产生和消失以及纺锤体极体(SPC42-mCherry)的复制和分离, 可以测量细胞周期中 G1 期、S 期、Early M 期和 Late M 期的时间长度。在此报告系统的基础上, 分别构建 S期细胞周期蛋白 CLB5-GFP 和 M 期细胞周期蛋白 CLB2-GFP 菌株, 实现单细胞观测, 检验报告系统工作的稳定性和准确性。该报告系统可作为研究酵母细胞周期不同时期时间长度等定量生物学问题的背景菌株。
关键词 芽殖酵母; 单细胞观测; 细胞周期; 时间标记
芽殖酵母(budding yeast)是研究真核细胞中细胞周期过程的重要模式生物。真核细胞的细胞周期过程分为 DNA 和染色体复制的 S 期(DNA synthesis phase)、遗传物质分离的 M 期(mitosis phase, 包括核分裂(mitosis)和胞质分裂(cytokinesis))以及 S 期之前的间隔期 G1 期、S 期与 M 期之间的间隔期 G2 期[1–2]。本文中简称芽殖酵母为酵母。
酵母细胞通过 G1 期、S 期和 M 期细胞周期蛋白以及其他调控蛋白组成的复杂网络, 实现对染色体复制和有丝分裂等过程的控制[3]。目前, 许多研究关注某一具体进程或相邻进程间的转换过程, 忽视酵母细胞周期过程的全局性质[4–6]。为了在单细胞水平上研究细胞周期过程中这些性质, 需要对细胞周期的不同时期加以区分。本文构建区分酵母细胞周期过程中不同时期的荧光蛋白报告系统, 为后续对酵母细胞周期全局性质的定量研究提供基础。
我们标记酵母细胞中芽环的相关蛋白 CDC10-mCherry[7]和纺锤体极体蛋白 SPC42-mCherry[8]作为区分酵母细胞周期不同时期的荧光蛋白报告系统。如图 1 所示, 用 CDC10-mCherry 标记芽环[7], 在细胞进入 S 期时, 芽环产生, 在一个细胞周期结束时, 芽环消失; 用 SPC42-mCherry 标记纺锤体极体[8], 纺锤体极体在进入 M 期时完成复制, 在通过纺锤体检验点前, 两个纺锤体极体一直紧挨在一起, 并在通过纺锤体检验点时迅速移向细胞两极(其中一个纺锤体极体进入芽孢)。通过芽环的产生、消失和纺锤体极体的动态行为, 可以将整个细胞周期划分为 G1 期、S 期、Early M 期和 Late M 期。为了验证所选时间标记的有效性, 我们还用绿色荧光蛋白(GFP)分别标记 CLB5 (S 期细胞周期蛋白)[9–10]和CLB2 (M 期细胞周期蛋白)[11–13], 在单细胞水平上观测其时序表达行为。

图1 芽殖酵母细胞周期示意图
Fig. 1 Schematic diagram of the budding yeast cell cycle
在野生型酵母菌株 W303 背景上, 首先构建两个突变株——SPC42-mCherry 和 CDC10-mCherry 菌株, 采用 URA 作为筛选标记基因。CDC10-mCherry菌株与 SPC42-mCherry 菌株的构建过程相似, 但是两者的原始菌株的交配型不同。在获得 SPC42-mCherry 和 CDC10-mCherry 两个菌株后, 将其共同培养获得合子(二倍体), 诱导其进行减数分裂产生四分体, 之后做四分体剖分[14]。在荧光显微镜下观察表型, 挑选出同时表达 SPC42-mCherry 和 CDC10- mCherry 的单倍体菌株, 作为构建后续菌株的背景菌株。在该菌株基础上, 分别构建 CLB5-GFP 菌株和 CLB2-GFP 菌株, 筛选标记为 HIS, 构建方法与SPC42-mCherry [14]相似。
利用微流控芯片技术和活体单细胞荧光显微镜, 拍摄得到酵母细胞的动态数据, 包括单个酵母细胞中不同报告蛋白表达水平随时间变化的数据。微流控芯片是微米尺度的实验平台, 我们使用的酵母芯片具有多腔室、液体扩散速度快和简单易控制等优点[15]。
使用 PerkinElmer 公司的转盘共聚焦显微镜观测单个酵母细胞中 CLB2-GFP/CLB5-GFP 的荧光水平, 拍摄时间间隔为 3 分钟[16]。实验中得到的图片是 512×512 像素的灰度图, 图像文件以矩阵形式存储在电脑中, 矩阵同时包含图片各像素的位置信息和像素亮度信息, 对于 GFP 通道的图片, 每个矩阵元的值表示该像素的绿色荧光强度。基于此矩阵, 可以对图像进行细胞识别以及细胞追踪等分析。
在明场拍摄时, 调整焦距稍稍偏离焦平面, 细胞会有较亮的边缘, 利用该特征性边缘可以实现单细胞的识别与追踪。所有处理均由 MATLAB 程序自动完成。在明场图片中识别并追踪各个细胞, 然后导入同一拍摄位置的绿色荧光(GFP)图片, 导出荧光强度等数据; 导入红色荧光(mCherry)图片, 提取芽环和纺锤体极体的动态行为, 并与 GFP 信息相关联; 根据 mCherry 数据, 识别芽环的产生和消失。类似地, 纺锤体极体的复制时刻也可以通过程序对纺锤体极体的识别和计数来确定, 但两个纺锤体极体的分离需要计算极体之间的距离。纺锤体极体的分离一般在 3~6 分钟内就可以完成, 为了减少人为误差, 我们用两个纺锤体极体的距离超过 10 个像素点作为判断纺锤体极体分离的标准。
由所述方法, 我们得到细胞内观测蛋白浓度的时序数据和细胞周期不同时期的进出时刻, 可以确定不同时期的时间长度, 并得到特定时期内的荧光强度等信息。
图 2(a)为酵母细胞细胞周期过程的实际拍摄结果。在 mCherry 荧光图中, 可以清晰地看到单个酵母细胞中芽环的产生和消失, 以及纺锤体极体的复制与分离。通过这些动态行为, 可以将细胞周期中不同时期与细胞周期蛋白表达序列相关联, 进行定量分析和研究。图 2(b)和(c)分别为 CLB2-GFP 和CLB5-GFP的时序表达情况以及细胞周期各时期的划分, 表明我们构建的报告系统可以很稳定地对细胞周期的不同时期进行划分。首先, 当芽环出现时, CLB5 表达量开始明显增加, 细胞进入 S 期; 纺锤体极体完成复制时, CLB5 表达量开始下降, CLB2 开始迅速增加, 细胞退出 S 期, 进入 Early M 期。当纺锤体极体分离时, CLB2 含量开始迅速下降, 细胞进入 Late M 期。芽环消失时, 细胞完成一个细胞周期, 进入下一个周期的 G1 期。CLB5 和 CLB2 的时序表达行为与报告系统表征的不同时期相一致。图 3(a)中多次的重复试验表明, 该报告系统可以稳定地表征酵母细胞周期的不同时期。以进入 G1 期(芽环消失)为零时刻, 将 33 组 CLB2-GFP 以及 25 组CLB5-GFP 的蛋白时序数据同步, 可以看到两者在表达时间上有明显的交叠。
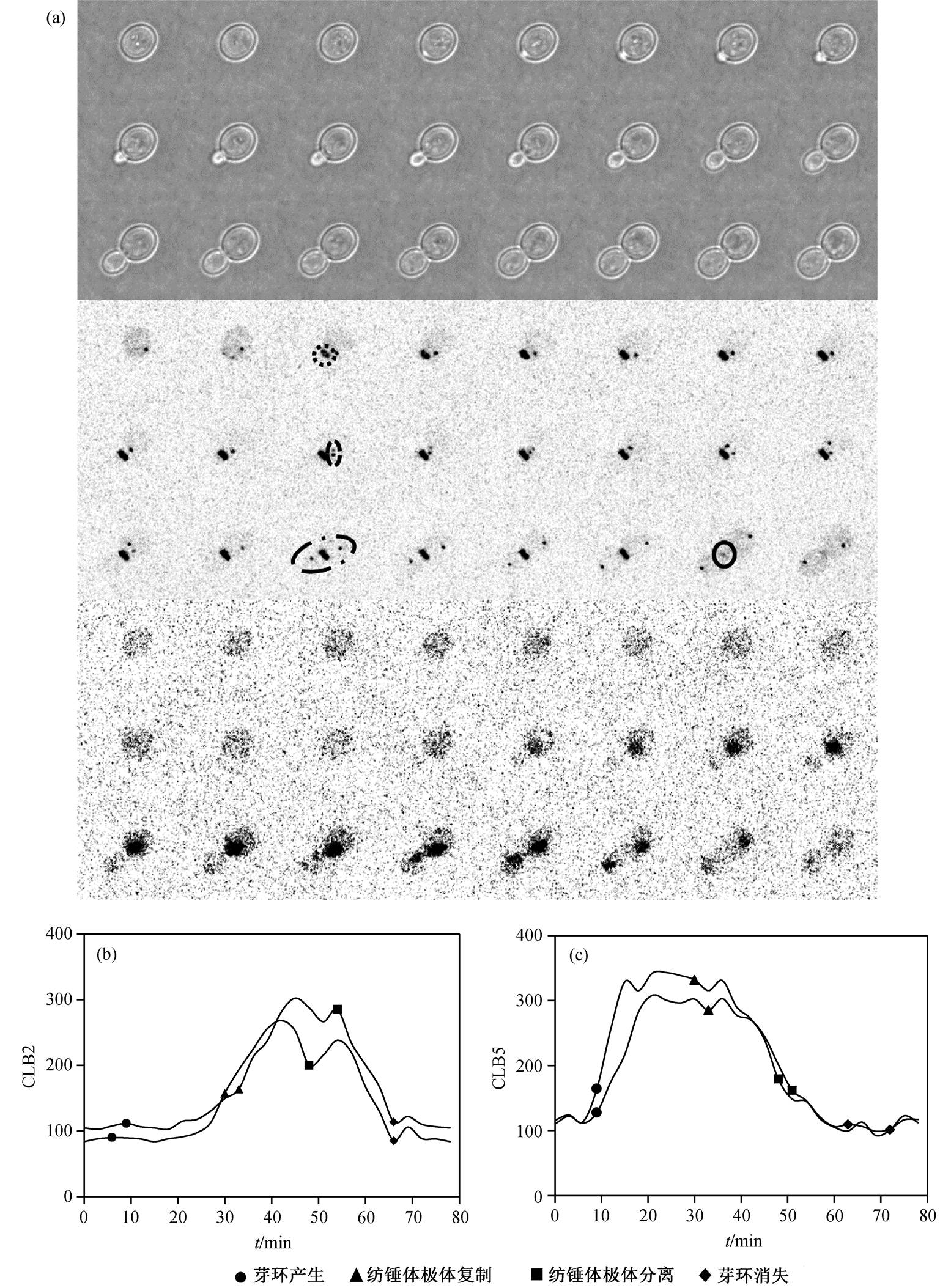
(a)中各小图按时间顺序从左到右、从上到下排列, 两张小图的时间间隔为3分钟, 按时间顺序, 在红色荧光 (mCherry)图中对芽环和纺锤体极体分别进行标记; (b)和(c)中数据同步在一个细胞周期结束(芽环消失)
图2 细胞周期各时期划分(以CLB2-GFP菌株为例) (a)以及两次独立实验中CLB2-GFP菌株(b) 和CLB5-GFP 菌株(c)的蛋白表达情况
Fig. 2 Division of the cell cycle (CLB2-GFP) (a), the expression of CLB2-GFP (b) and CLB5-GFP (c) in two independent experiments
如图 3(b)和(c)所示, 一方面, 单独一种蛋白的表达特征会随同步时刻的不同而不同, 如以纺锤体极体复制为零时刻时, CLB2-GFP 的时序数据存在单峰, 而以纺锤体极体分离为零时刻时, CLB2-GFP的时序数据明显为双峰。另一方面, 以不同的特征时刻对齐CLB5-GFP和CLB2-GFP时, 两者的表达顺序, 交叠程度均未出现明显的变化。这表明单一的时间标记不足以反映细胞周期的完整特征, 构建标记区分细胞不同时期的报告系统十分必要。细胞周期蛋白开始高表达时, 标准差迅速增大, 达到高表达状态时, 标准差基本上不再变化, 在对应时期结束后, 标准差随蛋白浓度降低迅速减小, CLB2- GFP 和 CLB5-GFP 的时序数据均表现出这一特征。这表明细胞内部可能存在某种机制, 对各事件的发生进行调控, 帮助细胞抵御外界环境的涨落, 控制细胞内部的噪声, 降低其对各事件发生的影响, 保证细胞分裂过程的精确性。
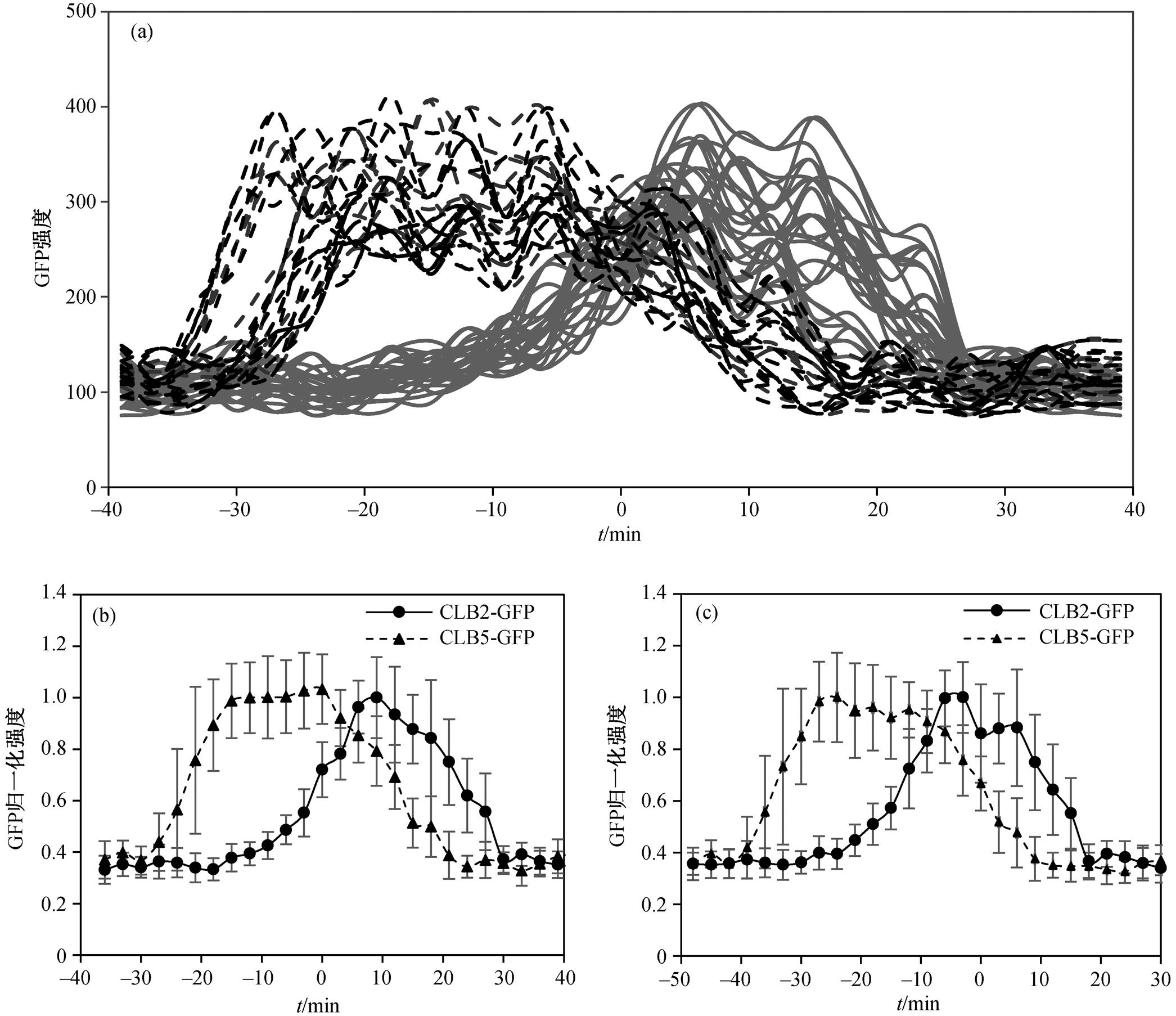
图3 以纺锤体极体复制为零时刻, 33组CLB2-GFP和25组CLB5-GFP时序数据同步(a)、CLB5-GFP与CLB2-GFP平均时序数据对齐(b)以及以纺锤体极体分离为零时刻, CLB5-GFP和CLB2-GFP的平均时序数据对齐(c)
Fig. 3 Synchronize CLB2-GFP/CLB5-GFP time series data (a) and synchronize the average time-series data of CLB5-GFP and CLB2-GFP (b) using spindle polar body separation as the timing reference, and synchronize CLB5-GFP and CLB2-GFP average time-series data using spindle elongation as the timing reference (c)
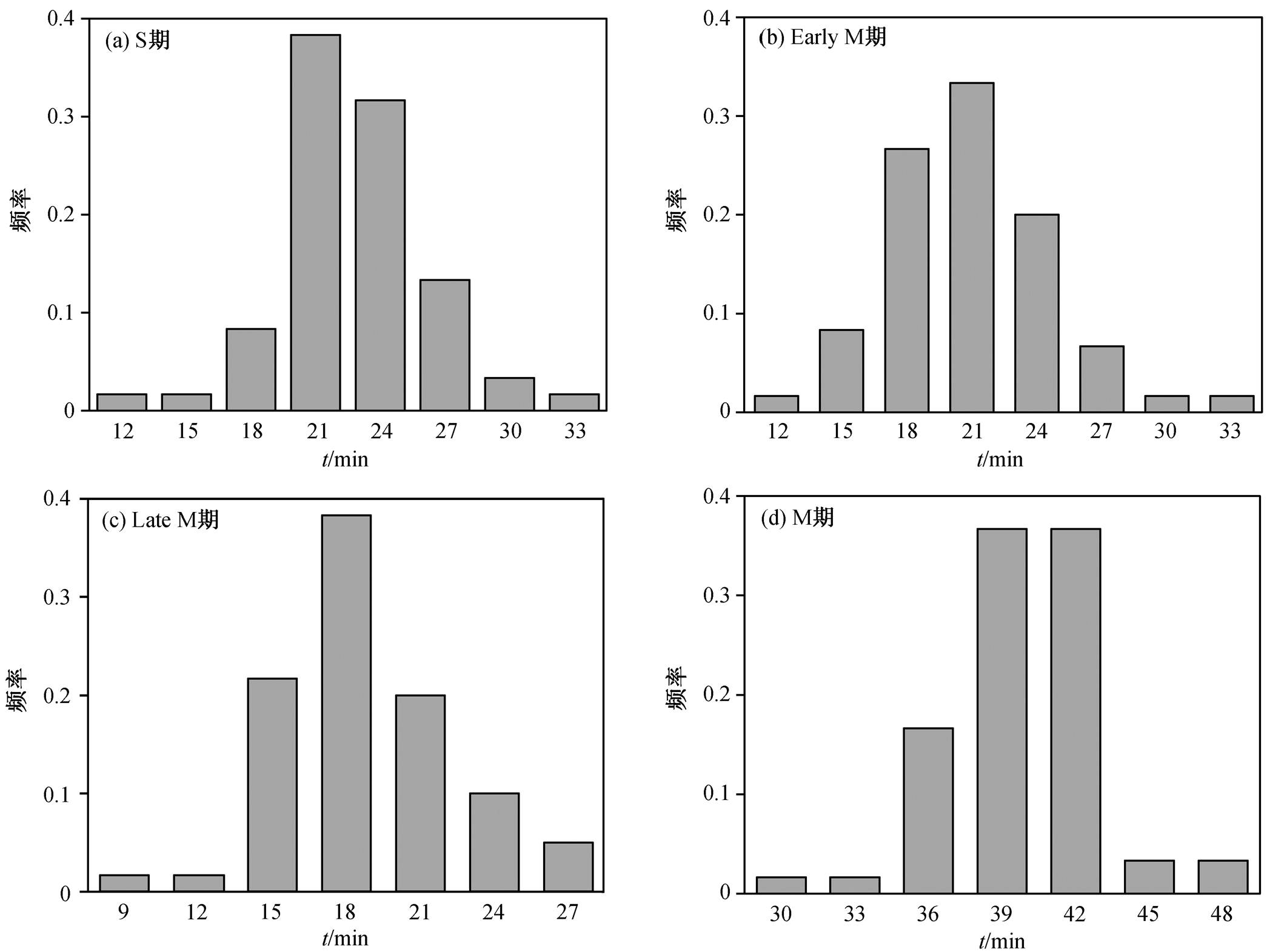
图4统计了野生型酵母细胞周期不同时期的时间长度。S 期、Late M 期和 M 期的时长似乎存在下限, 低于特定时长的细胞数量呈断崖式减少; S 期、Early M 期和 Late M 期随时长的增加细胞数量缓慢下降; M 期的时长分布特征为两侧均出现断崖式下降。各时期持续时间的下限可能源于 DNA 复制、染色体形态变化等具体事件完成的最短时间限制。各个时期随时间持续增长时, 细胞数量的变化差异表明各时期的时间长度控制可能存在不同的机制。S 期的平均时长为 22.75 分钟, 标准差为 3.5 分钟; Early M 期的平均时长为 20.9 分钟, 标准差为 3.8 分钟; Late M 期的平均时长为 18.95 分钟, 标准差为3.85 分钟; M 期的平均时长为 39.85 分钟, 标准差为 3.15 分钟。特别地, Early M 期和 Late M 期组成 M 期, 而 M 期时长的标准差要小于 Early M 期和 Late M 期的标准差。这表明 M 期前后两个阶段(EarlyM 和 Late M)可能存在相互作用, 以实现 M期整体上的稳健进行。考虑到拍摄时间间隔为 3 分钟, 各个时期的标准差均不超过 4 分钟, 表明细胞周期各个时期的时长非常稳定, 暗示某种时间调控机制的存在。在我们统计的几个时期中, M 期时长的标准差是最小的, 加上 M 期的时长呈两侧断崖式下降的分布特征, 表明统计的各个时期中 M 期是最具鲁棒性的。我们还无法解释总事件(M 期)比分事件(EarlyM 期和 LateM 期)更具鲁棒性的原因。目前的实验证据和理论分析表明, 细胞周期事件的鲁棒性可能主要得益于事件之间广泛存在的相互作用网络[17–18]。这种相互作用网络或许也会让事件之间具有很强的鲁棒性。
M 期由 Early M 和 Late M 组成
图4 细胞周期各时期时间长度统计直方图
Fig. 4 Statistical histograms of time lengths in each period of the cell cycle
本文在芽殖酵母细胞中构建了 CDC10和SPC42与红色荧光蛋白 mCherry 融合的细胞周期不同时期报告系统, 并在该体系的基础上进一步构建 CLB2和 CLB5 与绿色荧光蛋白 GFP 融合的两个报告系统, 用于检测所构建的报告系统的稳定性, 并对芽殖酵母细胞周期进行观测。实验结果表明, CDC10 和SPC42 与红色荧光蛋白融合的细胞周期时间报告系统的结果与细胞周期的不同时期的特征蛋白表达一致, 可以稳定地对细胞周期的不同时期进行区分, 并进行目标蛋白的荧光观测。在营养充足的条件下, 细胞周期不同时期的时间长度非常稳定; 特别地, EarlyM 期和 LateM 期组成 M 期, 而 M 期表现出比 Early M 期和 Late M 期更强的鲁棒性。这表明除不同时期局域的时间调控机制以外, 还存在一个全局性的时间调节机制, 维持着相关时期时长扰动的平衡。初步的应用结果表明, 该报告系统有利于更全面地反映细胞周期状况, 可以作为研究芽殖酵母细胞周期进程的背景体系。
参考文献
[1]翟中和, 王喜忠, 丁明孝. 细胞生物学. 4版. 北京: 高等教育出版社, 2011
[2]Morgan D O. The cell cycle: principles of control. London: New Science Press, 2007
[3]Murray A W, Hunt T. The cell cycle: an introduction. Quarterly Review of Biology, 1993, 2(2): 147–163
[4]Oikonomou C, Cross F R. Rising cyclin-CDK levels order cell cycle events. PloS One, 2011, 6(6): e20788
[5]Hartwell L, Weinert T. Checkpoints: controls that ensure the order of cell cycle events. Science, 1989, 246: 629–634
[6]Lu D, Hsiao J Y, Davey N E, et al. Multiple mechanisms determine the order of APC/C substrate degradation in mitosis. Journal of Cell Biology, 2014, 207(1): 23–39
[7]Gladfelter A S, Pringle J R, Lew D J. The septin cortex at the mother–bud neck. Current Opinion in Microbiology, 2002, 4(6): 681–689
[8]Donaldson A D, Kilmartin J V. Spc42p:a phos-phorylated component of the S. cerevisiae spindle pole body (SPD) with an essential function during SPB duplication. Journal of Cell Biology, 1996, 132(5): 887–901
[9]Schwob E, Nasmyth K. CLB5 and CLB6, a new pair of B cyclins involved in DNA replication in Saccharo-myces cerevisiae. Genes & Development, 1993, 7(7): 1160–1175
[10]Donaldson A D, Raghuraman M K, Friedman K L, et al. CLB5-Dependent Activation of Late Replication Origins in S. cerevisiae. Molecular Cell, 1998, 2(2): 173–182
[11]Yeong F M, Lim H H, Padmashree C G, et al. Exit from mitosis in budding yeast: biphasic inactivation of the Cdc28-Clb2mitotic kinase and the role of Cdc20. Molecular Cell, 2000, 5(3): 501–511
[12]Bäumer M, Braus G H, Irniger S. Two different modes of cyclin Clb2 proteolysis during mitosis in Saccharomyces cerevisiae. Febs Letters, 2000, 468 (2/3): 142–148
[13]Bailly E, Cabantous S, Sondaz D, et al. Differential cellular localization among mitotic cyclins from Saccharomyces cerevisiae: a new role for the axial budding protein Bud3 in targeting Clb2 to the mother-bud neck. Journal of Cell Science, 2003, 116(20): 4119–4130
[14]Amberg D C, Burke D J, Strathern J N. Methods in yeast genetics: a cold spring harbor laboratory course manual. New York: Cold Spring Harbor Laboratory Press, 2005
[15]Luo Chunxiong, Jiang Lingli, Liang Shibo, et al. High-throughput microfluidic system for monitoring diffusion-based monolayer yeast cell culture over long time periods. Biomedical Microdevices, 2009, 11(5): 981–986
[16]Gräf R, Rietdorf J, Zimmermann T. Live cell spinning disk microscopy. Advances in Biochemical Enginee-ring / Biotechnology, 2005, 95: 57–75
[17]Li Fangting, Long Tao, Lu Ying, et al. The yeast cell-cycle network is robustly designed. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(14): 4781–4786
[18]Lv Cheng, Li Xiaoguang, Li Fangting, et al. Energy landscape reveals that the budding yeast cell cycle is a robust and adaptive multi-stage process. PloS Computational Biology, 2015, 11(3): e1004156
Time Marker and Quantitative Measurement of Budding Yeast Cell Cycle
Abstract The budding yeast strains containing the CDC10-mCherry and SPC42-mCherry were constructed as a reporter system for different phases in the yeast cell cycle process. According to the generation and disappearance of bud neck (CDC10-mCherry) and the duplication and separation of spindle polar body (SPC42-mCherry), the durations of G1 phase, S phase, Early M phase and Late M phase in the yeast cell cycle can be quantitatively measured. Based on the reporter system, CLB5 (S-phase cyclin)-GFP and CLB2 (M-phase cyclin)-GFP strains were constructed and single-cell observations were performed to verify the stability and accuracy of the reporter system. The reporter system can be used as a background strain to study the time length of different phase in the yeast cell cycle.
Key words budding yeast; single-cell level observation; cell cycle; time marker
doi: 10.13209/j.0479-8023.2019.050
国家自然科学基金(11174011)和国家重点研发计划(2015CB910300)资助
收稿日期: 2018–06–13;
修回日期: 2018–12–25